Descrição
Kit de preparação conjunta da biblioteca Hieff NGS® DNA&RNA V2 é um kit de co-biblioteca de DNA&RNA para a plataforma de sequenciamento Illumina®&MGI®, que contém reagente de síntese de cDNA eficiente e reagente de digestão enzimática. Comparado com a biblioteca tradicional construção método, este produto pode completar eficientemente a síntese de cDNA e a biblioteca de DNA e RNA construção em um tubo. O kit contém enzima de alta qualidade para fragmentação de DNA e combina fragmentação de DNA, reparo final e dA-tailing em uma única etapa, o que reduz significativamente o tempo e o custo da preparação da biblioteca. Este kit de preparação de biblioteca tem uma excelente taxa de conversão de biblioteca e é aplicável para amostras de todos os animais, plantas, microrganismos comuns, etc., e também as amostras FFPE. Este kit atualizado usando a mais recente ligase otimizada diminui muito a taxa de autoligação durante a ligação do adaptador. Além disso, a introdução de uma nova polimerase de alta fidelidade melhora ainda mais a homogeneidade e a fidelidade da amplificação.
Todos os componentes fornecidos no kit passaram por rigoroso controle de qualidade e verificação funcional, o que garante a estabilidade e a repetibilidade da construção da biblioteca ao máximo.
Especificações
| Cat.No. | 12305ES08 / 12305ES24 / 12305ES96 |
| Tamanho | 8 T/24 E / 96 E |
Componentes
| Não. | Nome | 12305ES08 | 12305ES24 | 12305ES96 | |
| 12305-A | Primer aleatório | 20 μL | 60 μL | 240 μL | |
| 12305-B | Tampão de reação de cDNA | 64 μL | 192 μL | 768 μL | |
| 12305-C | Mistura de enzimas cDNA | 16 μL | 48 μL | 192 μL | |
| 12305-D | Tampão de mancha | 80 μL | 240 μL | 960 μL | |
| 12305-E | Mistura de enzimas Smearase | 40 μL | 120 μL | 480 μL | |
| 12305-F | Intensificador de Ligação | 240 μL | 720 μL | 2×1440 μL | |
| 12305-G | Romance T4 DNA Ligase | 40 μL | 120 μL | 480 μL | |
| 12305-H | 2×Super Canace® II Mix de alta fidelidade | 200 μL | 600 μL | 2×1200 μL | |
| * | Mistura de primer* | 40 μL | 120 μL | 480 μL | |
Observação: * indica que este reagente não está incluído neste kit e são necessários reagentes adicionais. O kit é compatível com plataformas duplas de Iluminar®&MGI®, mas mistura de primer adicional (CAT # 13334 Primer Mix para MGI® e Cat# 13335 Primer Mix para Illumina®) é necessário.
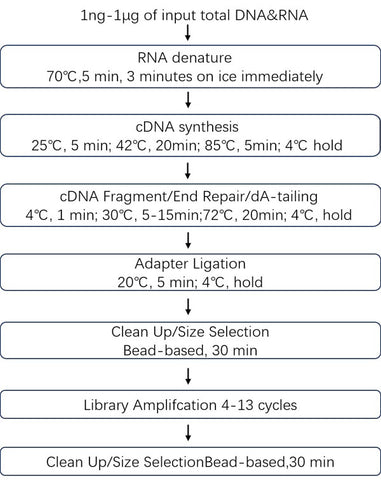
Fluxo de trabalho:
Armazenar
Este produto deve ser armazenado a -25~-15℃ por 1 ano.
Notas
Sobre a operação
- 1. Por favor, opere com jalecos e luvas descartáveis, para sua segurança.
- Descongele os componentes em temperatura ambiente.Após o descongelamento, misture bem no vórtex, gire o tubo brevemente e coloque-o no gelo para uso posterior.
- É recomendado executar cada etapa da reação em um termociclador com tampa aquecida. O termociclador deve ser pré-aquecido à temperatura definida antes do uso.
- Por favor, use consumíveis sem RNasecontaminação., e limpeza da área experimental regularmente. RNAZap do ThermoFisherTM Foi recomendado spray de remoção de ácido nucleico de alta eficiência para remover a contaminação por RNase.
- Operações inadequadas podem muito provavelmente causar contaminações por aerossóis, afetando a precisão do resultado.É recomendado o isolamento físico obrigatório das regiões de mistura de reação de PCR e regiões de ensaio de purificação de produto de PCR. Equipado com equipamentos como pipetas especializadas para construção de biblioteca.
- 6. Este produto é somente para uso em pesquisa.
Ligação do adaptador
- Os kits de adaptador longo (adaptador com código de barras) e kits de adaptador curto da Illumina ou MGI estão disponíveis para os clientes escolherem de acordo com suas necessidades experimentais.
- Foi recomendado selecionar adaptadores comerciais de alta qualidade. Se forem selecionados adaptadores feitos por você mesmo, confie a uma empresa com experiência em síntese de primers NGS e observe a necessidade de controle rigoroso de contaminação. Além disso, é recomendado preparar a solução de recozimento de DNA em uma bancada limpa e operar apenas um tipo de adaptador por vez para evitar contaminação cruzada.
- Descongele os adaptadores no gelo ou a 4 °C; ao operar em temperatura ambiente, a temperatura do laboratório não deve exceder 25 °C para evitar que os adaptadores desnaturem.
- A concentração do adaptador afeta diretamente a eficiência da ligação e o rendimento da biblioteca. O volume do adaptador adicionado ao kit é fixado em 5 μl. Recomenda-se que os adaptadores sejam diluídos com tampão 0,1×TE e os adaptadores diluídos podem ser armazenados a 4°C por 48 horas. Tabela1 lista a quantidade de adaptador recomendada para diferentes quantidades de RNA de entrada.
Tabela 1-1 Illumina recomendado® quantidade de adaptador para entrada diferente ADN e ARN
| Total de entrada ADN&ARN | Iluminar® Concentração de estoque do adaptador |
| <10 ng | 3 μM |
| ≥10 ng | 15 μM |
Tabela 1-2 O MGI recomendado® quantidade de adaptador para entrada diferente ADN e ARN
| Total de entrada ADN e ARN | MGI® Concentração de estoque do adaptador |
| <10 ng | 5 μM |
| ≥10 ng | 10 μM |
*O uso do adaptador pode ser ajustado de acordo com diferentes tipos de amostras de RNA total e quantidade de entrada.
Amplificação de biblioteca
Os números do ciclo de amplificação devem ser rigorosamente controlados. A amplificação insuficiente pode levar a um baixo rendimento da biblioteca; A superamplificação pode introduzir aumento de viés, erros, leitura duplicada e produtos quiméricos. Tabela 2 lista os números de ciclos recomendados visando o rendimento da biblioteca de 1 μg.
Mesa 2 O número recomendado de ciclos para gerar biblioteca de DNA e RNA *
| Entrada Total de DNA e RNA | Número de ciclos |
| <1 ng | 10~12 |
| 1 ng | 9~10 |
| 10 ng | 6~7 |
| 50 ng | 4~5 |
| 100~1000 ng | 4 |
Nota: *O rendimento da biblioteca não está relacionado apenas à quantidade de entrada e ao número de ciclos de amplificação, mas também é afetado pela qualidade das amostras, condições de fragmentação e condições de classificação. No processo de construção da biblioteca, escolha as condições mais apropriadas de acordo com a situação real.
Limpeza de DNA baseada em esferas e seleção de tamanho
- 1. Existem várias etapas no processo de construção da biblioteca que exigem esferas magnéticas de purificação de DNA. Recomendamos as esferas de seleção de DNA Hieff NGS™ (Yeasen Cat#12601) ou esferas magnéticas AMPure® XP (Beckman Cat#A63880) para purificação de DNA e seleção de tamanho.
- 2. As esferas magnéticas devem ser equilibradas em temperatura ambiente antes do uso, caso contrário o rendimento diminuirá e o efeito de seleção do tamanho será afetado.
- 3. As esferas magnéticas devem ser bem misturadas por vórtice ou pipetagem antes do uso.
- 4. Não aspire as esferas ao transferir o sobrenadante, mesmo pequenas quantidades das esferas podem afetar as reações seguintes.
- 5. O etanol 80% deve ser preparado recentemente, caso contrário afetará a eficiência da recuperação.
- 6. As esferas magnéticas devem ser secas à temperatura ambiente antes da eluição do produto. Secagem insuficiente facilmente fará com que o resíduo de etanol afete as reações subsequentes; secura excessiva fará com que as esferas magnéticas quebrem e reduzam o rendimento da purificação. Normalmente, a secagem à temperatura ambiente por 3-5 minutos é suficiente para permitir que as esferas sequem completamente.
- 7.Se necessário, as amostras de DNA purificadas ou selecionadas por tamanho eluídas em1× O tampão TE pode ser armazenado a 4°C por 1-2 semanas ou a -20°C por um mês.
Análise de qualidade da biblioteca
- A qualidade das bibliotecas construídas é geralmente analisada pela medição das concentrações e distribuições de tamanho.
- As concentrações das bibliotecas podem ser medidas por métodos baseados em fluorescência, como Qubit e PicoGreen ou qPCR.
- NÃO é recomendado usar métodos de quantificação baseados em absorbância, como o NanoDrop.
- É recomendado usar o método qPCR para quantificação de biblioteca: métodos baseados em fluorescência, como Qubit e PicoGreen, não conseguem diferenciar as estruturas de dsDNA incompletas (inserções sem adaptador ou com apenas uma das extremidades ligadas com adaptador) das bibliotecas completas. O método qPCR amplificará e medirá apenas as bibliotecas completas com ambas as extremidades ligadas com adaptadores (as bibliotecas sequenciáveis), fornecendo assim uma medição mais precisa para carregamento.
- A distribuição de tamanho das bibliotecas pode ser analisada usando o Agilent Bioanalyzer ou outros dispositivos baseados nos princípios de eletroforese capilar ou microfluídica.
Outros materiais
- 1. Esferas magnéticas de purificação de DNA: Hieff NGS™ DNA Selection Beads (Yeasen Cat#12601) ou AMPure®XP Beads (A63880) ou outros produtos equivalentes.
2.Adaptadores: Adaptador completo para Illumina (Yeasen Cat#13519-13520 ou outros produtos equivalentes) ou Adaptador completo para MGI (Yeasen Cat#13360-13362 ou outros produtos equivalentes).
- Análise de qualidade da biblioteca: Agilent 2100 Bioanalyzer DNA 1000 Chip/High Sensitivity Chip ou outros produtos equivalentes; reagentes quantitativos da biblioteca.
- Outros materiais: etanol absoluto, água ultrapura estéril, pontas de pipeta de baixa retenção, tubo de PCR, suportes magnéticos, termociclador, etc.
Documentos:
12305ES-Kit de preparação conjunta da biblioteca Hieff NGS® DNA&RNA V2.pdf
Pagamento e segurança
Suas informações de pagamento são processadas com segurança. Não armazenamos detalhes do cartão de crédito nem temos acesso às informações do seu cartão de crédito.
Investigação
Você também pode gostar
Perguntas frequentes
O produto é apenas para fins de pesquisa e não se destina ao uso terapêutico ou diagnóstico em humanos ou animais. Os produtos e o conteúdo são protegidos por patentes, marcas registradas e direitos autorais de propriedade da
Certos aplicativos podem exigir direitos adicionais de propriedade intelectual de terceiros.

