Descrição
Descrição do produto
O Hieff NGS™ Ultima Dual-mode RNA Library Prep Kit é um kit de construção de biblioteca de sequenciamento de RNA total para a plataforma de sequenciamento Illumina® e MGI®, incluindo reagentes de fragmentação de RNA, reagentes de transcrição reversa, reagentes de síntese de ds-cDNA convencionais e específicos de fita e reagentes de amplificação de biblioteca. A biblioteca de sequenciamento pode ser construída seguida pelo kit de purificação de mRNA ou kit de remoção de rRNA. O módulo de síntese de duas fitas é equipado com dois tampões para atender à necessidade de uma biblioteca convencional ou biblioteca específica de fita. Entre eles, o dTTP é substituído por dUTP no tampão de síntese de duas fitas específico de fita, para que o dUTP possa ser adicionado à segunda fita de cDNA. A DNA polimerase de alta fidelidade usada neste kit não pode amplificar o molde de DNA contendo uracila, alcançando especificidade de fita. Todos os reagentes fornecidos passaram por rigoroso controle de qualidade e verificação funcional, garantindo a estabilidade e reprodutibilidade da construção da biblioteca ao máximo.
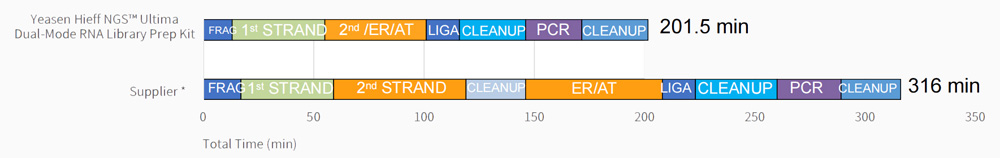
Fluxo de trabalho
Componentes do produto
| Componentes | 12308ES24 | 12308ES96 | |||
| 12308-A | 2× Tampão de Fragmentação/Primário | 250 μL | 930 μL | ||
| 12308-B | 1ª mistura de enzimas de fita | 48 μL | 192 μL | ||
| 12308-C | Reagente de especificidade de fita | 150 μL | 580 μL | ||
| 12308-D | Tampão de 2ª fita (dNTP) | 720 μL | 2×1440 μL | ||
| 12308-E | Buffer de 2ª fita (dUTP) | 720 μL | 2×1440 μL | ||
| 12308-F | 2ª fita de enzima Master Mix | 120 μL | 480 μL | ||
| 12308-G | Intensificador de Ligação | 720 μL | 2×1440 μL | ||
| 12308-H | Nova ligase de DNA T4 | 120 μL | 480 μL | ||
| 12308-I | 2×Super Canace® II Mix de alta fidelidade | 600 μL | 2×1200 μL | ||
| 12308-K | Nuclease livre H2O | 300 μL | 1000 μL | ||
Nota: Este kit é compatível com as plataformas Illumina e MGI, mas Illumina adicional ou MGI Primer Mix (Cat# 13335 Primer Mix para Illumina e Cat# 13334 Primer Mix para MGI ) é obrigatório.
Envio e armazenamento
Os componentes do kit de preparação de biblioteca de mRNA de modo duplo Hieff NGS™ Ultima na caixa I são enviados com bolsas de gelo e podem ser armazenados de 2 a 8 °C por um ano.
Os componentes do kit de preparação da biblioteca de mRNA de modo duplo Hieff NGS™ Ultima na caixa II são enviados com gelo seco e podem ser armazenados a -20 °C por um ano.
Cuidados
1 Operação
1.1 Para sua segurança e saúde, use equipamento de proteção individual (EPI), como jalecos de laboratório e luvas descartáveis, ao operar com este produto. Este produto é SOMENTE para uso em pesquisa!
1.2 Descongele os componentes em temperatura ambiente. Misture bem invertendo para cima e para baixo várias vezes, gire para baixo brevemente e coloque no gelo para uso.
1.3 É recomendado executar cada reação de etapa em um termociclador com tampa aquecida. O termociclador deve ser pré-aquecido à temperatura definida antes do uso.
1.4 São necessários suprimentos livres de contaminação por RNase e limpeza regular da área experimental.
1.5 Operações impróprias podem muito provavelmente causar contaminações por aerossol, impactando a precisão do resultado. O isolamento físico obrigatório das regiões de mistura de reação de PCR e regiões de ensaio de purificação de produto de PCR é recomendado. Equipado com equipamentos como pipetas especializadas para construção de biblioteca.
2 Ligação do adaptador
2.1 Os kits de adaptador longo (adaptador com código de barras) e kits de adaptador curto da Illumina ou MGI estão disponíveis para os clientes escolherem de acordo com suas necessidades experimentais.
2.2 Foi recomendado selecionar adaptadores comerciais de alta qualidade. Se forem selecionados adaptadores feitos por você mesmo, confie a uma empresa com experiência em síntese de primers NGS e comente sobre a necessidade de controle rigoroso de contaminação. Além disso, é recomendado preparar a solução de recozimento de DNA em uma bancada limpa e operar apenas um tipo de adaptador por vez para evitar contaminação cruzada.
2.3 Descongele os adaptadores no gelo ou a 4°C; ao operar em temperatura ambiente, a temperatura do laboratório não deve exceder 25°C para evitar que os adaptadores desnaturem.
2.4 A concentração do adaptador afeta diretamente a eficiência da ligação e o rendimento da biblioteca. O volume do adaptador adicionado ao kit é fixado em 5 μl. Recomenda-se que os adaptadores sejam diluídos com tampão 0,1×TE e os adaptadores diluídos podem ser armazenados a 4°C por 48 horas. A Tabela 1 lista a quantidade de adaptador recomendada para diferentes quantidades de RNA de entrada.
Tabela 1-1 Quantidade recomendada de adaptador Illumina para diferentes RNAs de entrada
| Entrada RNA total | Iluminar Concentração de estoque do adaptador |
| 10 ng | 1 μM |
| 100 ng | 1,5 μM |
| 500 ng | 3 μM |
| ≥1 μg | 5 μM |
Tabela 1-2 Quantidade recomendada de adaptador MGI® para diferentes RNAs de entrada
| Entrada RNA total | MGI Concentração de estoque do adaptador |
| 100-499 ng | 2 μM |
| 500-4000 ng | 5 μM |
*O uso do adaptador pode ser ajustado de acordo com diferentes tipos de amostras de RNA total e quantidade de entrada.
3 Amplificação de biblioteca
3.1 Com base na DNA polimerase de primeira geração, a DNA polimerase de alta fidelidade do kit melhorou muito sua uniformidade de amplificação e não apresenta viés de amplificação.
3.2 Se o adaptador indexado (também conhecido como adaptador longo ou adaptador Y grande) for ligado ao DNA alvo, a mistura de primers fornecida neste kit pode ser usada para amplificação; se um "adaptador curto" ou "adaptador Y pequeno" for usado para ligação do DNA, primers de índice serão necessários para amplificação.
3.3 Os números de ciclos de amplificação devem ser rigorosamente controlados. A amplificação insuficiente pode levar a um baixo rendimento da biblioteca; A superamplificação pode introduzir aumento de viés, erros, leitura duplicada, produtos quiméricos e acúmulo de mutações de expansão. A Tabela 2 lista os números de ciclos recomendados para amplificação por PCR.
Tabela 2 Número recomendado de ciclos para gerar biblioteca de RNA*
| Entrada RNA total | Número de ciclos | |
| Não encalhado | Encalhado | |
| 10 ng | 15 | 15 |
| 100 ng | 14 | 14 |
| 500 ng | 12 | 13 |
| 1 μg | 11 | 12 |
Nota:*O rendimento da biblioteca não está relacionado apenas à quantidade de entrada e ao número de ciclos de amplificação mas também afetado pela qualidade das amostras, condições de fragmentação e condições de classificação. No processo de construção da biblioteca, escolha as condições mais apropriadas de acordo com a situação real.
4 Limpeza de DNA baseada em esferas e seleção de tamanho
4.1 Há várias etapas no processo de construção da biblioteca que exigem esferas magnéticas de purificação de DNA. Recomendamos as esferas de seleção de DNA Hieff NGS™ (Yeasen Cat#12601) ou esferas magnéticas AMPure® XP (Beckman Cat#A63880) para purificação de DNA e seleção de tamanho.
4.2 As esferas magnéticas devem ser equilibradas em temperatura ambiente antes do uso, caso contrário o rendimento diminuirá e o efeito de seleção de tamanho será afetado.
4.3 As esferas magnéticas devem ser bem misturadas por vórtice ou pipetagem antes do uso.
4.4 Não aspire as esferas ao transferir o sobrenadante, mesmo pequenas quantidades das esferas podem afetar as seguintes reações.
4.5 O etanol 80% deve ser preparado recentemente, caso contrário afetará a eficiência da recuperação.
4.6 As esferas magnéticas devem ser secas à temperatura ambiente antes da eluição do produto. Secagem insuficiente facilmente fará com que o resíduo de etanol afete as reações subsequentes; secagem excessiva fará com que as esferas magnéticas quebrem e reduzam o rendimento da purificação. Normalmente, a secagem à temperatura ambiente por 3-5 minutos é suficiente para permitir que as esferas sequem completamente.
4.7 Se necessário, as amostras de DNA purificadas ou selecionadas por tamanho eluídas em tampão TE podem ser armazenadas a 4°C por 1-2 semanas ou a -20°C por um mês.
5 Análise de qualidade da biblioteca
5.1 Normalmente, a qualidade da biblioteca construída pode ser avaliada pela distribuição de comprimento e detecção de concentração.
5.2 Detecção de concentração de biblioteca: métodos baseados em corantes fluorescentes de DNA fita dupla, como Qubit®, PicoGreen®, etc.; quantificação absoluta baseada em qPCR.
5.3 Métodos baseados em detecção espectral, como NanoDrop®, etc., não são aplicáveis à detecção de concentração de biblioteca.
5.4 qPCR é recomendado para detecção de concentração de biblioteca: Por meio de Qubit®, PicoGreen® e outros métodos baseados em corantes fluorescentes de DNA fita dupla, ele não consegue distinguir efetivamente entre produtos ligados a adaptadores em uma extremidade, produtos não ligados a adaptadores em ambas as extremidades e outros produtos fita dupla incompletos. A quantificação absoluta de qPCR é baseada no princípio da amplificação por PCR, que quantifica apenas a biblioteca completa do adaptador em ambas as extremidades da amostra (a biblioteca que pode ser sequenciada), excluindo a interferência de bibliotecas não sequenciadoras que não são ligadas ao adaptador em extremidades de extremidade simples ou dupla.
5.5 A detecção da distribuição do comprimento da biblioteca pode ser realizada pelo Agilent Bioanalyzer 2100 e outros equipamentos baseados no princípio da eletroforese capilar ou microfluídica.
Documentos:
12308ES-Hieff NGS™ Ultima Kit de preparação de biblioteca de RNA de modo duplo-Ver.EN20230327.pdf
Citações e referências:
[1] Decifrando o microbioma intestinal da carpa capim por meio da abordagem multiômica
M Li, H Liang, H Yang, Q Ding, R Xia, J Chen, W Zhou… - Microbioma, 2024 IF:19.4
[2] A triagem CRISPR agrupada identifica corpos P como repressores da transição epitelial-mesenquimal do câncer
L Fang, L Zhang, M Wang, Y He, J Yang, Z Huang… - Pesquisa sobre câncer, 2024 IF:12.7
[3] O peptídeo 1 derivado de Klotho inibe a senescência celular no rim fibrótico restaurando a expressão de Klotho por meio da regulação pós-transcricional
X Zhang, L Li, H Tan, X Hong, Q Yuan, FF Hou… - Teranóstica, 2024 IF:12.4
[4] Células-tronco parcialmente revestidas de exoesqueleto para restauração do miocárdio infartado
H He, Y Yuan, Y Wu, J Lu, X Yang, K Lu… - Materiais Avançados, 2023 IF: 29,4
[5] Inovação genômica e reconfiguração regulatória durante a evolução do gênero de algodão Gossypium
M Wang, J Li, Z Qi, Y Long, L Pei, X Huang… - Nature Genetics, 2022 IF:41.379
[6] Os alelos de risco MTMR3 aumentam a imunidade IgA induzida pelo receptor Toll Like 9 na nefropatia por IgA
Y Wang, T Gan, S Qu, L Xu, Y Hu, L Liu, S Shi, J Lv… - Kidney International, 2023 IF:19.6
[7] Complementação dividida dos editores base para minimizar edições fora do alvo
X Xiong, K Liu, Z Li, FN Xia, XM Ruan, X He, JF Li - Plantas da Natureza, 2023 IF:18.6
[8] Vesículas de membrana externa bacteriana projetadas que encapsulam adenovírus oncolíticos aumentam a eficácia da viroterapia do câncer ao aumentar a autofagia das células tumorais
W Ban, M Sun, H Huang, W Huang, S Pan… - Comunicações da Natureza, 2023 IF:16.6
[9] A resistência das células cancerígenas ao IFNγ pode ocorrer por meio da atividade aprimorada da via de reparo de quebra de fita dupla
T Han, X Wang, S Shi, W Zhang, J Wang, Q Wu… - Pesquisa em Imunologia do Câncer, 2023 IF: 12,0
[10] Terapia combinada baseada em sistema de nano-entrega biomimética de alvo duplo para superar a resistência à cisplatina no carcinoma hepatocelular
Y Huang, Q Kou, Y Su, L Lu, X Li, H Jiang… - Journal of Nanobiotechnology, 2023 IF: 10.9
[11] PIAS3 promove ferroptose regulando TXNIP via via de sinalização TGF-β no carcinoma hepatocelular
W Bao, J Wang, K Fan, Y Gao, J Chen - Pesquisa Farmacológica, 2023 IF:9.3
[12] Identificação de alvos de proteína de ligação a RNA com HyperTRIBE em Saccharomyces cerevisiae
W Piao, C Li, P Sun, M Yang, Y Ding, W Song… - International Journal of molecular Science, 2023 IF:5.6
[13] A microbiota regula a transição do ciclo de vida e a dinâmica dos nematócitos em medusas
S Peng, L Ye, Y Li, F Wang, T Sun, L Wang, W Hao… - Iscience, 2023 SE: 5,08
[14] Perfis de transcriptoma e miRNAs revelam rede regulatória e reguladores-chave da formação do xilema secundário em choupo “84K”
H Wang, P Zhao, Y He, Y Su, X Zhou… - Revista Internacional de Ciência Molecular, 2023 IF:5.6
Pagamento e segurança
Suas informações de pagamento são processadas com segurança. Não armazenamos detalhes do cartão de crédito nem temos acesso às informações do seu cartão de crédito.
Investigação
Você também pode gostar
Perguntas frequentes
O produto é apenas para fins de pesquisa e não se destina ao uso terapêutico ou diagnóstico em humanos ou animais. Os produtos e o conteúdo são protegidos por patentes, marcas registradas e direitos autorais de propriedade da
Certos aplicativos podem exigir direitos adicionais de propriedade intelectual de terceiros.

