Descrição
As esferas de seleção de DNA Hieff NGS™ são preparadas com base no princípio SPRI (Solid Phase Reverse Immobilization) e são aplicáveis para purificação de DNA e seleção de tamanho durante a preparação de bibliotecas de sequenciamento de próxima geração (NGS). As esferas de seleção de DNA Hieff NGS™ são compatíveis com vários kits de preparação de bibliotecas de DNA e RNA e são uma boa alternativa de Contas AMPure.
Componentes
| Componentes No. | Nome | 12601ES08 | 12601ES56 | 12601ES75 |
| 12601 | Contas de seleção de DNA Hieff NGS™ | 5 mL | 60 mL | 450 mL |
Especificações
| Linha de produtos | Contas de limpeza e seleção de DNA |
| Matéria-prima inicial | ADN |
| Compatibilidade | ADN |
| Tecnologia de isolamento | Conta magnética |
| Tipo de produto final | ADN |
| Para uso com (aplicativo) | DNA celan up, seleção de tamanho de DNA |
Envio e armazenamento
As contas são enviadas com bolsas de gelo e podem ser armazenadas entre 2°C e 8°C por um ano.
Instruções
- 1. Preparação
Equilibre as esferas de seleção em temperatura ambiente por pelo menos 30 minutos antes de usar.
- 2. Seleção do tamanho do DNA
O fluxo de operação de seleção de tamanho é mostrado na Figura 1 e o protocolo é o seguinte.
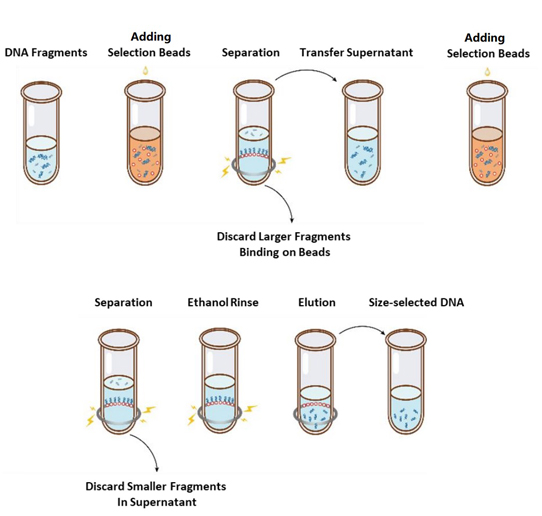
Figura 1. Fluxograma de seleção do tamanho do DNA
2.1 Misture bem as contas agitando ou pipetando para cima e para baixo sempre antes de usar.
2.2 Adicione a primeira rodada de esferas de seleção à amostra (consulte a Tabela 1). Misture bem agitando ou pipetando para cima e para baixo pelo menos 10 vezes.
2.3 Incube em temperatura ambiente por 5 min.
2.4 Centrifugue o tubo brevemente e coloque-o no suporte magnético. Quando a solução estiver límpida (cerca de 5 min), transfira o sobrenadante para um novo tubo de PCR.
2.5 Adicione a segunda rodada de esferas de seleção à amostra da etapa 2.4 de acordo com a Tabela 1. Misture bem agitando ou pipetando para cima e para baixo pelo menos 10 vezes.
2.6 Incube em temperatura ambiente por 5 min.
2.7 Centrifugue o tubo brevemente e coloque-o no suporte magnético. Quando a solução estiver límpida (cerca de 5 min), aspire o sobrenadante e descarte.
2.8 Mantenha o tubo no suporte magnético e adicione 200 μL de etanol 80% recém-preparado sem perturbar as esferas, incubar em temperatura ambiente por 30 seg. Aspire o etanol e descarte.
2.9 Repita a etapa 2.8 uma vez para um total de duas lavagens.
2.10 Remova o etanol residual com pontas de pipeta de 10 µL. Mantenha o tubo no suporte magnético, seque as esferas de seleção ao ar com a tampa aberta até que as rachaduras apareçam (cerca de 5 min).
Observação: não seque demais as esferas de seleção. Isso pode resultar em menor recuperação do DNA alvo.
2.11 Remova o tubo do suporte magnético. Adicione uma quantidade apropriada de ddH2O (≥20 µL) e misture bem agitando ou pipetando para cima e para baixo pelo menos 10 vezes.
2.12 Incube em temperatura ambiente por 5 min.
Gire o tubo brevemente e coloque-o no suporte magnético. Quando a solução estiver límpida (cerca de 5 minutos), transfira 20 μL do sobrenadante para um novo tubo.
- 3. Condições recomendadas para seleção do tamanho do DNA
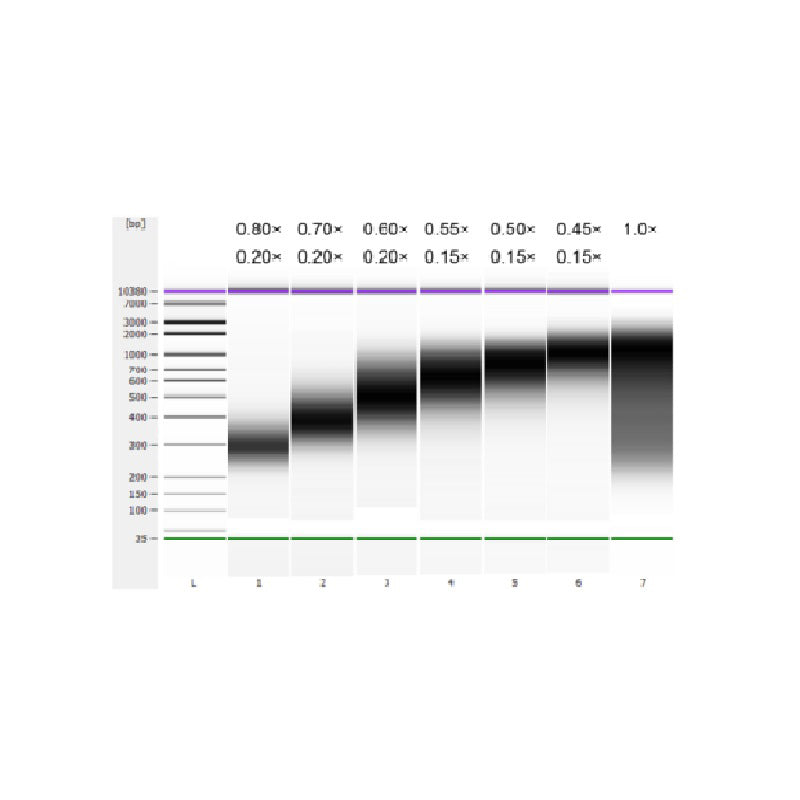
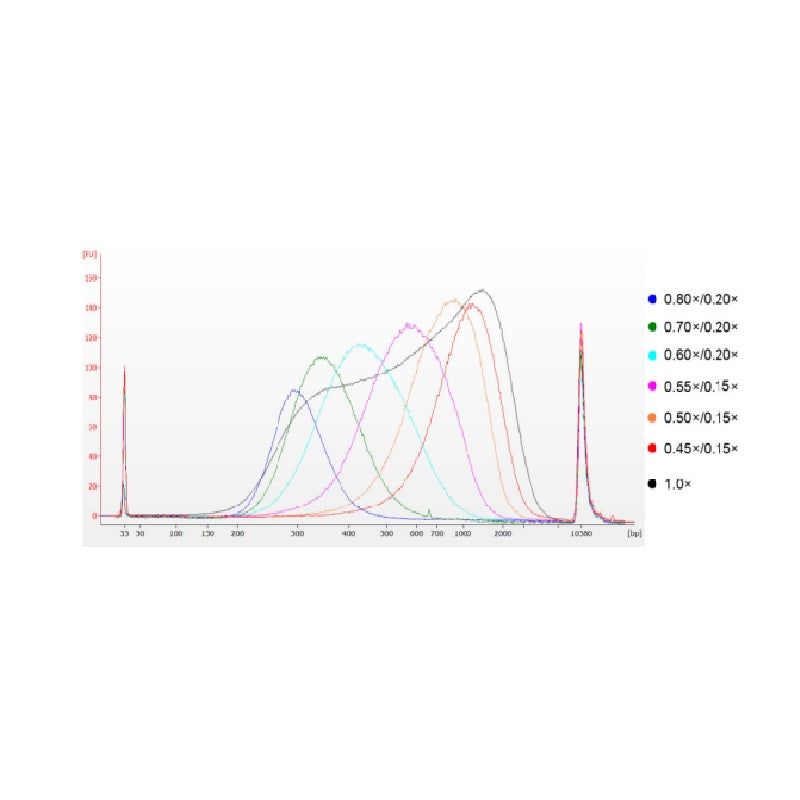
O DNA do timo do bezerro foi fragmentado por sonicação para preparar um fragmento de 100-1.000 pb, e duas rodadas de seleção de tamanho foram realizadas de acordo com a Tabela 1. Os resultados foram analisados usando o Agilent 2100 Bioanalyzer (Figura 2).
Tabela 1. Condição recomendada para seleção do tamanho do DNA
| Comprimento do fragmento de DNA | 250-350 pb | 320-420 pb | 450-550 pb | 550-700 pb | 700-900 pb | 800-1.000 pb |
| Proporção de contas: DNA para a 1ª Rodada | 0,80× | 0,70× | 0,60× | 0,55× | 0,50× | 0,45× |
| Proporção de contas: DNA para o 2º Redondo | 0,20× | 0,20× | 0,20× | 0,15× | 0,15× | 0,15× |
Nota: "×" na tabela indica o volume do DNA da amostra. Por exemplo, se o comprimento de inserção da biblioteca for 250 pb e o volume do DNA da amostra for 100 μL, o volume de esferas magnéticas usadas na primeira rodada de classificação será 0,80×100 μL=80 μL; o volume de esferas magnéticas usadas na segunda rodada de classificação será 0,20×100 μL=20 μL.
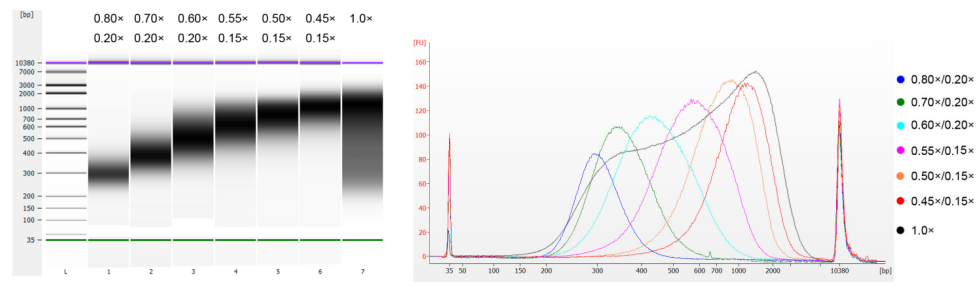
Figura 2. Eletroferograma do chip de DNA de alta sensibilidade Agilent 2100
Notas:
1. Para sua segurança e saúde, use jalecos e luvas descartáveis para a operação.

Citado de "Integração específica de sequência pela família 1 casposase de Candidatus Nitrosopumilus koreensis AR1. Ácidos Nucleicos Res. 2021;49(17):9938-9952. doi:10.1093/nar/gkab725"

Citado de "Infecção recente por Wolbachia altera comunidades microbianas em populações selvagens de Laodelphax striatellus. Microbioma. 2020;8(1):104. Publicado em 2020 Jul 2. doi:10.1186/s40168-020-00878-x"
[1] Wang X, Yuan Q, Zhang W, et al. Integração específica de sequência pela família 1 casposase de Candidatus Nitrosopumilus koreensis AR1. Ácidos Nucleicos Res. 2021;49(17):9938-9952. doi:10.1093/nar/gkab725(IF:16.971)
[2] Duan XZ, Sun JT, Wang LT, et al. A infecção recente por Wolbachia altera as comunidades microbianas em populações selvagens de Laodelphax striatellus. Microbioma. 2020;8(1):104. Publicado em 2 de julho de 2020. doi:10.1186/s40168-020-00878-x(IF:11.607)
[3] Canção B, Almatrafi E, Sang F, et al.Gerenciando sedimentos tratados com Fenton com biochar e composto de esterco de ovelha: efeitos nas características evolutivas da comunidade bacteriana. J Environ Manage. 2022;316:115218. doi:10.1016/j.jenvman.2022.115218(IF:6.789)
[4] Huang C, Mei Q, Lou L, et al. Colite ulcerativa em resposta ao transplante de microbiota fecal por meio da modulação da microbiota intestinal e do equilíbrio de células Th17/Treg. Células. 2022;11(11):1851. Publicado em 5 de junho de 2022. doi:10.3390/cells11111851(IF:6.600)
[5] Ghosh S, Yang X, Wang L, Zhang C, Zhao L. A alimentação prebiótica em fase ativa altera a microbiota intestinal, induz alívio independente do peso da esteatose hepática e do colesterol sérico em camundongos alimentados com dieta rica em gordura. Comput Struct Biotechnol J. 2020;19:448-458. Publicado em 24 de dezembro de 2020. doi:10.1016/j.csbj.2020.12.011(IF:6.018)
[6] Gao X, Yu B, Yu J, et al. Perfil de desenvolvimento da digestão de carboidratos na dieta em leitões. Front Microbiol. 2022;13:896660. Publicado em 29 de abril de 2022. doi:10.3389/fmicb.2022.896660(IF:5.640)
[7] Li P, Zhang Y, Yan F, Zhou X. Características de um bacteriófago, vB_Kox_ZX8, isolado de Klebsiella oxytoca clínica e seu efeito terapêutico na bacteremia de camundongos. Front Microbiol. 2021;12:763136. Publicado em 3 de dezembro de 2021. doi:10.3389/fmicb.2021.763136(IF:5.640)
[8] Lin Z, Luo P, Huang D, Wu Y, Li F, Liu H. Estratégia baseada em multiômicas para análise de toxicidade de acrilamida no modelo Saccharomyces cerevisiae. Chem Biol Interact. 2021;349:109682. doi:10.1016/j.cbi.2021.109682(IF:5.194)
[9] Sun X, Lv W, Wang Y, et al. O gene Mrgprb2 desempenha um papel nas reações anafilactóides induzidas pela injeção de Houttuynia cordata. J Etnofarmacol. 2022;289:115053. doi:10.1016/j.jep.2022.115053(IF:4.360)
[10] Ma H, Lai B, Zan C, Di X, Zhu X, Wang K. GLO1 contribui para a resistência a medicamentos de Escherichia coli por meio da indução do tipo PER de β-lactamases de espectro estendido. Infect Drug Resist. 2022;15:1573-1586. Publicado em 5 de abril de 2022. doi:10.2147/IDR.S358578(IF:4.003)
[11] Zhong Y, Zhao W, Tang Z, et al. Análise transcriptômica comparativa dos diferentes estágios de desenvolvimento do ovário em lagostins vermelhos do pântano Procambarus clarkii. BMC Genomics. 2021;22(1):199. Publicado em 21 de março de 2021. doi:10.1186/s12864-021-07537-x(IF:3.969)
[12] Lian C, Yang H, Lan J, et al. Análise comparativa de genomas de cloroplasto revela relações filogenéticas e variação intraespecífica na planta medicinal Isodon rubescens. PLoS One. 2022;17(4):e0266546. Publicado em 6 de abril de 2022. doi:10.1371/journal.pone.0266546(IF:3.240)
[13] Diao G, Huang J, Zheng X, et al. Prostaglandina E2 desempenha um papel duplo na regulação da migração de células dendríticas. Int J Mol Med. 2021;47(1):207-218. doi:10.3892/ijmm.2020.4801(IF:3.098)
[14] Bing XL, Zhao DS, Peng CW, Huang HJ, Hong XY. Semelhanças e variações espaciais de comunidades bacterianas e fúngicas em populações de cigarrinhas do arroz (Hemiptera: Delphacidae). Insect Sci. 2020;27(5):947-963. doi:10.1111/1744-7917.12782(IF:2.791)
[15] Li X, Zhou S, Zhang J, Zhou Z, Xiong Q. Mudanças direcionais na comunidade bacteriana intestinal em larvas de mosca-soldado-negra (Hermetia illucens). Animais (Basel). 2021;11(12):3475. Publicado em 6 de dezembro de 2021. doi:10.3390/ani11123475(IF:2.752)
[16] Yang J, Peng Y, Kong W. Identificação e sequência completa do genoma do vírus críptico da amoreira 1. Arch Virol. 2022;167(2):687-690. doi:10.1007/s00705-021-05350-1(IF:2.574)
[17] Chang Y, Xia X, Sui L, et al. A colonização endofítica de fungos entomopatogênicos aumenta a resistência das plantas a doenças ao alterar a comunidade bacteriana endofítica. 2021;61(12):1098-1112. doi:10.1002/emprego.202100494(SE:2.281)
[18] Ding CY, Ma YM, Li B, et al. Identificação e análise funcional de genes diferencialmente expressos em Myzus persicae (Hemiptera: Aphididae) em resposta ao trans-anetol. J Insect Sci. 2022;22(1):3. doi:10.1093/jisesa/ieab094(IF:1.857)
Pagamento e segurança
Suas informações de pagamento são processadas com segurança. Não armazenamos detalhes do cartão de crédito nem temos acesso às informações do seu cartão de crédito.
Investigação
Você também pode gostar
Perguntas frequentes
O produto é apenas para fins de pesquisa e não se destina ao uso terapêutico ou diagnóstico em humanos ou animais. Os produtos e o conteúdo são protegidos por patentes, marcas registradas e direitos autorais de propriedade da
Certos aplicativos podem exigir direitos adicionais de propriedade intelectual de terceiros.