Descrição
Ribonuclease R (RNase R) de E. coli, é uma exoribonuclease 3′→5′ dependente de magnésio que pode digere todos os RNAs lineares, mas não digere estruturas de RNA lariat ou circular. A maioria dos RNAs celulares será digerida completamente pela RNase R, com exceção de tRNAs, RNA 5S e lariats de íntron. A RNase R é usada para enriquecimento de circRNAs em amostras biológicas, identificação de sequências de lariat intrônicas, identificação de circRNAs exônicos, estudo de splicing alternativo.
Características
Eficácia e qualidade garantidas com certificação ISO 13485.
Disponível em grandes quantidades ou em configurações personalizadas.
Decompõe efetivamente o RNA linear, preservando os laços de ligação e os RNAs circulares.
A digestão seletiva de RNAs lineares melhora o isolamento de RNAs circulares para produção de proteínas ou construção de biblioteca de cDNA intrônica.
Oferecendo RNAse R de grau GMP
Especificações
| Fonte | Um recombinante E. coli estirpe portadora do gene RNAse R de E. coli |
| Temperatura Ótima | 37℃ |
| Buffer de armazenamento | 50 mM Tris-HCl, 100 mM NaCl, 0,1 mM de EDTA, 0,1% Triton® X-100, 1 mM de DTT, 50% glicerol, pH 7,5 em 25℃ |
| Definição de unidade | Uma unidade converte 1 μg de poli-r(A) em nucleotídeos solúveis em ácido em 10 minutos a 37°C em 20 mM Tris-HCl (pH 8,0), 100 mM KCl e 0,1 mM MgCl2. |
Aplicativo
RNase R serve como um componente fundamental na síntese de RNA circular (circRNA). CircRNA promete melhorar a estabilidade farmacêutica e a bioestabilidade. Com suas estruturas covalentemente fechadas, circRNAs resistem a exonucleases de RNA. [1,2] RNase R encontra ampla aplicação em vários estudos, incluindo splicing alternativo, produção de cDNA de íntron e isolamento de intermediários de splicing e lariats.
Componentes
| Componentes No. | Nome | 14615ES25 | 14615ES72 | 14606ES80 |
| 14615 | RNase R (grau GMP, 20 U/μL) | 25 μL | 250 μL | 1 mL |
Armazenar
Este produto deve ser armazenado entre -25 e -15℃ por 2 anos.
Instruções
Prepare o seguinte sistema de reação em um tubo de microcentrífuga estéril
| componente | Volume |
| Tampão de reação 10×RNase R | 2 μL |
| Amostra de RNA | 1 micrograma |
| RNase R (20 U/μL) | 2-4 você |
| ddH livre de RNase2O | Até 20 μL |
Incube a 37 ℃ por 10 - 30 minutos.
Figuras
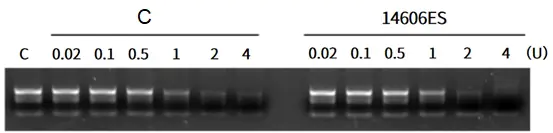
Figura 1: Moléculas de RNA linear podem ser digeridas pela Yeasen RNAse R.
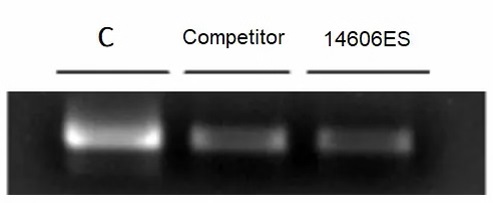
Figura 2: CircRNA não pode ser digerido pela Yeasen RNAse R.
Notas
- A atividade da RNase R requer 0,1-1,0 mM Mg2+;
- Com o aumento do RNA, o tempo de reação pode ser estendido e a quantidade de enzima pode ser aumentada adequadamente;
- O conteúdo de EDTA em amostras de RNA pode afetar a atividade da RNase R.
- Incubar a 70 ℃ por 10 minutos pode inativar a enzima.
Referência
- Qu L, Yi Z, Shen Y, et al. Vacinas circulares de RNA contra SARS-CoV-2 e variantes emergentes. Célula. 2022;185(10):1728-1744.e16. doi:10.1016/j.cell.2022.03.044
- Chen L, Wang C, Sun H, et al. A caixa de ferramentas de bioinformática para descoberta e análise de circRNA. Breve Bioinform. 2021;22(2):1706-1728. doi:10.1093/bib/bbaa001
Pagamento e segurança
Suas informações de pagamento são processadas com segurança. Não armazenamos detalhes do cartão de crédito nem temos acesso às informações do seu cartão de crédito.
Investigação
Você também pode gostar
Perguntas frequentes
O produto é apenas para fins de pesquisa e não se destina ao uso terapêutico ou diagnóstico em humanos ou animais. Os produtos e o conteúdo são protegidos por patentes, marcas registradas e direitos autorais de propriedade da
Certos aplicativos podem exigir direitos adicionais de propriedade intelectual de terceiros.

