Descrição
Hieff NGSTM UCF.EU As esferas de seleção de DNA são preparadas com base no princípio SPRI (Imobilização Reversa em Fase Sólida) e é aplicável para purificação de DNA e seleção de tamanho durante a preparação de bibliotecas de sequenciamento de próxima geração (NGS). Hieff NGSTM UCF.EU As esferas de seleção de DNA são compatíveis com vários kits de preparação de bibliotecas de DNA e RNA. Este produto é fabricado em uma instalação ultralimpa com alto nível de limpeza e pode ser usado para a etapa de purificação de DNA durante processos de detecção de patógenos.
Especificações
| Gato.No. | 17266ES03 /17266ES75 |
| Tamanho | 1 mL /450 mL |
Componentes
| Componentes No. | Nome | 17266ES03 | 17266ES75 |
| 17266 | Hieff NGS UCF.ME Contas de seleção de DNA | 1 mL | 450 mL |
Armazenar
Este produto deve ser armazenado em 2~8℃ por 18 meses.
Envio e armazenamento
As contas são enviadas com bolsas de gelo e podem ser armazenadas entre 2°C e 8°C por um ano.
Instruções
- 1. Preparação
Equilibre as esferas de seleção em temperatura ambiente por pelo menos 30 minutos antes de usar.
- 2. Seleção do tamanho do DNA
O fluxo de operação de seleção de tamanho é mostrado na Figura 1 e o protocolo é o seguinte.
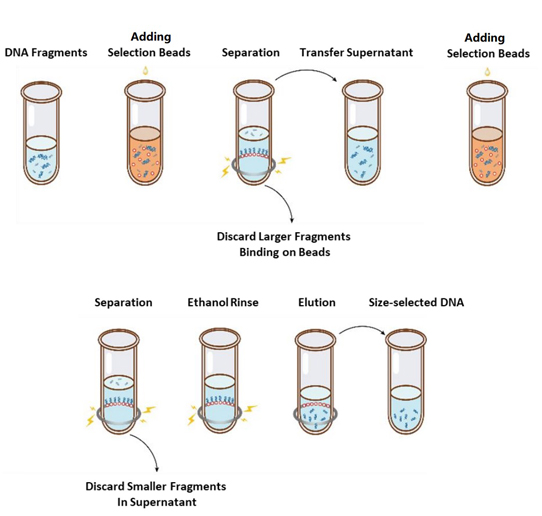
Figura 1. Fluxograma de seleção do tamanho do DNA
2.1 Misture bem as contas agitando ou pipetando para cima e para baixo sempre antes de usar.
2.2 Adicione a primeira rodada de esferas de seleção à amostra (consulte a Tabela 1). Misture bem agitando ou pipetando para cima e para baixo pelo menos 10 vezes.
2.3 Incube em temperatura ambiente por 5 min.
2.4 Centrifugue o tubo brevemente e coloque-o no suporte magnético. Quando a solução estiver límpida (cerca de 5 min), transfira o sobrenadante para um novo tubo de PCR.
2.5 Adicione a segunda rodada de esferas de seleção à amostra da etapa 2.4 de acordo com a Tabela 1. Misture bem agitando ou pipetando para cima e para baixo pelo menos 10 vezes.
2.6 Incube em temperatura ambiente por 5 min.
2.7 Centrifugue o tubo brevemente e coloque-o no suporte magnético. Quando a solução estiver límpida (cerca de 5 min), aspire o sobrenadante e descarte.
2.8 Mantenha o tubo no suporte magnético e adicione 200 μL de etanol 80% recém-preparado sem perturbar as esferas, incubar em temperatura ambiente por 30 seg. Aspire o etanol e descarte.
2.9 Repita a etapa 2.8 uma vez para um total de duas lavagens.
2.10 Remova o etanol residual com pontas de pipeta de 10 µL. Mantenha o tubo no suporte magnético, seque as esferas de seleção ao ar com a tampa aberta até que as rachaduras apareçam (cerca de 5 min).
Observação: não seque demais as esferas de seleção. Isso pode resultar em menor recuperação do DNA alvo.
2.11 Remova o tubo do suporte magnético. Adicione uma quantidade apropriada de ddH2O (≥20 µL) e misture bem agitando ou pipetando para cima e para baixo pelo menos 10 vezes.
2.12 Incube em temperatura ambiente por 5 min.
Gire o tubo brevemente e coloque-o no suporte magnético. Quando a solução estiver límpida (cerca de 5 minutos), transfira 20 μL do sobrenadante para um novo tubo.
- 3. Condições recomendadas para seleção do tamanho do DNA
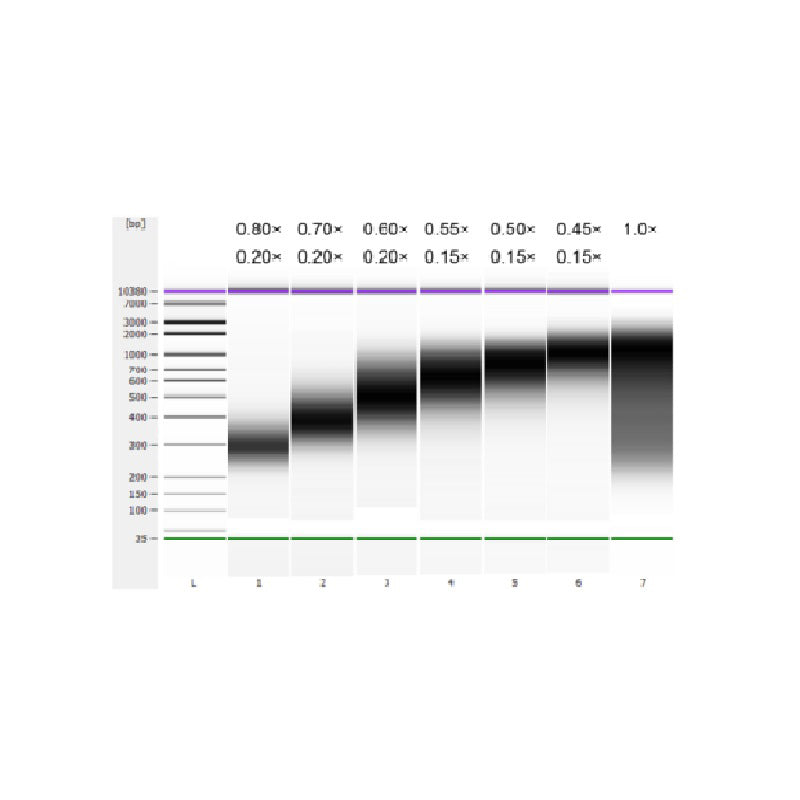
O DNA do timo do bezerro foi fragmentado por sonicação para preparar um fragmento de 100-1.000 pb, e duas rodadas de seleção de tamanho foram realizadas de acordo com a Tabela 1. Os resultados foram analisados usando o Agilent 2100 Bioanalyzer (Figura 2).
Tabela 1. Condição recomendada para seleção do tamanho do DNA
| Comprimento do fragmento de DNA | 250-350 pb | 320-420 pb | 450-550 pb | 550-700 pb | 700-900 pb | 800-1.000 pb |
| Proporção de contas: DNA para a 1ª Rodada | 0,80× | 0,70× | 0,60× | 0,55× | 0,50× | 0,45× |
| Proporção de contas: DNA para o 2º Redondo | 0,20× | 0,20× | 0,20× | 0,15× | 0,15× | 0,15× |
Nota: "×" na tabela indica o volume do DNA da amostra. Por exemplo, se o comprimento de inserção da biblioteca for 250 pb e o volume do DNA da amostra for 100 μL, o volume de esferas magnéticas usadas na primeira rodada de classificação será 0,80×100 μL=80 μL; o volume de esferas magnéticas usadas na segunda rodada de classificação será 0,20×100 μL=20 μL.
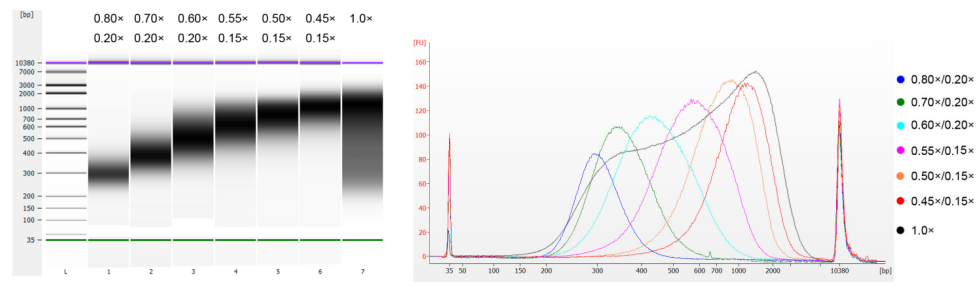
Figura 2. Eletroferograma do chip de DNA de alta sensibilidade Agilent 2100
Notas:1. Para sua segurança e saúde, use jalecos e luvas descartáveis para a operação.
Pagamento e segurança
Suas informações de pagamento são processadas com segurança. Não armazenamos detalhes do cartão de crédito nem temos acesso às informações do seu cartão de crédito.
Investigação
Você também pode gostar
Perguntas frequentes
O produto é apenas para fins de pesquisa e não se destina ao uso terapêutico ou diagnóstico em humanos ou animais. Os produtos e o conteúdo são protegidos por patentes, marcas registradas e direitos autorais de propriedade da
Certos aplicativos podem exigir direitos adicionais de propriedade intelectual de terceiros.