Descrição
O GMyc-PCR Mycoplasma Detection Kit usa principalmente o método PCR para detectar infecção por Mycoplasma de vários materiais biológicos (como cultura de células, secreções de animais experimentais, soro animal, etc.). Ele combina várias vantagens: sensível, específico, rápido e pode ser detectado diretamente com sobrenadantes de cultura de células. Este produto detecta micoplasma em materiais biológicos, como células cultivadas, pelo método PCR. Os primers usados são projetados de acordo com a região conservada da sequência 16S-23S rRNA do micoplasma e amplificam especificamente apenas o DNA do micoplasma, com alta sensibilidade e especificidade de detecção. A amplificação por PCR e a análise por eletroforese levam apenas algumas horas, e a operação é conveniente e simples.
A cultura de células é um experimento comum em pesquisas de ciências biológicas. Ao contrário de outros métodos experimentais comumente usados, a cultura de células é um processo dinâmico contínuo, e as células frequentemente respondem a erros de manipulação ou contaminantes que frequentemente exibem estados celulares anormais ou aparência média. Se estiver contaminada por micoplasma, a morfologia da célula não tem nenhuma mudança óbvia, e é fácil de ser esquecida. Muitas vezes não é encontrada até que a poluição seja muito séria. Pode haver centenas de micoplasmas na membrana celular contaminada, esses micoplasmas competem por nutrientes e liberam metabólitos tóxicos, afetando seriamente os resultados experimentais.
Estudos mostram que pelo menos 20 tipos de micoplasma podem contaminar as células, entre os quais os mais comuns são: Mycoplasma oral (M. orale), Mycoplasma arginina (M.arginina), Mycoplasma hyorhinis (M. hyorhinis), Mycoplasma fermentum (M. fermentans), Mycoplasma hominis (M. hominis), Mycoplasma salivarius (M. salivarium), Mycoplasma pulmonar (M. pulmonis) e pêra Mycoplasma (M. pirum). A taxa de contaminação por micoplasma de células cultivadas varia de 4% a 92%. As fontes de contaminação incluem o ambiente de trabalho, o próprio operador (alguns micoplasmas são flora normal do corpo humano), meio de cultura, soro, contaminação cruzada de células, equipamento experimental e contaminação usada do tecido ou órgão original do qual as células foram preparadas.
Identificar a causa subjacente de problemas durante a cultura de células é uma tarefa difícil e demorada, onde qualquer mudança repentina deve ser suspeitada, e boas práticas de teste e testes regulares para contaminação por micoplasma são necessários. Existem muitos métodos para a detecção de micoplasma, como cultura direta, coloração de fluorescência de DNA, ELISA e métodos de PCR.
Recurso
- Os primers utilizados foram desenhados de acordo com a região conservada da sequência 16S-23S rRNA do Mycoplasma
- Apenas o DNA do micoplasma amplificado especificamente
- Excelente sensibilidade e especificidade
- Mais de 20 espécies de micoplasma podem ser detectadas
Aplicativo
- Detecção de micoplasma
Componentes
| Componentes No. | Nome | 40601ES10 (10 ensaios) | 40601ES20 (20 ensaios) |
| 40601-A | GMyc-1ª mistura de PCR | 250 µL | 2×250 µL |
| 40601-B | Mistura de PCR GMyc-2nd | 250 µL | 2×250 µL |
| 40601-C | Modelo de controle positivo N | 20 µL | 20 µL |
[Notas] 1. Quando não for usado por um longo período, pode ser armazenado congelado a -85~-65℃.
2. A reação de PCR é extremamente sensível.Para evitar falsos positivos, um controle positivo é adicionado no final ao adicionar amostras.
Armazenar
Este produto pode ser armazenado a -25~-15℃ por 18 meses. Se não for usado por um longo período, mantenha-o longe da luz.
Figuras
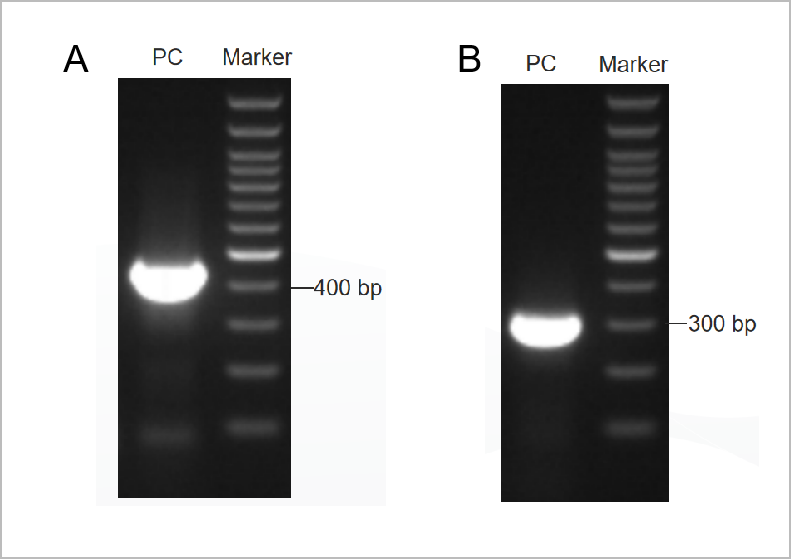
Figura 1. Resultado do eletroforetograma do kit de teste PCR Mycoplama.
O primeiro eletroforetograma da rodada foi mostrado na Figura 1A e o segundo eletroforetograma da rodada foi mostrado na Figura 1B. (M: marcador de 1 kb, 1-2: controles positivos, 3: controle negativo)
[1] Rao XS, Cong XX, Gao XK, et al. A fosforilação mediada por AMPK aumenta a autoinibição de TBC1D17 para promover a captação de glicose dependente de Rab5. Morte celular diferente. 2021;28(12):3214-3234. doi:10.1038/s41418-021-00809-9(IF:15.828)
[2] Guo F, Li L, Li J, et al. Sequenciamento multiômico de célula única de embriões iniciais de camundongo e células-tronco embrionárias. Cell Res. 2017;27(8):967-988. doi:10.1038/cr.2017.82(IF:15.606)
[3] Hao Y, He B, Wu L, et al. Translocação nuclear de p85β promove tumorigênese de câncer mutante de domínio helicoidal PIK3CA. Nat Commun. 2022;13(1):1974. Publicado em 13 de abril de 2022. doi:10.1038/s41467-022-29585-x(IF:14.919)
[4] Shu X, Liu M, Lu Z, et al. O mapeamento de todo o genoma revela que a desoxiuridina é enriquecida no DNA centromérico humano. Nat Chem Biol. 2018;14(7):680-687. doi:10.1038/s41589-018-0065-9(IF:13.843)
[5] Li X, Xiong X, Wang K, et al. Mapeamento em todo o transcriptoma revela metiloma N(1)-metiladenosina reversível e dinâmico. Nat Chem Biol. 2016;12(5):311-316. doi:10.1038/nchembio.2040(IF:12.709)
[6] Sun L, Yang X, Huang X, et al. 2-Hidroxilação de ácidos graxos reprime a tumorigênese colorretal e a metástase por meio do eixo transcricional YAP. Cancer Res. 2021;81(2):289-302. doi:10.1158/0008-5472.CAN-20-1517(IF:12.701)
[7] Sun Z, Zhang Z, Wang QQ, Liu JL. A inativação combinada de CTPS1 e ATR é sinteticamente letal para células cancerígenas com superexpressão de MYC. Cancer Res. 2022;82(6):1013-1024. doi:10.1158/0008-5472.CAN-21-1707(IF:12.701)
[8] Song J, Zhuang Y, Zhu C, et al. Funções diferenciais do PUS10 humano no processamento de miRNA e pseudouridilação de tRNA. Nat Chem Biol. 2020;16(2):160-169. doi:10.1038/s41589-019-0420-5(IF:12.154)
[9] He B, Pan H, Zheng F, et al. O RNA não codificador longo LINC00930 promove a glicólise tumoral mediada por PFKFB3 e a proliferação celular no carcinoma nasofaríngeo. J Exp Clin Cancer Res. 2022;41(1):77. Publicado em 24 de fevereiro de 2022. doi:10.1186/s13046-022-02282-9(IF:11.161)
[10] Tang B, Liu BH, Liu ZY, Luo MY, Shi XH, Pang DW. Pontos quânticos com um revestimento zwitteriônico anfifílico compacto. ACS Appl Mater Interfaces. 2022;14(24):28097-28104. doi:10.1021/acsami.2c04438(IF:9.229)
[11] Huang C, Zhang Z, Chen L, et al.Acetilação dentro dos domínios N- e C-terminais de Src regula papéis distintos da tumorigênese mediada por STAT3. Cancer Res. 2018;78(11):2825-2838. doi:10.1158/0008-5472.CAN-17-2314(IF:9.130)
[12] Wu X, Yu M, Zhang Z, et al. DDB2 regula a replicação do DNA por meio da degradação independente de PCNA de CDT2. Cell Biosci. 2021;11(1):34. Publicado em 8 de fevereiro de 2021. doi:10.1186/s13578-021-00540-5(IF:7.133)
[13] Wang J, Zhang Y, Liu X, Liu H. Otimizando a terapia adaptativa com base na acessibilidade à subpopulação resistente a tumores. Cânceres (Basel). 2021;13(21):5262. Publicado em 20 de outubro de 2021. doi:10.3390/cancers13215262(IF:6.639)
[14] Feng W, Liu R, Xie X, et al. A SUMOilação da α-tubulina é uma nova modificação que regula a dinâmica dos microtúbulos. J Mol Cell Biol. 2021;13(2):91-103. doi:10.1093/jmcb/mjaa076(IF:6.216)
[15] Yu M, Hu X, Yan J, Wang Y, Lu F, Chang J. O inibidor RIOK2 NSC139021 exerce efeitos antitumorais no glioblastoma por meio da indução da parada do ciclo celular mediada por Skp2 e apoptose. Biomedicines. 2021;9(9):1244. Publicado em 17 de setembro de 2021. doi:10.3390/biomedicines9091244(IF:6.081)
[16] Ren S, Cai Y, Hu S, et al. Berberina exerce atividade antitumoral em linfoma difuso de grandes células B modulando o eixo c-myc/CD47. Biochem Pharmacol. 2021;188:114576. doi:10.1016/j.bcp.2021.114576(IF:5.858)
[17] Wen F, Sun X, Sun C, et al. TAGLN é regulado negativamente pela degradação proteassomal mediada por TRAF6 em células de câncer de próstata. Mol Cancer Res. 2021;19(7):1113-1122. doi:10.1158/1541-7786.MCR-20-0513(IF:5.852)
[18] Tang B, Sun EZ, Zhang ZL, et al. Domínio de colesterol sequestrado por esfingomielina recruta proteína de ligação à formina 17 para constrição de fossos revestidos de clatrina na entrada do vírus da gripe. J Virol. 2022;96(5):e0181321. doi:10.1128/JVI.01813-21(IF:5.103)
[19] Hu J, Ren W, Qiu W, et al. Geração de linhagem de células-tronco pluripotentes induzidas (XDCMHi001-A) de um paciente com espondilite anquilosante com mutação JAK2. Stem Cell Res. 2020;45:101788. doi:10.1016/j.scr.2020.101788(IF:4.495)
[20] Xiao S, Yao X, Ye J, Tian X, Yin Z, Zhou L. A modificação epigenética facilita a expressão aberrante da prolina sintase PYCR1 no câncer gástrico [publicado online antes da impressão, 30 de maio de 2022]. Biochim Biophys Acta Gene Regul Mech. 2022;1865(6):194829. doi:10.1016/j.bbagrm.2022.194829(IF:4.490)
[21] Wang J, Zhang Y, Liu X, Liu H. O tratamento periódico fixo é eficaz para o sistema tumoral sem informações completas?. Cancer Manag Res. 2021;13:8915-8928. Publicado em 30 de novembro de 2021. doi:10.2147/CMAR.S339787(IF:3.989)
[22] Yang X, Ren S, Rehman ZU, et al. Caracterização molecular, expressão e identificação funcional da cinase de ligação a TANK 1 (TBK1) da vaca (Bos taurus) e da cabra (Capra hircus). Dev Comp Immunol. 2022;133:104444. doi:10.1016/j.dci.2022.104444(IF:3.636)
[23] Zheng D, Chang X, Liu Y, et al. 2-Metoxi-5((3,4,5-trimetoxifenil)seleninil)fenol reverte a migração e invasão celular induzida por EGF por meio da regulação negativa de MDM2 em linhagens de células de câncer de mama. Cancer Biol Ther. 2019;20(4):513-523. doi:10.1080/15384047.2018.1537578(IF:3.373)
[24] Xu F, Zhang S, Liu Z, et al. TEX9 e eIF3b sinergizam funcionalmente para promover a progressão do carcinoma espinocelular do esôfago. BMC Cancer. 2019;19(1):875. Publicado em 3 de setembro de 2019. doi:10.1186/s12885-019-6071-9(IF:2.933)
[25] Pan H, Sun L, Wang W, et al. RNA não codificador longo sérico LOC553103 como biomarcador diagnóstico e prognóstico não específico para tipos comuns de câncer humano. Clin Chim Acta. 2020;508:69-76. doi:10.1016/j.cca.2020.05.017(IF:2.615)
[26] Li N, Lin SM, Li Y, Sun J, Zhang L, Chen M.Uma linhagem de células-tronco pluripotentes induzidas (GZHMCi004-A) derivada de um feto com mutação heterozigótica G380R no gene FGFR3 causando acondroplasia. Stem Cell Res. 2021;53:102322. doi:10.1016/j.scr.2021.102322(IF:2.020)
[27] Luo Q, Wei C, Long Y, et al. Geração de uma linhagem de células-tronco embrionárias humanas knockout ELTD1 pelo sistema iCRISPR/Cas9. Stem Cell Res. 2021;53:102350. doi:10.1016/j.scr.2021.102350(IF:2.020)
[28] Liu YQ, Ling TW, Wang HY, Yang YH, Song WJ, Wang TC. Geração de uma linhagem de células-tronco pluripotentes induzidas sem integração (LZUSHI001-A) de um paciente epiléptico com mutação DGKG. Stem Cell Res. 2022;61:102768. doi:10.1016/j.scr.2022.102768(IF:2.020)
[29] Chen M, Lin SM, Li N, Li Y, Li Y, Zhang L. Uma linha de células-tronco pluripotentes induzidas (GZHMCi003-A) derivada de um feto com deleção heterozigótica do exon 3 no gene RUNX2 causando displasia cleidocraniana. Stem Cell Res. 2021;51:102166. doi:10.1016/j.scr.2021.102166(IF:2.020)
[30] Xu Y, Wang X, Qiu T, et al. Geração de uma linhagem de células-tronco pluripotentes induzidas (FDCHI007-A) derivada de um paciente com encefalopatia epiléptica e do desenvolvimento Tipo 31 portador de c.545C heterozigoto > Uma mutação no gene DNM1. Stem Cell Res. 2022;60:102709. doi:10.1016/j.scr.2022.102709(IF:2.020)
[31] Fan T, He J, Wang Y, Yu J, Sun W. Geração de uma linhagem de células-tronco pluripotentes induzidas (FDCHi006-A) de uma menina de 7 anos com puberdade precoce central. Stem Cell Res. 2021;56:102542. doi:10.1016/j.scr.2021.102542(IF:2.020)
[32] Gong X, Zheng Z, Yang T, Zheng H, Xiao X, Jia N. Geração de uma linha iPSC corrigida por gene isogênico (OGHFUi001-A-1) de um paciente com encefalopatia epiléptica infantil precoce tipo 1 (EIEE1) com uma mutação hemizigótica R330L no gene ARX. Stem Cell Res. 2022;60:102693. doi:10.1016/j.scr.2022.102693(IF:2.020)
[33] Jia N, Gong X, Chen J, et al. Geração de uma linhagem de células-tronco pluripotentes induzidas (OGHFUi001-A) a partir de uma encefalopatia epiléptica infantil precoce tipo 1 com mutação ARX. Stem Cell Res. 2021;53:102367. doi:10.1016/j.scr.2021.102367(IF:2.020)
[34] Zhu W, Zhou Y, Wang Q, et al. Geração de uma linha de células-tronco pluripotentes induzidas humanas (iPSC) a partir de fibroblastos de pele de um paciente portador de uma mutação E363Q no gene PSEN1. Stem Cell Res. 2022;61:102769. doi:10.1016/j.scr.2022.102769(IF:2.020)
[35] Luo F, Long K, Li X, et al. Deficiência de LRRC8A atenua necrose induzida por hipóxia em células 3T3-L1. Biosci Biotechnol Biochem. 2020;84(6):1139-1145. doi:10.1080/09168451.2020.1730689(IF:1.516)
Pagamento e segurança
Suas informações de pagamento são processadas com segurança. Não armazenamos detalhes do cartão de crédito nem temos acesso às informações do seu cartão de crédito.
Investigação
Você também pode gostar
Perguntas frequentes
O produto é apenas para fins de pesquisa e não se destina ao uso terapêutico ou diagnóstico em humanos ou animais. Os produtos e o conteúdo são protegidos por patentes, marcas registradas e direitos autorais de propriedade da
Certos aplicativos podem exigir direitos adicionais de propriedade intelectual de terceiros.

