Descrição
O kit de detecção de lentivírus competente para replicação (RCL) é usado para detectar quantitativamente lentivírus replicantes que pode ocorrer em um variedade de produtos celulares associados a lentivírus vetores riscos potenciais.
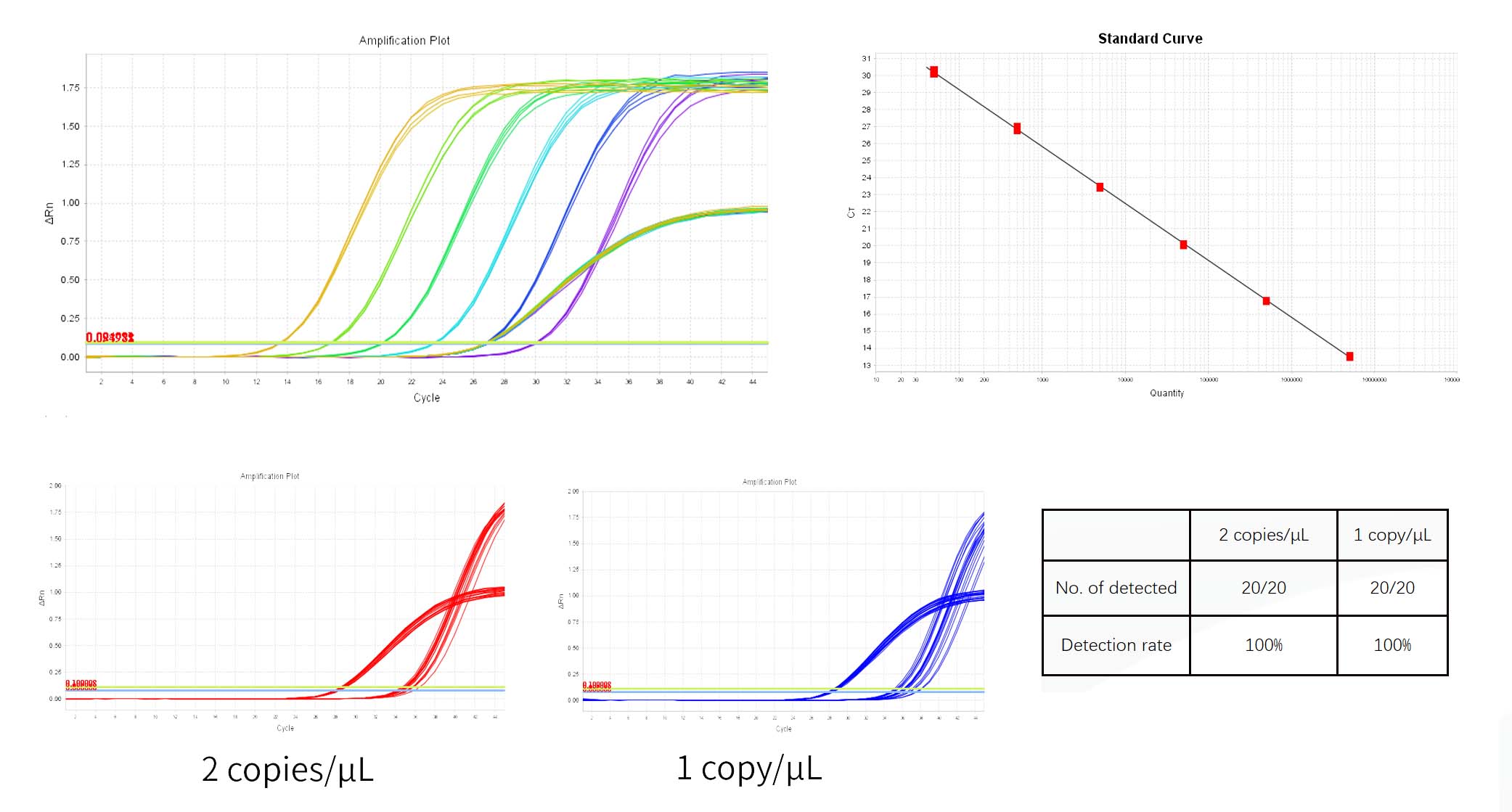
Este kit projeta primers específicos para o Sequência do gene VSV-G de proteínas do envelope lentiviral. E adota taqman sonda fluorescente e o método de reação em cadeia da polimerase (PCR), que tem limite de detecção de nível de 1 cópias/μL e pode detectar específica e rapidamente o risco de lentivírus competente para replicação. O kit precisa para ser usado junto com o Kit de Preparação de Amostras de DNA Residual (Cat# 18461ES).
Componentes
| Componentes No. | Nome | 41311ES50 | 41311ES60 |
| 41311-A | Mistura qPCR RCL | 0,75 mL | 1.5 mL |
| 41311-B | Mistura de primer e sonda RCL | 200 μL | 400 μL |
| 41311-C | Diluição de DNA Tampão | 2×1,8 mL | 4×1,8 mL |
| 41311-D | Controle de DNA RCL(5×10E8 cópias/μL)) | 25 μL | 50 μL |
| 41311-E | CI* | 50 μL | 100 μL |
* CI: Interno controlar.
Armazenar
Este produto deve ser armazenado a -25~-15℃ para 2 anos.
Tanto 41311-A quanto 41311-B devem ser armazenados protegidos da luz.
Aplicável instrumento modelos
Inclui, mas não se limita a: Bio-Rad: Módulo Óptico CFX96; Thermo Scientific: ITB 7500; Estúdio ABI Quant 5; Etapa ABI OnePlus.
Instruções
- ADN RCL Padrão diluição e Padrão curva preparação
O controle de DNA RCL foi diluído em gradiente usando o tampão de diluição de DNA fornecido no kit* , e a diluição concentração é 5×10E7 cópias/μL, 5×10E6 cópias/μL, 5×10E5 cópias/μL, 5×10E4 cópias/μL, 5×10E3 cópias/μL, 5×10E2 cópias/μL, 5×10E1 cópias/μL.
Veja instruções detalhadas abaixo:
1) Descongele o controle de DNA RCL e o tampão de diluição de DNA no gelo. Depois de completamente descongelado, vórtice suavemente para misture, e centrifugar em baixa velocidade por 10 segundos.
2) Retire sete tubos limpos de 1,5 mL, marcados com Std0, Std1, Std2, Std3, Std4, Std5, Std6.
3) Adicionar 90 μL de tampão de diluição de DNA e 10 μL de controle de DNA RCL para o tubo de microcentrífuga de 1,5 mL rotulado Std0, nomeadamente diluir para 5×10E7 cópias/μL. Misture e depois centrifugue por 10 segundos. Embale o padrão de DNA diluído e ele pode ser armazenado em curto prazo (não mais que 3 meses) a -25~-15℃** .Evite congelamento e descongelamento repetidos.
4) Adicionar 90 μL de tampão de diluição de DNA em outros tubos*** , então siga o procedimento abaixo para as diluições seriais**** .
| Tubo | Razão de diluição | Concentração padrão |
| Padrão 1 | 10 μL Std0 + 90 μL Diluição de DNA Tampão | 5×10E6 cópias/μL |
| Padrão 2 | 10 μL Padrão1 + 90 Diluição de DNA μL Tampão | 5×10E5 cópias/μL |
| Padrão 3 | 10 μL Padrão2 + 90 Diluição de DNA μL Tampão | 5×10E4 cópias/μL |
| Padrão 4 | 10 μL Std3 + 90 μL Diluição de DNA Tampão | 5×10E3 cópias/μL |
| Padrão 5 | 10 μL Padrão4 + 90 Diluição de DNA μL Tampão | 5×10E2 cópias/μL |
| Padrão 6 | 10 μL Padrão5 + 90 μL de ADN Diluição Tampão | 5×10E1 cópias/μL |
Tabela 1 Diluição do gradiente padrão
*Três replicar poços são obrigatório para cada concentração.A detecção faixa é 5×10E1 cópias/μL~5×10E6 cópias/μL e esse faixa pode ser expandido se necessário.
** Para reduzir o número de repita congelar-descongelar e evitar contaminação, isso é recomendado para loja o ADN controlar em alíquotas no -25~-15℃ para o primeiro tempo.
*** Uma vez descongelado, ADN diluição amortecedor poderia ser armazenado no 2-8°C para 7 dias, se não usado para um longo tempo, por favor loja no -25~-15℃ .
**** Fazer claro o modelo é completamente misturado, suavemente sacudir o mistura para 15 segundos para 1 minuto para cada gradiente diluição.
- Recuperação de Extração Controlar (ERC) preparação
Defina a concentração de DNA RCL no ERC conforme necessário (a amostra ERC foi preparada com 5×10E4 copia o DNA RCL como um exemplo), como segue:
1) Adicionar 100 μL de amostra de teste em um tubo limpo de 1,5 mL e, em seguida, adicione 10 μL 5×10E3 cópias/μL Padrão de DNA RCL (Std4) e misture bem, marcado como ERC.
2) Realize a extração de DNA da amostra de ERC junto com as amostras de teste para preparar a amostra de ERC purificada.
- Controle Negativo Solução (NCS) preparação
Defina o controle negativo no experimento, as etapas específicas da operação são as seguintes:
1) Adicionar 100 μL de matriz de amostra (ou tampão de diluição de DNA) em um tubo limpo de 1,5 mL, então marcado como NCS.
2) Realizar a extração de DNA do NCS amostra juntamente com as amostras de teste para preparar o NCS purificado amostra.
- Nenhum modelo Controlar (NTC) preparação
Defina o controle sem modelo no experimento, as etapas específicas da operação são as seguintes:
1) O NTC não requer pré-tratamento de amostra e pode ser configurado no estágio de detecção de qPCR de DNA residual contente.
2) A amostra de NTC em cada tubo ou poço é 20 μL Mistura (ou seja, 15 μL RCL qPCR Mistura + 4 μL RCL Primer&sonda Misturar + 1 μL IC) + 10 μL de tampão de diluição de DNA. É recomendado configurar três poços replicados.
- PCR reação sistema
| Componente | Volume(μL) |
| Mistura qPCR RCL* | 15 |
| Mistura de primer e sonda RCL | 4 |
| CI | 1 |
| Modelo de DNA | 10 |
| Total volume** | 30 |
Tabela 2 Sistema de reação
* Calcular o total PCR reação volume por o número de reações: qPCR Mistura =(o número de reações+2) × (15+4+1) μL (incluindo o perdas de dois poços de reação). Mais de três réplicas para cada amostra são recomendadas no experimento.
** Depois tampando o tubo ou selagem o placa, centrífuga o reação tubo ou placa no baixo velocidade para 10 segundos depois suficiente tremendo e misturando para 5 segundos, repita centrifugar para coletar o líquido de o tampa ou parede para o fundo. Evite bolhas durante operação.
Veja a tabela abaixo para a configuração de placa recomendada:
|
| 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | 12 |
| UM | NTC |
| TS 1 | TS 1 | TS 1 |
| Padrão 1 | Padrão 1 | Padrão 1 |
|
|
|
| B | NTC |
| TS 2 | TS 2 | TS 2 |
| Padrão 2 | Padrão 2 | Padrão 2 |
|
|
|
| C | NTC |
| TS 3 | TS 3 | TS 3 |
| Padrão 3 | Padrão 3 | Padrão 3 |
|
|
|
| E |
|
|
|
|
|
| Padrão 4 | Padrão 4 | Padrão 4 |
|
|
|
| E | NCS |
| ERC 1 | ERC 1 | ERC 1 |
| Padrão 5 | Padrão 5 | Padrão 5 |
|
|
|
| F | NCS |
| ERC2 | ERC2 | ERC2 |
| Padrão 6 | Padrão 6 | Padrão 6 |
|
|
|
| G | NCS |
| ERC3 | ERC3 | ERC3 |
|
|
|
|
|
|
|
| E |
|
|
|
|
|
|
|
|
|
|
|
|
Tabela 3 Computador ligado referência quadro
O layout da placa inclui: 6 Std (a curva padrão de 6 concentrações padrão), 1 NTC (sem controle de modelo), 1 NCS (solução de controle negativo), 3 TS (amostras de teste), 3 ERC (controle de extração e recuperação).Três poços replicados para cada amostra.
- Diretrizes de configuração para um PCR Instrumento
As instruções a seguir se aplicam somente a Termo Instrumento qPCR ABI 7500 (Software versão 2.0). Se você usa um instrumento diferente, consulte o guia do instrumento aplicável para obter diretrizes de configuração.
1) Gerar um novo experimento, escolher o modelo de quantificação absoluta ou definido pelo usuário.
2) Crie 1 sonda de detecção, denominada "RCL-DNA", selecione o fluoróforo repórter como "FAM" e o fluoróforo extintor como "nenhum"; crie mais 1 sonda de detecção, nomeie "IC" e selecione o fluoróforo repórter como "CY5" e extinção fluoróforo como "nenhum". A fluorescência de referência é ROX" (a fluorescência de referência pode ser baseada na modelo de instrumento, etc., selecione se você precisa adicioná-lo).
3) No Painel 'Amostras', adicione todas as informações das amostras por vez. Em seguida, selecione os poços, escolha o alvo e as amostras correspondentemente. Definir a tarefa do padrão de DNA RCL como padrão e atribuir o valores 5000000, 500000, 50000, 5000, 500, 50 (a unidade de concentração de DNA em cada poço é cópias/μL) na coluna Quantidade e nomeie o poços Std 1, Std 2, Std 3, Std 4, Std 5, Std 6, correspondentemente. Defina o tarefa do NTC como NTC. Defina o NCS, TS e ERC como Desconhecido, e os nomeou de acordo com o layout da placa acima. Em seguida, clique em Avançar.
4) Defina o programa de amplificação: defina o volume de reação como 30 μL.
| Ciclo de Passo | Temperatura (℃) | Tempo | Ciclos |
| Digestão contaminada | 37℃ | 5 mínimo | 1 |
| Desnaturação inicial | 95℃ | 5 mínimo | 1 |
| Desnaturação | 95℃ | 15 segundo |
45 |
| Recozimento/Extensão (Coleta de fluorescência) | 60℃ | 30 segundos |
Tabela 4 Procedimento de amplificação
- Análise de qPCR resultados
1) O sistema dará automaticamente o Limiar no Painel de gráfico de amplificação de Análise. O Limiar dado pelo sistema às vezes fica muito próximo da linha de base, resultando em uma grande diferença em Ct entre poços replicados. Você pode ajustar manualmente o Limiar para uma posição apropriada e clique Analisar. Então você pode verificar inicialmente se a curva de amplificação é normal no gráfico multicomponente.
2) No Resultado Guia Análise, revise o gráfico da Curva Padrão. Verifique o valores para R2, Eficiência, Inclinação e Interceptação Y. Para uma curva padrão normal, R²>0,99, 90%≤Eff%≤110%, -3,6≤Slope≤-3,1.
3) No 'Visualizar painel de mesa bem em Análise, as concentrações de cada amostra são mostradas em Quantidade, a unidade são cópias/μL, as unidades podem ser convertidas no relatório do ensaio.
4) As configurações de parâmetros de a análise dos resultados precisa ser baseada no modelo específico e no software versão usado e geralmente pode ser interpretado automaticamente pelo instrumento.
5) Calcule a taxa de recuperação de pico com base nos resultados do teste de a amostra TS a ser medido e o pico da amostra recuperação ERC, a taxa de recuperação de picos são necessários estar entre 50%~150%.Fórmula do medidor de taxa de recuperação de pico:
Recuperação (%) = {Sample spiked assay (eg.copies/μL) - Sample assay (eg.copies/μL)} x Volume de eluição (μL) / Teórico valor da quantidade de adição de DNA (por exemplo, cópias) x 100%。
6) O Ct valor de o controle negativo NCS deve ser maior que a média de a menor concentração Ct de o padrão.
7) Modelo de controle livre NTC deve ser indeterminado ou Ct valor ≥38.
Notas
- Este produto é somente para uso em pesquisa.
- Para sua segurança, opere com jalecos e luvas descartáveis.
3. Leia este manual cuidadosamente antes de usar este reagente, e o experimento deve ser padronizado, incluindo manuseio de amostras, preparação do sistema de reação e adição de amostra.
4. Certifique-se de que cada componente esteja totalmente agitado no vórtice e centrifugado em baixa velocidade antes do uso.
Pagamento e segurança
Suas informações de pagamento são processadas com segurança. Não armazenamos detalhes do cartão de crédito nem temos acesso às informações do seu cartão de crédito.
Investigação
Você também pode gostar
Perguntas frequentes
O produto é apenas para fins de pesquisa e não se destina ao uso terapêutico ou diagnóstico em humanos ou animais. Os produtos e o conteúdo são protegidos por patentes, marcas registradas e direitos autorais de propriedade da
Certos aplicativos podem exigir direitos adicionais de propriedade intelectual de terceiros.