DNase I och deras tillämpningar inom biomedicin
Deoxiribonukleas I (DNas I) är ett endonukleas, dess tillämpning är inte bara för att upprätthålla RNA-integritet utan även för DNA-fotavtrycksanalys, generering av slumpmässiga DNA-bibliotek, minskning av klibbighet i cellysat eller proteinextrakt, etc. Kort sagt, DNas I kan användas i nästan alla tillämpningar som kräver enzymklyvning av DNA. Följande är en detaljerad introduktion till DNas I och dess specifika tillämpning.
1. Vad är DNase I?
2. DNas I för framställning av DNA-fri RNA-extraktion
3. DNas I för in vitro-transkription för att avlägsna mall-DNA
4. DNas I för rRNA-borttagning
5. DNas I för DNA-märkning
6. Andra tillämpningar
7. DNase I produktvalsguide
1. Vad är DNase I?
Deoxiribonukleas I (DNas I) är ett ospecifikt endonukleas som kan smälta enkel- eller dubbelsträngat DNA, som finns i olika vävnader och kroppsvätskor. Det kan hydrolysera fosfodiesterbindningar för att producera mono- och oligodeoxinukleotider som innehåller en 5'-fosfatgrupp och en 3'-OH-grupp. Det optimala arbets-pH-intervallet för DNas I är 7-8, dess aktivitet beror på Ca2+ och kan aktiveras av tvåvärda metalljoner såsom Mn2+, Mg2+, Zn2+, etc. I närvaro av Mg2+, klipper DNas I slumpmässigt vilken plats som helst av dubbelsträngat DNA; i närvaro av Mn2+ kan DNas I klippa DNA dubbelsträngat på samma ställe för att bilda en trubbig ände eller en 1-2 nukleotiders klibbiga ändar överhänger.
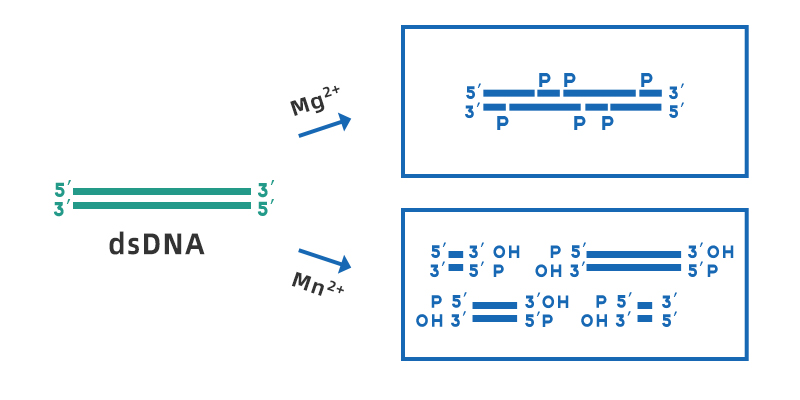
Figur 1. Schematiskt diagram över klyvningen av dsDNA med DNas I i närvaro av Mg2+ och Mn2+.
Även om DNas I-klyvningar generellt anses vara icke-specifika klyvningar, är det mer sannolikt att DNas I agerar på vissa sekvensfragment, såsom minor groove-regionen, och är mer benägen för klyvningar av purin-pyrimidinsekvenser. Men när DNas I verkar på heterogent dsDNA kommer alla fyra baserna att klyvas, och effekten på en specifik bas kommer inte att vara mer än 3 gånger större än den för andra baser.
2. DNas I för framställning av DNA-fri RNA-extraktion
I biologiska experiment är det första steget att förbereda nukleinsyra för att studera olika funktioner hos RNA. Men eftersom DNA och RNA ofta frisätts tillsammans under cellyseringsprocessen, kan interferensen i de två inte undvikas oavsett vilken extraktionslösning som används, så specifika enzymer måste användas för att ta bort interferensen. För högkvalitativ RNA-extraktion används DNas I för att avlägsna resterande DNA från provet.
DNas I kan bryta ned dubbelsträngat och enkelsträngat DNA till oligonukleotider och enkla nukleotider, och DNA:t i RNA-beredningsprodukten kan effektivt brytas ned. DNas I inaktiveras sedan genom uppvärmning med en stoppbuffert. Under uppvärmningsprocessen kan hårnålsstrukturen hos RNA-molekylen öppnas, vilket underlättar det direkta inträdet av RNA:t i den omvända transkriptionsprocessen.
Kvaliteten på RNA kommer att direkt påverka experimentdata i stor utsträckning. I allmänhet kan gDNA-rester inte helt undvikas under RNA-extraktion, därför rekommenderas det i allmänhet att behandla RNA-prover med DNas I för att smälta kvarvarande gDNA innan man utmanar nedströmsapplikationer (t.ex. mRNA-expressionsanalys, transkriptomanalys, etc.). Steget att smälta gDNA kan utföras under RNA-extraktion, efter RNA-extraktion eller före RNA-omvänd transkription.Enligt produktpositioneringen är produkterna som tillhandahålls av Yeasen följande:
Tabell 1: Lista över produkter relaterade till DNA-borttagning av RNA-extraktion eller före omvänd transkription
| Produktpositionering | Produktnamn | Katt # |
| RNA-extraktion | TRIeasy™ Total RNA-extraktionsreagens [fråga] | 10606ES |
| 19221ES | ||
| MolPure™ Plant Plus RNA Kit [fråga] | 19292ES | |
| MolPure™ viralt DNA/RNA-kit [fråga] | 19321ES | |
| gDNA-borttagning | 10325ES | |
| Omvänd transkription | Hifair™Ⅲ1st Strand cDNA Synthesis SuperMix för qPCR (gDNA digester plus) | 11141ES |
| qPCR | 11184ES |
3. DNas I för in vitro-transkription för att avlägsna mall-DNA
In vitro-transkription (IVT) använder huvudsakligen DNA som mall, plus motsvarande substrat och buffertar för att erhålla RNA genom in vitro-transkription. I in vitro-transkriptionsexperiment används vanligtvis RNA-polymeraser som T7, T3 och SP6 för RNA-syntes. Det syntetiserade RNA:t kan ha DNA-rester. Att eliminera DNA-resterna är fördelaktigt för utvecklingen av nedströmsexperiment. Till exempel, i utvecklingsstadiet för mRNA-vaccin, är avlägsnandet av rester ett kritiskt steg, vilket kan minska svårigheten med nedströmsrening och öka produktens renhet. DNA-mallen avlägsnas vanligtvis med användning av rekombinant DNas I (RNas-fri).Enligt mRNA-syntesprocessen är produkterna som tillhandahålls av Yeasen följande:
| mRNA-syntesprocess | Produktnamn | Katt# |
| Förberedelse av mall | Hieff Canace™ Plus High-Fidelity DNA-polymeras [fråga] | 10148ES |
| 10922ES | ||
| 10125ES | ||
| FuniCut™BsaI [fråga] | 15005ES | |
| FuniCut™ XbaI [fråga] | 15033ES | |
| BspQI[fråga] [fråga] | 16215ES | |
| In vitro transkription | 10623ES | |
| 10624ES | ||
| T7 RNA-polymeras (50 U/μL)[fråga] | 10618ES | |
| NTP-uppsättningslösning (ATP, CTP, UTP, GTP, 100 mM vardera) | 10133ES | |
| 10620ES | ||
| 10621ES | ||
| Ta bort mall-DNA | 10611ES | |
| mRNA-modifiering | 10614ES | |
| 10612ES | ||
| 10132ES | ||
| 10619ES | ||
| mRNA-rening | 12602ES |
4. DNas I för rRNA-borttagning
In vivo är rRNA mycket rikligt och mycket konservativt, vilket är av liten betydelse för att erhålla biologisk information, så rRNA tas ofta bort först i RNA-bibliotekskonstruktion och -sekvensering.För närvarande är borttagningsmetoden för rRNA huvudsakligen RNas H-digestion. Huvudstegen för enzymatisk baserad rRNA-utarmning visas i figur 2: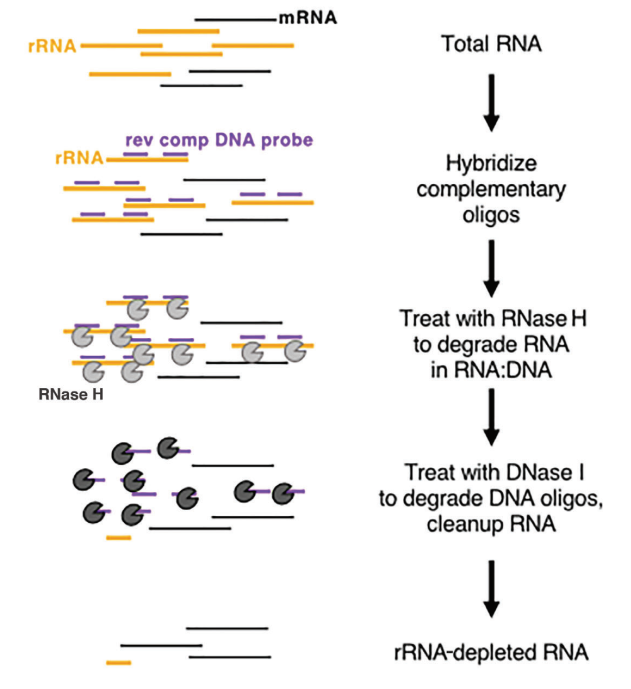
Figur 2: Schematiskt diagram av principen för enzymatisk-baserad rRNA-utarmning (Baldwin, A. et al. 2021, Current Protocols)
Extrahera först totalt RNA, hybridisera sedan enkelsträngad DNA-sond med rRNA, designa och syntetisera rRNA-specifik enkelsträngad DNA-sond, använd sedan RNase H för att bryta ned det hybridiserade rRNA:t och använd DNas I för att bryta ner DNA-proben. Äntligen lämnar icke-rRNA RNA-mallen. Produkterna relaterade till rRNA-borttagning som tillhandahålls av Yeasen är följande:
| mRNA-syntesprocess | Produktnamn | Katt# |
| Människa/mus/råtta rRNA-utarmning | Hieff NGS™ MaxUp rRNA Depletion Kit (människa/mus/råtta) MaxUp [fråga] | 12253ES |
| Plant rRNA utarmning | 12254ES | |
| Avlägsnande av ribosomalt RNA och 45S ITS/ETS-regioner från humant totalt RNA | 12257ES | |
| Nedbrytning av rRNA | 12906ES | |
| Nedbrytning av DNA-sond | 10325ES |
5. DNas I för DNA-märkning
Nick-translationen är en av laboratoriets mest använda metoder för märkning av deoxiribonukleinsyrasond. Denna metod använder olika enzymatiska aktiviteter av DNA-polymeras I för att införliva märkta deoxiribonukleosidtrifosfater i nysyntetiserade DNA-kedjor. Således syntetiseras enhetligt märkta DNA-sonder för hög specifik aktivitet. Egenskaperna för nick-translation är snabb, enkel, avsiktlig, hög specificitet och enhetligt märkta prober, som är lämpliga för längre dubbelsträngat DNA.Metoden realiseras genom den kombinerade verkan av DNas I och E. coli DNA-polymeras I. Huvudstegen för DNA-märkning genom nick-translation visas i figur 3:
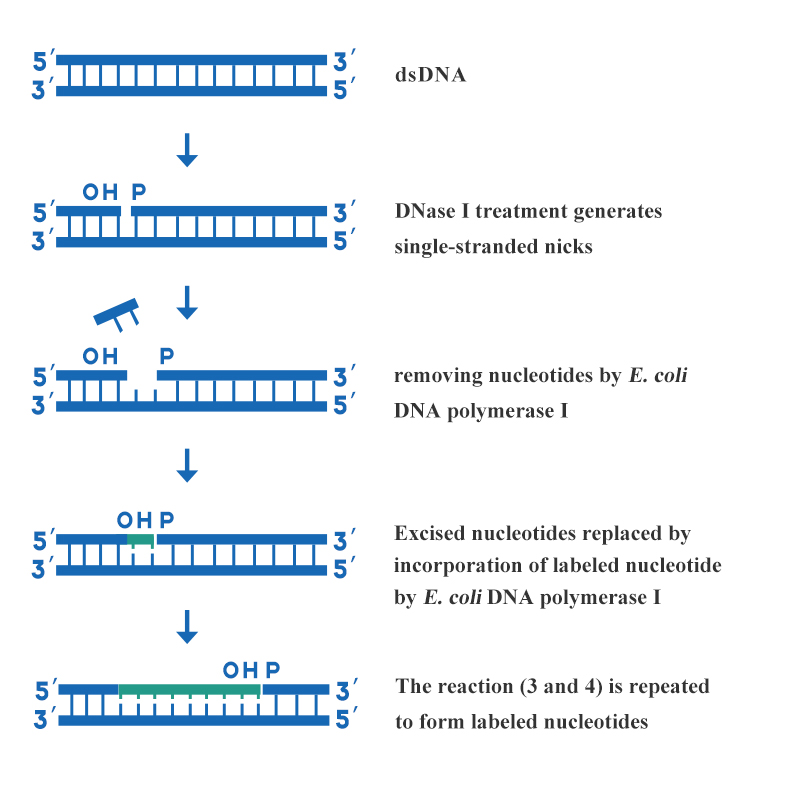
Figur 3: Schematiskt diagram av DNA-märkning genom nick-translation
En lämplig koncentration av DNas I används för att skapa flera enkelsträngade luckor på varje sträng av det dubbelsträngade DNA som ska märkas, och 3'-hydroxylterminalen bildas vid gapet. Använd 5'→3'-exonukleasaktiviteten av E. coli DNA-polymeras I för att skära en nukleotid från 5'-änden av hacket, och samtidigt introducerar 5'→3'-polymerasaktiviteten av E. coli DNA-polymeras I en nukleotid märkt med 3'-änden av gapet för att reparera gapet. När gapet rör sig längs DNA-strängen, inkorporeras de märkta nukleotiderna i den nyligen syntetiserade strängen.Produkterna relaterade till DNA-märkning som tillhandahålls av Yeasen är följande:
| Produktpositionering | Produktnamn | Katt# |
| Vanlig | Deoxiribonukleas I (DNas I) från bovin pankreas [fråga] | 10607ES/10608ES |
| RNase fri | 10325ES | |
| E.coli källa | 12903ES |
6. Andra tillämpningar
Ovanstående är flera vanliga applikationer. Fler tillämpningar av DNas I inkluderar följande, såsom DNas I-footprinting-analys och DNas I-överkänsliga ställen. DNase I footprinting assay är en detektionsmetod som exakt kan identifiera bindningsställena för DNA-bindande proteiner på DNA. När ett protein binder till ett DNA-fragment kan det skydda bindningsstället från att skadas av DNas I och DNA-fragmenten kommer att lämnas kvar efter enzymdigestion ("fotavtryck") och dess sekvens kan bestämmas. I gelbilden finns inga band där DNA:t binder till proteinet. För att läsa mer klicka på länken.DNas I-överkänsliga ställen avser klyvningar vid ett litet antal specifika ställen när kromatin behandlas med lågt DNas I och dessa specifika ställen kallas DNas I-överkänsliga ställen. Principen är att när en gen är i ett transkriptionellt aktivt tillstånd, är kromatinet som innehåller genen betydligt mer känsligt för DNas-nedbrytning än den inaktiva regionen. För att läsa mer klicka på länken.
7. DNase I produktvalsguide
Yeasen Biotechnology (Shanghai) Co., Ltd., grundat 2014, är ett högteknologiskt företag som är engagerat i FoU och produktion av verktygsenzymråmaterial och antigenantikroppar. Dess produkter inkluderar molekylära diagnostiska enzymer, proteiner och antikroppar som används i läkemedel, livsmedelssäkerhetstestning, avel, rättvisa och andra industrier. Vi har åtagit oss att förse kunder inom biovetenskap med högkvalitativa produkter och tjänster. Riktlinjerna för produktköp för DNase I är följande:| Produktnamn (Cat#) | Produktpositionering | Rekommenderade applikationer |
| DNas I från bovin bukspottkörtel (CAT#10607,10608)[fråga] | RNase borttaget, inte upptäckt | Används främst inom proteinforskning: DNA-borttagning från proteinpreparat. |
| Rekombinant DNas I (RNas-fri)(CAT#10325) | RNase-fri, för forskning | Idealisk för en mängd olika applikationer: DNA-borttagning från RNA och proteinberedningar som RNase-känsliga cDNA-bibliotek eller provberedning för RT-PCR-experiment. |
| RNase-fri, GMP farmaceutisk kvalitet. | Idealisk för en mängd olika applikationer: DNA-borttagning från RNA och proteinberedningar som RNas-känsliga cDNA-bibliotek eller provberedning för RT-PCR-experiment. |
Angående läsning:
GMP-grade reagens för mRNA in vitro syntes
Principerna för DNase I-footprinting och dess biomedicinska tillämpningar
Referenser
1. Baldwin A, Morris AR, Mukherjee N. En enkel, kostnadseffektiv och skalbar metod för att utarma humant ribosomalt RNA för RNA-seq[J]. Nuvarande protokoll, 2021.
2. Song C, Zhang S, Huang H. Att välja en lämplig metod för identifiering av replikationsursprung i mikrobiella genom[J]. Frontiers in Microbiology, 2015, 6:1049.
