Vad ska man göra när qPCR-resultaten är konstiga?
qPCR-experiment kan kännas lätta och ibland svåra. Efter att primerdesignen är klar läggs qPCR-prover till steg för steg som PCR, och experimentet kan slutföras på maskinen. Det finns flera vanliga problem i qPCR-experiment med färgmetod och med tanke på möjliga orsaker och lösningar hoppas vi att du kan använda dem för att lösa problemen i qPCR-experiment.
1. Vad är qPCR?
2. Onormal qPCR-amplifieringskurva
3. Onormal qPCR-smältkurva
4. Beställningsinformation
5. Relaterade produkter
6. Angående läsning
1. Vad är qPCR?
Sedan polymeraskedjereaktionstekniken (PCR) uppfanns är PCR förmodligen den mest använda tekniken inom molekylärbiologi på grund av dess enkelhet, billighet, tillförlitlighet, snabbhet och höga känslighet. qPCR är en teknik utvecklad av PCR-teknik. Under DNA-amplifieringsprocessen har metoden för att detektera den totala mängden produkter efter varje PCR-cykel med fluorescerande färgämnen inte bara snabbheten och känsligheten hos PCR utan har också mer specificitet och Hög, realtidsövervakning, repeterbar och exakt kvantifiering och andra fördelar. qPCR är en teknik som använder en kvantitativ realtids-PCR-amplifieringsenhet, nämligen ett qPCR-instrument, för att övervaka nukleinsyraamplifieringsprodukter i realtid under PCR-processen. qPCR realiserar den kvantitativa analysen av startmallen genom analys av Ct-värdet och standardkurvan. 1992 föreslog japanska Higuchi först "realtidsfluorescerande kvantitativ PCR-teknik". 1996 lanserade American Biological Company världens första fluorescerande kvantitativa PCR-instrument, som är sammansatt av ett PCR-amplifieringstermiskt cykelsystem, optiskt system för fluorescensdetektering och dator- och applikationsprogramvara. Den kan övervaka nukleinsyraamplifieringsprodukter i realtid genom fluorescerande färgämnen eller fluorescerande prober. , genom det matematiska funktionsförhållandet, kombinerat med programvaran för resultatanalys, realiseras beräkningen av den initiala mallmängden av provet som ska testas. Därför har realtidsfluorescerande kvantitativ PCR-teknik använts i stor utsträckning. Fluorescerande märkningsmetoder för qPCR är uppdelade i fluorescerande färgmosaikmetod baserad på SYBR Green I-färgmetoden, fluorescerande sondmetod baserad på Taqman-probmetoden (Cycling Probe, Molecular Bracon, etc.), quencher dye primer-metod.
qPCR-experiment kan kännas lätta och ibland svåra. Efter att primerdesignen är klar läggs qPCR-prover till steg för steg som PCR, och experimentet kan slutföras på maskinen. Många små detaljer kräver dock uppmärksamhet, som att ingen vätska hänger på rörväggen, inga bubblor i röret och behovet av att minimera den direkta kontakten mellan rörväggen, rörlocket etc. för att minska experimentella fel eller konstiga experimentella resultat. Xiaoyi har sammanställt flera vanliga problem i qPCR-experiment med färgmetoden och givet de möjliga orsakerna och lösningarna, som vi hoppas att du kan använda för att lösa problemen i qPCR-experiment.
2. Onormal qPCR-amplifieringskurva
2.1 Avvikelser i baslinjefasen för den logaritmiska kurvan för amplifieringskurvan
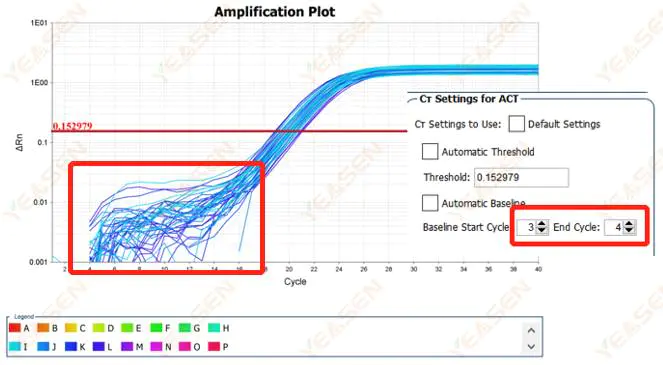
Figur 1. Amplifieringsdiagram
Möjliga orsaker:
Felaktig grundinställning.
Lösningar:
Det rekommenderas att höja ändpunktsvärdet för baslinjen.Justeringsexempeldiagrammet är som följer.

Figur 2. Amplifieringsdiagram
2.2 Segmentering av kurvan för amplifieringskurvans log-kurva
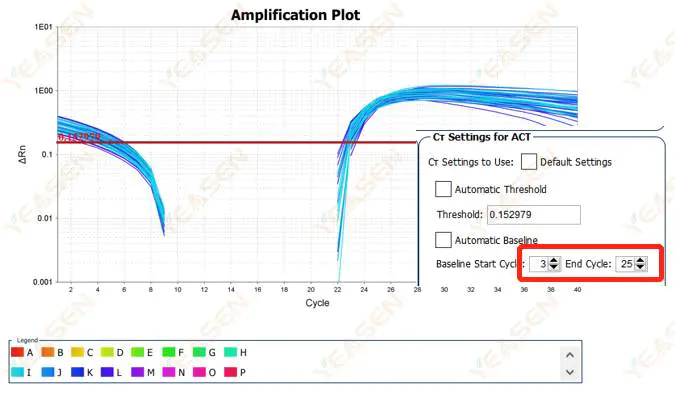
Figur 3. Amplifieringsdiagram
Möjliga orsaker:
Baslinjen är för högt inställd.
Lösningar:
Det rekommenderas att reducera ändpunktsvärdet för baslinjen. Justeringsexempeldiagrammet är som följer.
2.3 Ojämn förstärkningskurvans linearitetsdiagram
Figur 4. Amplifieringsdiagram
Möjliga orsaker:
1) PCR-reaktionsröret var inte tätt tillslutet och reaktionslösningen läckte.
2)PCR-reaktionslösning med hängande vägg.
3) Instrumentet är inte kalibrerat (inklusive automatisk kalibrering eller ROX-kalibrering).
4) Systemet har många inhibitorer, vilket leder till instabil fluorescens.
5) Överdriven användning av instrumentet resulterar i instabil fluorescensuppsamling.
Lösningar:
1)Tryck till rörlocket hårt.
2) Blanda reagenset noggrant och lägg det försiktigt i det kvantitativa instrumentet efter full centrifugering.
3) Kalibrera instrumentet.
4) Förbättra RNA-renheten och välj lämpliga reagenser för omvänd transkription.
2.4 Linjär graf för amplifierad kurva sicksackar vid en platå
Figur 5. Amplifieringsdiagram
Möjliga orsaker:
1) Renheten hos RNA är dålig och det finns många föroreningar.
2) Instrumentet har använts för länge.
Lösningar:
1) Återextraktera högkvalitativt RNA.
2) Späd RNA-mallen för att minska koncentrationen av föroreningar.
3) Kalibrera instrumentet.
2.5 Linjär plot av amplifieringskurvan når inte en platå
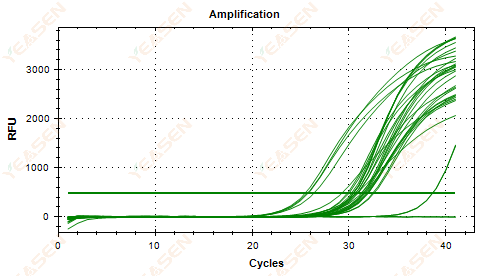
Figur 6. Amplifieringsdiagram
Möjliga orsaker:
1) Låg mallkoncentration (Ct-värde runt 35).
2) För få förstärkningscykler.
3) Låg effektivitet av reagensamplifiering (liten Ct, men kan inte nå platå).
Lösningar:
1) Öka koncentrationen av mallen.
2) Öka antalet cykler.
3) Ökande Mg2+ koncentration.
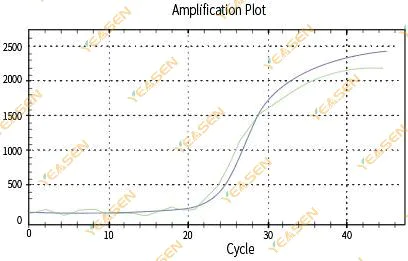
2.6 Amplifieringskurvan linjär plottplatå hängande
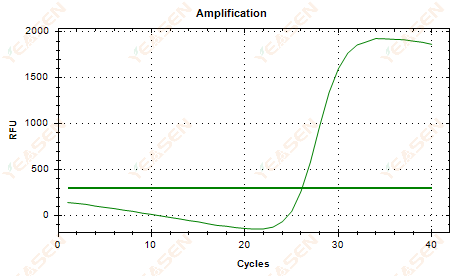
Figur 7. Amplifieringsdiagram
Möjliga orsaker:
1) Förekomst av nedbrytning (förstärkningsproduktnedbrytning, SYBR-nedbrytning).
2) Rörets lock är inte ordentligt täckt och reagenset förflyktigas.
3) cDNA-koncentrationen är för hög (om Ct-värdet är för litet dras fluorescenströskeln högt och hängningen är allvarligare).
4) Luftbubblor i röret, sedan försvinner de.
Lösningar:
1) Förbättra systemets renhet.
2) Minska cDNA-mängden (utspädd mall).
3) Minska slutpunktsvärdet för baslinjen.
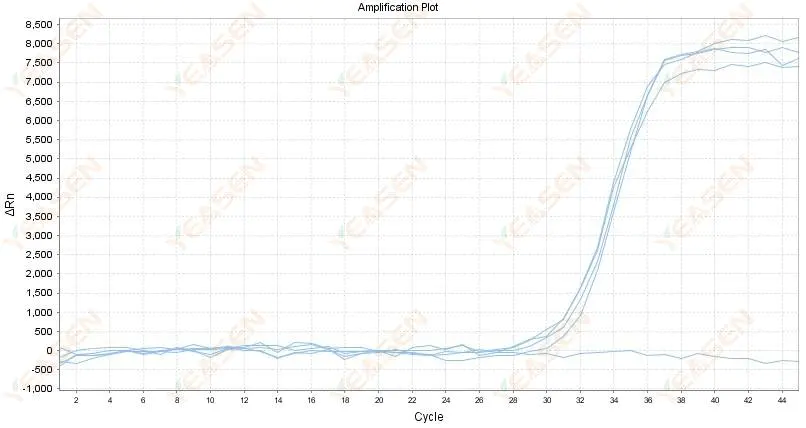
2.7 Stort värde på Ct i amplifieringskurvan
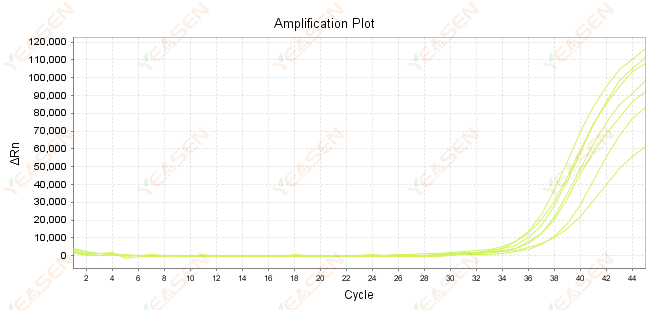
Figur 8. Amplifieringsdiagram
Möjliga orsaker:
1) Låg mängd mallar.
2) Låg förstärkningseffektivitet.
3) PCR-fragmentet är för långt.
4) Förekomst av inhibitorer i reaktionssystemet.
Lösningar:
1) Minska utspädningstiderna eller öka mängden mall så att Ct-värdet faller mellan 15 och 30 så mycket som möjligt.
2) Optimera reaktionsförhållandena, prova en amplifieringsprocedur i tre steg eller designa om primers.
3) PCR-produktens längd är utformad för att vara inom 100 -150 bp, rekommenderas inte att överstiga 300 bp.
4) Reproduktion av mallar med högre renhet.
2.8 Dålig repeterbarhet av amplifieringskurvor
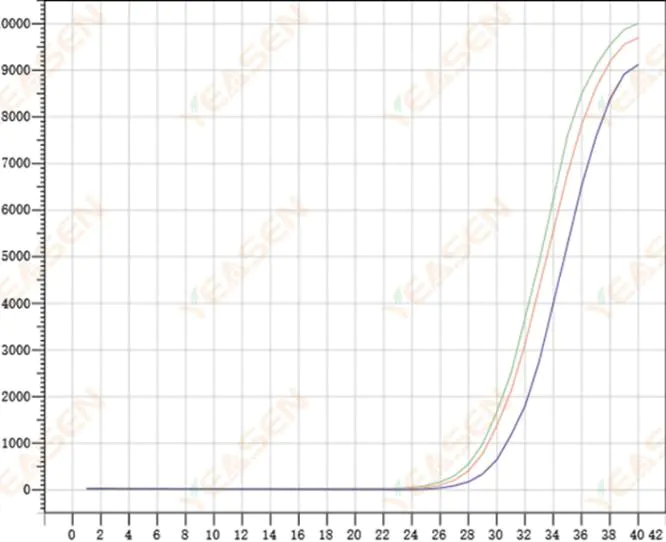
Figur 9. Amplifieringsdiagram
Möjliga orsaker:
1) Stora spikfel.
2) Reagenser och system är inte väl blandade.
3) Lågt antal kopior av cDNA.
4) Ingen Rox-kalibrering användes.
Lösningar:
1) Kalibrering av pipetter.
2) Noggrann blandning av reaktionssystemet.
3) Låg mallkoncentration, dålig repeterbarhet, 4-6 sammansatta hål och 1-2 avvikelser av större värden kan kasseras på lämpligt sätt.
4) Det är bäst att använda Rox-kalibrering. Om det använda reagenset inte innehåller Rox, måste referensfärgämnet väljas som Ingen.
2.9 Slumpmässiga förstärkningskurvor
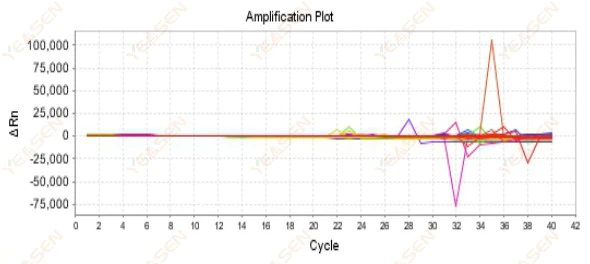
Figur 10. Amplifieringsdiagram
Möjliga orsaker:
Missmatchningen mellan Rox-koncentrationen och modellen.
Lösningar:
Ändra referensfärgämnesinställningen på instrumentet från ROX till NONE och se om amplifieringskurvan återgår till det normala.
2.10 NTC Peaking
2.10.1 Ct>35, smältkurva Tm-värde <80℃
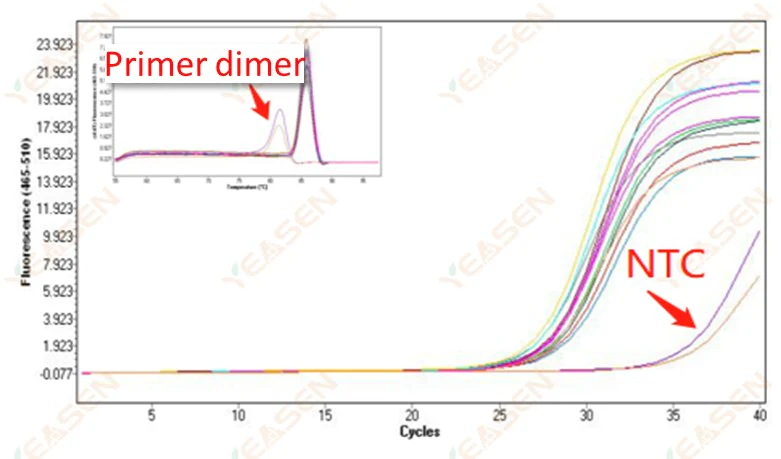
Figur 11. Amplifieringsdiagram
Möjliga orsaker:
Primerdimeriseringsresultat.
Lösningar:
Optimerade primers.
2.10.2 Ct-värde <35, NTC-smältkurvan och gensmältkurvans toppform överlappar varandra
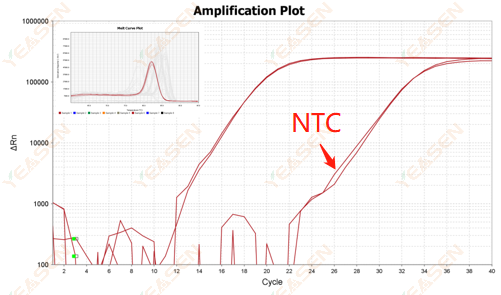
Figur 12. Amplifieringsdiagram
Möjliga orsaker:
Kontaminering av reaktionssystem.
Lösningar:
Kontrollera föroreningskällorna en efter en.
3. Onormal qPCR-smältkurva
3.1 Enstaka toppsmältkurva men inte skarp
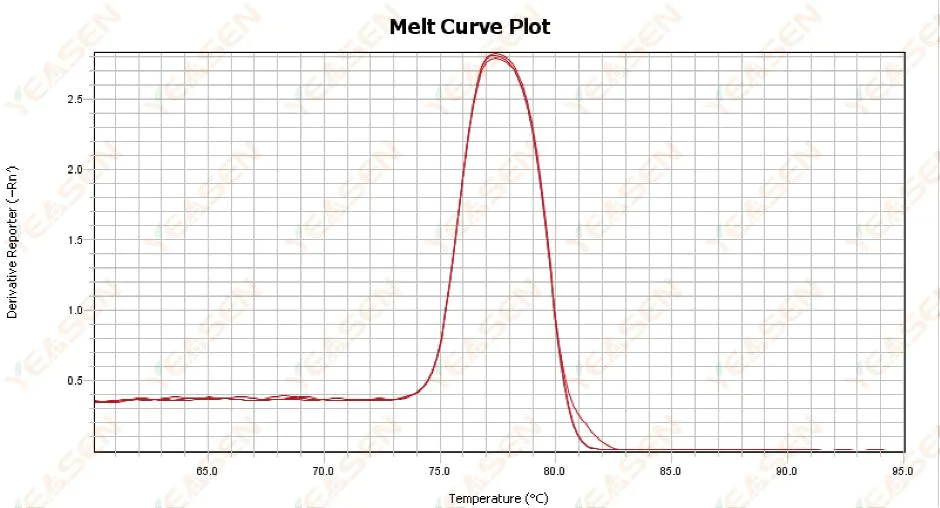
Figur 13. Plott för smältkurva
Möjliga orsaker:
1) Det är relaterat till reagenssammansättning och instrumentmodell.
2) Närvaro av icke-specifik amplifiering av fragment av liknande storlek.
Lösningar:
1) Temperaturintervallet från toppstart till toppstopp är inte högre än 7°C. Resultaten anses användbara, dvs enstaka topp.
2) Högkoncentrerad agaroselektrofores (t.ex. 3 % agaros) utfördes för att hjälpa till vid bestämningen.
3.2 Smältkurvan är dubbeltoppad och den nedre toppen Tm är före 80°C
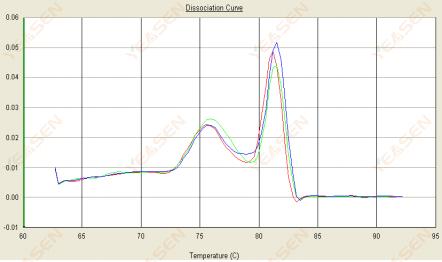
Figur 14. Dissociationskurva
Möjliga orsaker:
Närvaro av primerdimerer.
Lösningar:
Öka glödgningstemperaturen, minska primerkoncentrationen eller designa om primers.
3.3 Smältkurvan är dubbeltoppad och den nedre toppen Tm är efter 80°C
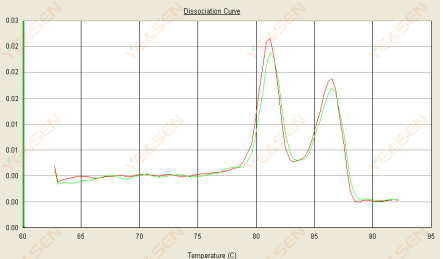
Figur 15. Dissociationskurva
Möjliga orsaker:
1) Dålig primerspecificitet leder till amplifiering av icke-specifika produkter.
2) gDNA-kontamination.
Lösningar:
1) Sprängkontrollera primerspecificiteten, omdesign om den är dålig.
2) Bekräftelse av NRC:s negativa kontroll, om någon, måste återförbereda mallen.
3.4 Smutsiga toppar i smältkurvan
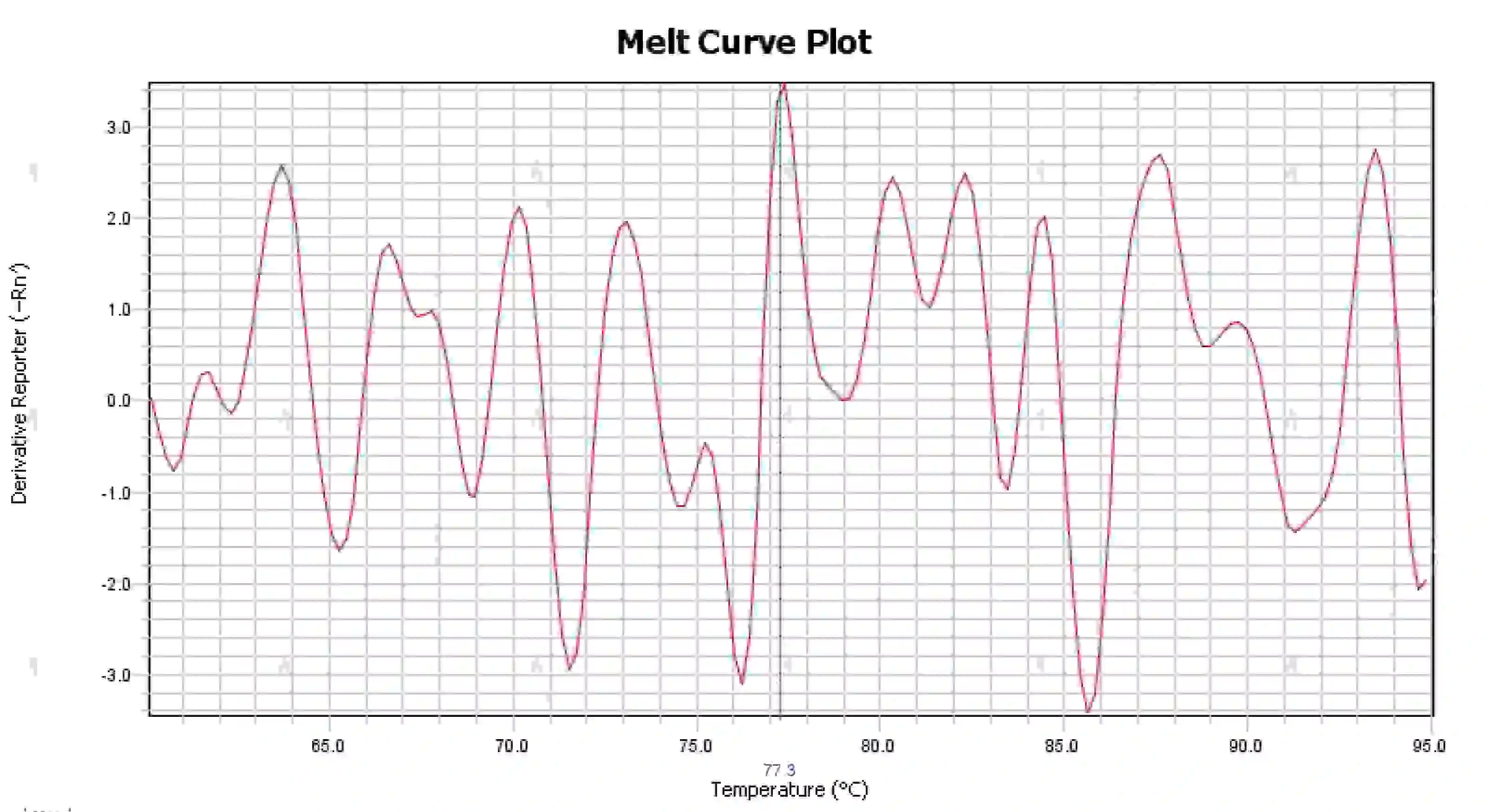
Figur 16. Plott för smältkurva
Möjliga orsaker:
1) Kontaminering av reaktionssystemet.
2) Exponering av reagenser för starkt ljus eller hög temperatur leder till reagensfel.
3) Instrumentet har inte kalibrerats på länge.
4) Missmatch mellan förbrukningsvaror och instrument.
Lösningar:
1) Genom att kombinera NTC- och NRC-resultat för att bekräfta kontaminering, rekommenderas att utesluta kontaminering från vatten, primers, enzymer och miljön en efter en.
2) Det rekommenderas att göra jämförelseexperiment med nya reagenser.
3) Regelbundet underhåll av instrumentkalibrering rekommenderas.
4) Bekräfta kraven för motsvarande instrument för förbrukningsvaror.
3.5 Falska toppar i främre änden av smältkurvan
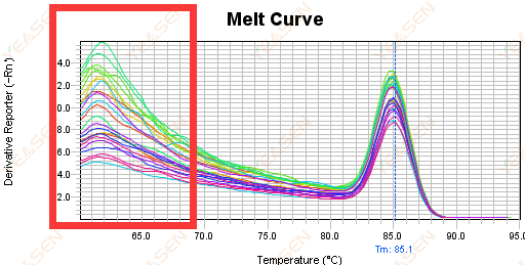
Figur 17. Plott för smältkurva
Möjliga orsaker:
Rox-koncentrationen matchar inte modellen.
Lösningar:
Det rekommenderas att avbryta Rox-korrigeringen för att se om smältkurvan är normal.
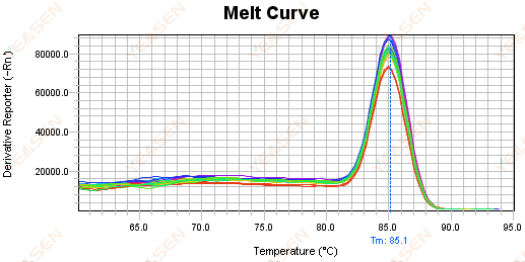
Figur 18. Plott för smältkurva
4. Beställningsinformation
Tabell 1. Beställningsinformation
| Produktnamn | Produktkod | Specifikation |
| Hieff Unicon™ Universal Blue qPCR Master Mix (färgämnebaserad) | 11184ES03 | 1 ml |
| 11184ES08 | 5×1 ml | |
| 11184ES50 | 50×1 ml | |
| 11184ES60 | 100×1 ml |
5. Relaterade produkter
Produkterna som tillhandahålls av Yeasen är följande.
Tabell 2. Relaterade produkter
| Produktpositionering | Produktnamn | Produktkod |
| Ettstegs gDNA-borttagning och omvänd transkription | Hifair™V ettstegs RT-gDNA-digestion SuperMix för qPCR (Förfrågan) | 11142ES |
| High Sensitivity Master Mix (med steg för borttagning av gDNA) | Hifair™ III 1st Strand cDNA Synthesis SuperMix för qPCR (gDNA digester plus) | 11141ES |
| High Sensitivity Kit (med steg för borttagning av gDNA) | Hifair™ III 1st Strand cDNA Synthesis Kit (gDNA digester plus) (Förfrågan) | 11139ES |
| Vanlig kvantitativ mastermix (probmetod) | Hieff™ qPCR TaqMan Probe Master Mix (Förfrågan) | 11205ES |
6. Angående läsning
DNase I och deras tillämpningar inom biomedicin
Riktlinjen för val av omvänt transkriptas
