Hur väljer jag dina NGS-adaptrar?
NGS Adapter, en viktig del av nästa generations sekvenseringsbibliotek, spelar en roll för att koppla ihop det testade DNA-fragmentet och Flow-cellen (Sequencing chip). Ledningens effektivitet är en viktig faktor för att bestämma bibliotekets kvalitet och utbyte. Så vad är en NGS-adapter? Vilka är de vanligaste typerna av NGS-adaptrar? Och hur väljer man rätt NGS-adaptrar för dina sekvenseringsplattformar?
1. Vad är NGS Adapter?
2. Vilka faktorer bör du tänka på när du väljer NGS-index?
3. Vilka är de vanligaste typerna av index?
4. Vilka är de vanligaste typerna av NGS-adaptrar?
- UMI-adapter
- Kompletta adaptrar
- Ofullständiga adaptrar
- Tn5-adaptrar
5. Hur väljer man rätt NGS-adaptrar för dina sekvenseringsplattformar?
6. Angående läsning
1. Vad är NGS Adapter?
NGS Adapter, en serie adaptrar i sekvensering, är en kort nukleotidsekvens med en känd sekvens. Det ligeras till båda ändarna av målnukleinsyrafragmentet. Under sekvensering börjar den sekvensering genom att hybridisera med den kända sekvensen på Flow-cellen för att kombinera biblioteket med chipet. Så vad är strukturen för NGS-adaptern?
Med Illumina-plattformen som exempel kan en NGS-adapter delas in i tre delar:
P5 och P7: Sekvens kombinerad med P5 och P7 slutar på Flow-cellen för att fixera biblioteket på sekvenseringschipet, vilket underlättar klusterreaktion genom Bridge-PCR.
Rd1 SP och Rd2 SP (Read1/Read2-sekvenseringsprimer): Bindande regioner av sekvenseringsprimrar, som indikerar positionen där sekvensen börjar läsas.
Index (även känd som streckkod): en känd syntetisk sekvens som används för att särskilja olika prover i sekvenseringen av det blandade biblioteket.
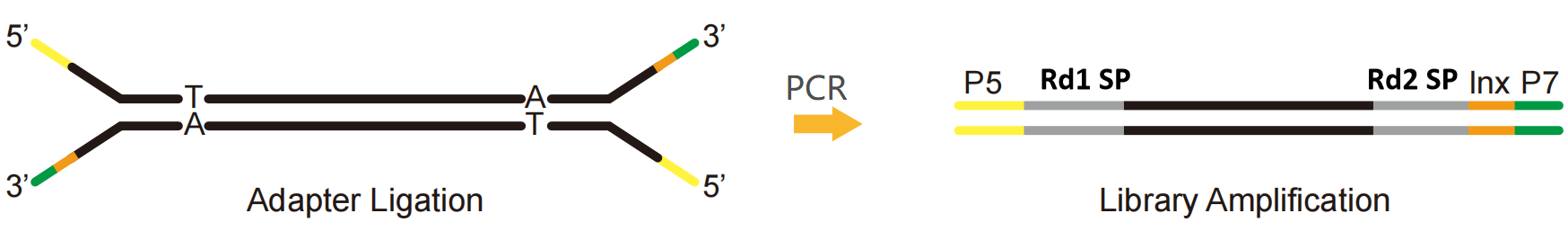
Fikon. 1 Illumina-plattforms enda indexbibliotek

Fikon. 2 MGI-plattformens enda indexbibliotek
Med ökningen av sekvenseringsgenomströmningen kan flera prover sekvenseras samtidigt. Så hur man särskiljer olika prover är särskilt viktigt. Som nämnts tidigare används indexsekvenser av NGS-adapter för att differentiera olika prover i nästa generations sekvensering (NGS). Vilka faktorer bör du tänka på när du väljer index? Fortsätt gärna läsa...
2. Vilka faktorer bör du tänka på när du väljer index?
Indexet är vanligtvis på längden 6nt-18nt och är uppdelat i ett enkelindex och ett dubbelindex enligt antalet index. Det dubbla indexet finns i båda ändarna av fragmentet som ska testas. Basbalans och fluorescensbalans bör beaktas vid val av indexkombination.
Bassaldo avser balansen mellan flera index, snarare än basbalansen i ett enda index. Det måste beaktas från både bastyper och basfördelning. Kombinationsprincipen är att de fyra baserna A/T/C/G i samma grupp av index behöver inkluderas och andelen av dessa fyra baser är nära och står för cirka 25 % respektive.
Fluorescenssignalbalans hänvisar till valet att säkerställa balansen av fluorescerande signaler när basbalansen inte kan garanteras. I 4-kanalssekvenseraren i Illumina-plattformen är dG/dT märkt med grön fluorescens och dC/dA märkt med röd fluorescens. Under sekvensering måste både gröna och röda fluorescenssignaler finnas i varje cykel för att säkerställa framgångsrik sekvensering. Därför bör balansen mellan den gröna signalen och den röda signalen beaktas vid val av index.
3. Vilka är de vanligaste typerna av index?
De vanliga dubbla indexen inkluderar vanligtvis Unique Dual Index (UDI), Unique Dual Barcode (UDB) och Combined Dual Index (CDI), som avsevärt minskar indexhoppning och feltilldelning.
UDI&UDB: indexen i båda ändar är en-till-en-överensstämmande, utformade i grupper och kan korsverifieras i båda ändar;
Ett Stubby UDI Primer Kit för Illumina levererat av Yeasen (Cat#12404ES/12405ES)>>
CDI: indexen i båda ändar kan kombineras enligt vissa krav för att bilda ett dubbelsidigt indexbibliotek;
En 384 CDI Primer för Illumina, Set1-Set2 levererad av Yeasen (Cat#12412ES/12413ES)>>
För att förbättra genomströmnings- och amplifieringseffektiviteten och minska sekvenseringskostnaden introducerade Illumina array flow cell (PFCT) och exklusiv amplifiering (ExAmp) klustringsteknologi för Novaseq och andra high-throughput sekvenserare men förstärkte oavsiktligt fenomenet med provetikettens felmatchning och indexhoppning.

Fig. 3 Illumina olika instrumentmodeller använder icke-mönstrad flödescell eller mönstrad flödescell
För att kompensera för problemet med indexering som lyfts fram av sekvenseringsplattformarna som HiSeq3000/4000, HiSeq X Series och NovaSeq, föreslog Illumina strategin att placera indexet i båda ändarna av biblioteket, vilket kan utföra bilateral verifiering och eliminera de felaktiga adaptrarna. När de unika indexen används i båda ändar kommer indexfelsallokeringsgraden att minskas till 0,01 %. Jämfört med den tidigare konventionella indexpermutationsgruppkombinationsmetoden kommer indexhoppningen att reduceras med två storleksordningar.
I konstruktionen av ett PCR-fritt bibliotek finns en single-end indexadapter tillgänglig. Etikettfelmatchningen orsakas främst av sekvenseringsfel. Totalt sett är etikettens felmatchningsfrekvens låg (genomsnitt 0,0004 %, upp till 0,001 %). I konstruktionen av ett riktat infångningsbibliotek förstärks dock överhörningsproblemet eftersom flera steg kommer att leda till etikettfelmatchning och UDI/UDB/CDI-adaptrarna används vanligtvis.
4.Vilka är de vanligaste typerna av NGS-adaptrar?
Med utvecklingen av sekvenseringstekniken finns det fler och fler typer av adaptrar, såsom enkelindex/dubbelindexadaptrar (som nämnts i avsnitt 3), UMI-adaptrar, transposasadaptrar, kompletta/okompletta adaptrar, etc., som är lämpliga för en mängd olika applikationsscenarier. Denna del sorterar systematiskt ut dessa adaptrar för att ge dig grunden för adapterval.
4.1 UMI-adapter
Unique molecular identifier (UMI) adapter är ett kantverktyg för lågfrekvent mutationsdetektering och absolut kvantifiering. UMI är en slumpmässig syntetisk sekvens med en känd sekvens. Den kan utformas som en helt slumpmässig nukleotidkedja, partiell degenererad nukleotidkedja eller fixerad nukleotidkedja.Längden är vanligtvis 10nt (single-ended UMI) eller 5-8nt (dubbeländad UMI). Dess funktion är att frysa tillståndet av DNA-fragment före amplifiering, och varje DNA-molekyl motsvarar en UMI. Under analysen av bioinformatik kan den därför särskilja DNA-mallar från olika källor, särskilja vilka som är falskt positiva mutationer orsakade av slumpmässiga fel i processen för PCR-amplifiering och sekvensering, och som faktiskt utförs av patienter, för att filtrera bort bakgrundsbruset, inse den exakta detekteringen av lågfrekventa och extremt lågfrekventa mutationer av DNA-mutationer och extremt lågfrekventa DNA-mutationer. Det används i stor utsträckning vid lågfrekvent mutationsdetektion, särskilt inom området för tumörforskning.

Fig. 4 Schematisk bild av UMI-adapter strukturen på Illumina-plattformen
4.2 Kompletta adaptrar
Kompletta adaptrar, en nödvändig produkt för ett PCR-fritt bibliotek, innehåller alla sekvenser som krävs för sekvensering, såsom P5, P7, RdS1 och RdS2 i Illumina-plattformen, även indexsekvenser och UMI-sekvenser enligt kraven för sekvensering. Med de kompletta adaptrarna kan den sekvenseras direkt utan att introducera andra adaptrar via PCR. Så kompletta adaptrar kan användas för att bygga ett PCR-fritt bibliotek. De PCR-fria biblioteken kan minska PCR-amplifieringsbias, felfrekvens och sekvensduplicering, vilket ökar täckningen av vissa regioner med hög GC eller hög AT som används allmänt i forskning om populationsgenom.
En komplett adapterprodukt för Illumina-plattformen levererad av Yeasen (Cat#13519ES/13520ES)>>
En komplett adapterprodukt för MGI-plattform levererad av Yeasen(Cat#13360ES/13361ES)>>
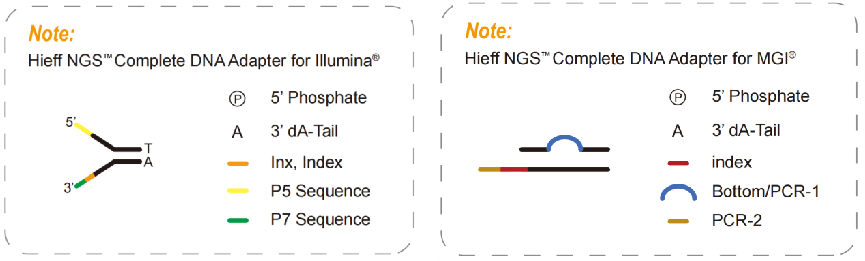
Fig. 5 Komplett adapterdiagram
4.3 Ofullständiga adaptrar
Ofullständiga adaptrar behöver introducera andra sekvenser genom PCR efter adapterligeringen för att bilda en komplett adapter. De kännetecknas av hög anslutningseffektivitet och hög effektiv bibliotekshastighet. PCR-processen är en anrikningseffekt för hela biblioteket för att säkerställa koncentrationen av det effektiva biblioteket, och kan också introducera dubbeländade index och UMI-sekvenser.
4,4 Tn5-adaptrar
Tn5-adaptrar kopplar en del av adaptersekvensen till båda ändarna av DNA-fragmenten genom restriktionsendonukleasaktiviteten hos Tn5. De gör fragmentering och adapterligering som utförs samtidigt för att spara tid och prover. Slutligen introduceras resten av länksekvensen, indexet, UMI och andra sekvenser genom PCR för att bilda ett komplett bibliotek. Det kan användas för att bygga ett Cut&tag-bibliotek.
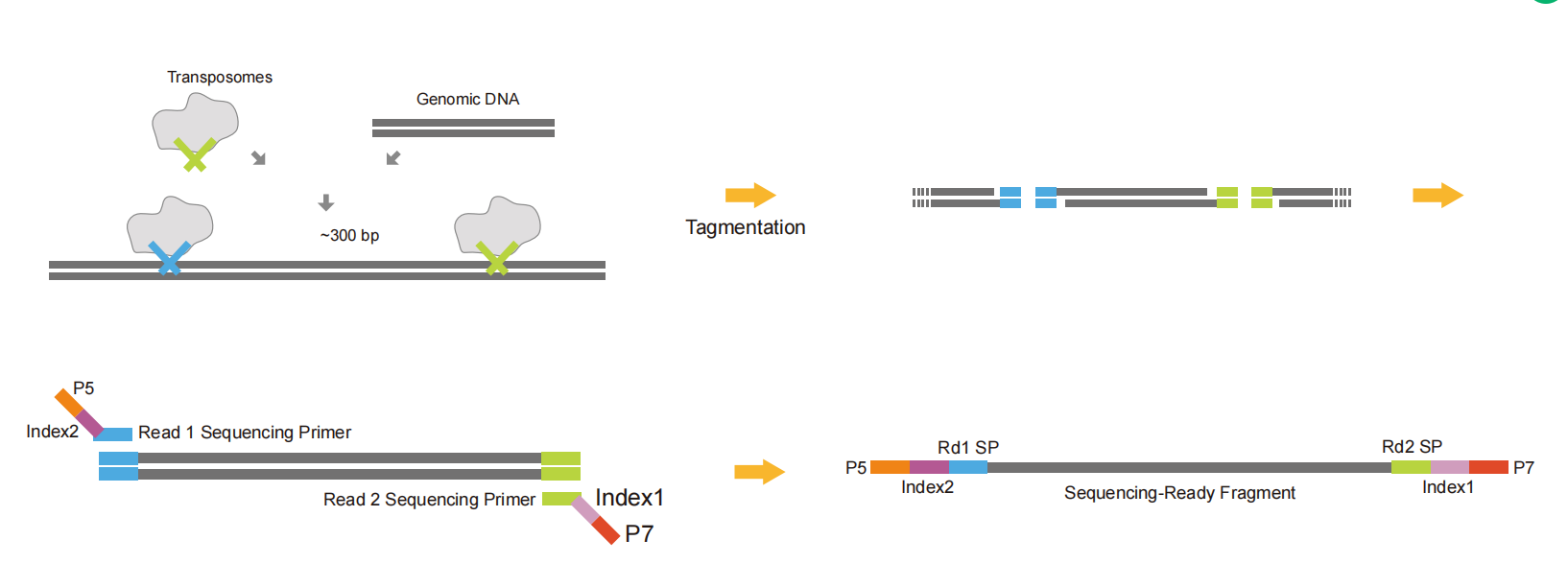
Fig.6 Schematiskt diagram över Tn5-adapterbibliotekets konstruktion
5. Hur väljer man rätt NGS-adaptrar för dina sekvenseringsplattformar?
För närvarande finns det två vanliga sekvenseringsplattformar, inklusive Illumina och MGI. Yeasen, en komplett lösning för NGS-leverantörer, har utvecklat flera NGS-adaptrar lämpliga för Illumina- eller MGI-plattformar.
När det gäller Illumina-plattformen innehåller Illumina NGS-adaptrar som levereras av Yeasen tre typer, inklusive UDI, CDI och ett enda index. När det gäller MGI-plattformen har MGI NGS-adaptrar som erbjuds av Yeasen två typer, som involverar Dual UMI -UDB och Single Index.Vi har listat produktinformationen i följande tabell, inklusive typer av adaptrar, tillgängliga storlekar och koncentration av adapter respektive primer.
Kompletta och UDI NGS-adaptrar behöver inte oroa sig för kopplingsproblem, lämpliga för kunder som vill vara lätta att använda; CDI NGS-adaptrar har färre rör och liten storlek, vilket är lämpligt för kunder som vill förvara och bära enkelt. PCR-fri kräver användning av kompletta NGS-adaptrar.
För Illumina
| InTube | Hieff NGS® DNA Lib Prep 384 CDI Primer för Illumina, set 1 (8*12, 96 index) | 12412ES |
| Hieff NGS® DNA Lib Prep 384 CDI Primer för Illumina, set 2 (8*12, 96 index) | 12413ES | |
| Hieff NGS® RNA Lib Prep 384 CDI Primer för Illumina, set 1 (96 index) | 12414ES | |
| Hieff NGS® RNA Lib Prep 384 CDI Primer för Illumina, set 1 (96 index) | 12415ES | |
| I tallrik | Hieff NGS® Stubby UDI Primer Kit för Illumina (1-384 Index) Set 1-4 | 12407ES |
| Hieff NGS® Stubby UDI Primer Kit för Illumina Set1(96 brunnars platta, 1-96 Index) Set 1 | 12327ES | |
| Hieff NGS® Stubby UDI Primer Kit för Illumina Set 2(96 brunnars platta, 97-192 Index) Set 2 | 12328ES | |
| Hieff NGS® Stubby UDI Primer Kit för Illumina Set 3(96 brunnsplatta, 193-288 Index) Set 3 | 12329ES | |
| Hieff NGS® Stubby UDI Primer Kit för Illumina Set 4(96 brunnars platta, 289-384 Index) Set 4 | 12330ES |
För MGI
| Hieff NGS™ Dubbel UMI UDB-adaptersats för MGI, Set1/Set2 | 96 typer av index | |
| Hieff NGS™ Komplett adaptersats för MGI, Set1/Set2/Set3(Fråga) | 13360ES | 8 sorters index, 41-48 |
| 13361ES | 16 sorters index, 57-72 | |
| 13362ES | 96 sorters index, 1-96 |
Angående läsning
Nyckelenzymer involverade i NGS bibliotekskonstruktion
Om NGS-relaterad teknik, hur mycket vet du?
Olika typer av magnetiska pärlor i NGS: DNA\RNA\mRNA magnetiska pärlor
NGS-bibliotekskvantifiering: Snabb och exakt Qubit eller Exakt qPCR?Allt behövs!
