การเรียนรู้เชิงลึกเกี่ยวกับวัตถุดิบเอนไซม์หลักที่ใช้ใน การตรวจหาเชื้อ SARS-CoV-2 ด้วย PCR
ตั้งแต่เดือนมีนาคม 2022 สายพันธุ์กลายพันธุ์ Omicron ที่เจ้าเล่ห์ได้ทำลายชีวิตที่สงบสุขของผู้คนอีกครั้ง การระบาดของไวรัสโคโรนาสายพันธุ์ใหม่ได้เกิดขึ้นทั่วประเทศและส่งผลกระทบต่อ 30 จังหวัด (เขตปกครองตนเองและเทศบาล) เนื่องจากเป็นวิธีที่มีประสิทธิภาพในการป้องกันและควบคุมการแพร่ระบาดของ SARS-CoV-2 ได้อย่างแม่นยำ การตรวจหากรดนิวคลีอิกจึงกลายเป็นวิถีชีวิตปกติ "คุณทำการตรวจกรดนิวคลีอิกวันนี้หรือไม่" นอกจากนี้ยังกลายเป็นคำทักทายประจำวันของผู้คนอีกด้วย เมื่อพูดถึงเรื่องนี้ คุณรู้หรือไม่ว่าวัตถุดิบหลักใดบ้างที่จำเป็นในการตรวจจับกรดนิวคลีอิก บทความนี้จะแนะนำวัตถุดิบหลักที่สำคัญในเอนไซม์ตรวจจับกรดนิวคลีอิก
1. กระบวนการตรวจจับกรดนิวคลีอิกสำหรับ SARS-CoV-2
2. เอนไซม์หลักในการสกัดกรดนิวคลีอิก
3. เอนไซม์หลักในระหว่างการทำ RT-qPCR
4. เอนไซม์หลักของการตรวจหากรดนิวคลีอิก SARS-CoV-2 จาก
1. กระบวนการตรวจจับกรดนิวคลีอิกสำหรับ SARS-CoV-2
เอนไซม์เป็นไบโอแคตาลิสต์ประเภทหนึ่งที่มีความสำคัญอย่างยิ่ง โดยมีประสิทธิภาพในการเร่งปฏิกิริยาสูงและปฏิกิริยาที่มีความจำเพาะสูง ปฏิกิริยาทางชีวเคมีส่วนใหญ่ต้องอาศัยเอนไซม์ ในกระบวนการตรวจจับ 2019-nCoV ด้วยกรดนิวคลีอิก (แสดงในรูปที่ 1) เอนไซม์โมเลกุลประเภทต่างๆ มีบทบาทสำคัญในขั้นตอนการทดลองต่างๆ เช่น การสกัดกรดนิวคลีอิกและ RT-qPCR ต่อไปนี้ เราจะแยกวัตถุดิบเอนไซม์หลักที่ใช้ในกระบวนการตรวจจับกรดนิวคลีอิกตามความเชื่อมโยงในการทดลองต่างๆ
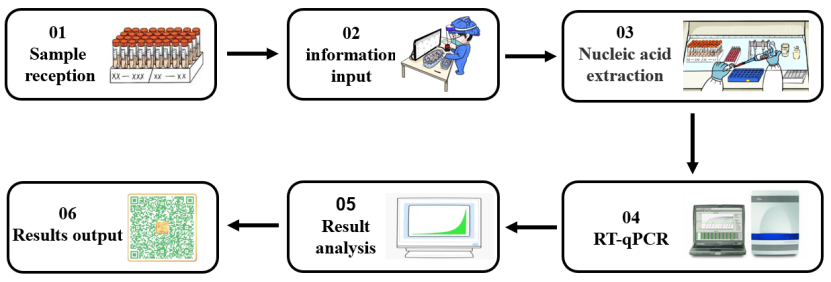
รูปที่ 1 กระบวนการตรวจจับกรดนิวคลีอิกสำหรับ SARS-CoV-2
2. เอนไซม์หลักในการสกัดกรดนิวคลีอิก
กระบวนการสกัดกรดนิวคลีอิกของไวรัสโคโรนาสายพันธุ์ใหม่ประกอบด้วย 2 ขั้นตอนหลัก ได้แก่ การไลซิสและการทำให้บริสุทธิ์ การไลซิสคือกระบวนการทำลายโครงสร้างเซลล์ของตัวอย่างเพื่อให้กรดนิวคลีอิกในตัวอย่างเป็นอิสระในระบบไลซิส การทำให้บริสุทธิ์คือการแยกกรดนิวคลีอิกออกจากส่วนประกอบอื่นๆ ในระบบไลซิสอย่างสมบูรณ์ เช่น โปรตีน เกลือ และสิ่งเจือปนอื่นๆ และกระบวนการปฏิกิริยาต้องอาศัยการมีส่วนร่วมของโปรตีเนส K, ดีออกซีไรโบนิวคลีเอส I และสารยับยั้ง RNase
2.1 โปรตีเอสเค
โปรตีเนสเคเป็นซีรีนโปรตีเอสที่มีกิจกรรมการแตกตัวที่กว้าง โดยไซต์การแตกตัวคือพันธะเปปไทด์ปลายคาร์บอกซีของกรดอะมิโนอะลิฟาติกและอะโรมาติก (รูปที่ 2) ในกระบวนการสกัดกรดนิวคลีอิก โปรตีเนสเคสามารถย่อยสลายฮิสโตนซึ่งจับกับกรดนิวคลีอิกได้อย่างแน่นหนา ส่งเสริมการแยกกรดนิวคลีอิก และทำให้สกัดกรดนิวคลีอิกตัวอย่างได้ง่ายขึ้น นอกจากนี้ โปรตีเนสเคยังสามารถย่อยสลายกิจกรรมของอาร์เอ็นเอไฮโดรเลส (RNase) และยับยั้งการไฮโดรไลซิสของอาร์เอ็นเอเทมเพลตด้วย RNase
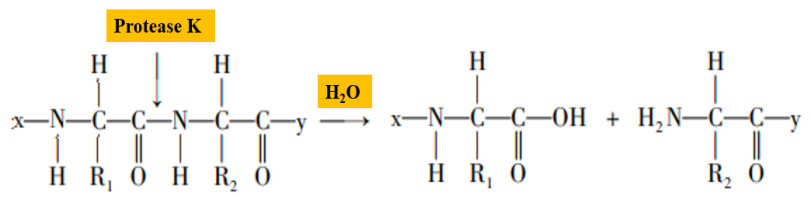
รูปที่ 2 แผนผังของพันธะเปปไทด์ไฮโดรไลซ์ของโปรตีเนส K
2.2 ดีออกซีไรโบนิวคลีเอส I
ดีออกซีไรโบนิวคลีเอส I (DNase I) สามารถเร่งปฏิกิริยาการสร้าง DNA ในรูปแบบต่างๆ มีเป้าหมายเพื่อตัดพันธะฟอสโฟไดเอสเตอร์ที่อยู่ติดกับไพริมิดีน และสร้างโพลีนิวคลีโอไทด์ที่มีกลุ่มฟอสเฟตอยู่ที่ปลาย 5' และกลุ่มไฮดรอกซิลอยู่ที่ปลาย 3' โดยผลิตภัณฑ์จากการย่อยโดยเฉลี่ยคือโพลีเตตระนิวคลีโอไทด์ที่เล็กที่สุดในกระบวนการสกัดกรดนิวคลีอิก SARS-CoV-2 นั้น DNase I จะถูกใช้เพื่อกำจัดการปนเปื้อนจีโนมในตัวอย่าง RNA หลีกเลี่ยงสารตกค้างของ DNA ในเทมเพลต RNA และปรับปรุงความบริสุทธิ์ของเทมเพลต
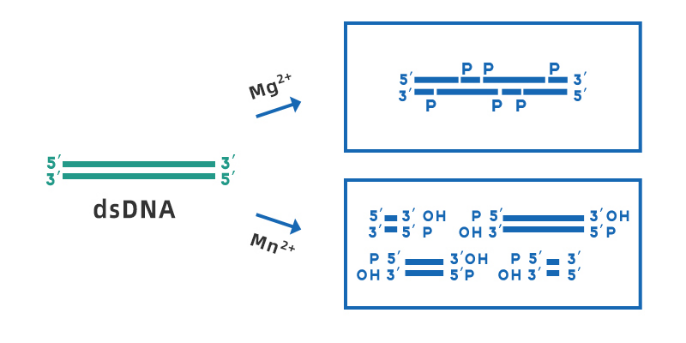
รูปที่ 3 แผนผังของ DNase I ที่ตัด dsDNA ในสภาพที่มี Mg2+ และแมงกานีส2+
2.3 สารยับยั้ง RNase
ในกระบวนการตรวจจับกรดนิวคลีอิกของ SARS-CoV-2 การสกัดและการทำให้บริสุทธิ์ของกรดนิวคลีอิกตัวอย่างหรือการเตรียมระบบปฏิกิริยาทดลองอาจทำให้เกิดการปนเปื้อนของไรโบนิวคลีเอส (RNase) ส่งผลให้เทมเพลต RNA เสื่อมสภาพ เพื่อหลีกเลี่ยงการปนเปื้อนของ RNase จำเป็นต้องใช้สารยับยั้ง RNase
RNase Inhibitor เป็นสารยับยั้ง RNase เฉพาะในรกมนุษย์ ซึ่งสามารถจับกับ RNase โดยเฉพาะเพื่อสร้างสารเชิงซ้อนที่มีพันธะที่ไม่ใช่โควาเลนต์และทำให้ RNase ไม่ทำงาน
3. เอนไซม์หลักในระหว่างการทำ RT-qPCR
หลังจากสกัดกรดนิวคลีอิกจากตัวอย่าง SARS-CoV-2 เสร็จแล้ว การตรวจจับกรดนิวคลีอิกสามารถทำได้โดย RT-qPCR ในกระบวนการทดลองเหล่านี้ DNA โพลิเมอเรส รีเวิร์สทรานสคริปเทส และยูราซิลดีเอ็นเอไกลโคซิเลส ล้วนเป็นวัตถุดิบเอนไซม์หลักที่จำเป็น
3.1 เอนไซม์ทรานสคริปเทสย้อนกลับ
หลังจากการสกัดและการทำให้บริสุทธิ์แล้ว RNA ของ SARS-CoV-2 จะต้องมีเอนไซม์ทรานสคริปเทสย้อนกลับเพื่อเร่งปฏิกิริยาพอลิเมอไรเซชันของ dNTP เพื่อสร้างลำดับ cDNA ที่เสริมกับเทมเพลต RNA (รูปที่ 4) สำหรับปฏิกิริยา RT-qPCR ควรเลือกเอนไซม์ทรานสคริปเทสย้อนกลับที่ทนต่ออุณหภูมิสูง ปัจจุบัน เอนไซม์ทรานสคริปเทสย้อนกลับ MMLV ถูกใช้กันอย่างแพร่หลายที่สุด เนื่องจากไม่มีกิจกรรมเอ็นโดนิวคลีเอสของ DNA และมีกิจกรรม RNase H ต่ำ จึงมีข้อได้เปรียบมากกว่าในการโคลน cDNA
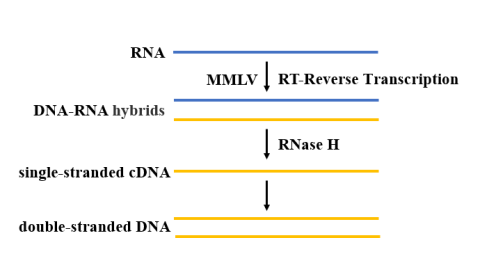
รูปที่ 4 แผนผังกระบวนการถอดรหัสย้อนกลับ
3.2 ดีเอ็นเอโพลีเมอเรส
หลังจากกระบวนการถอดรหัสย้อนกลับของเทมเพลตเสร็จสมบูรณ์เพื่อสร้าง cDNA สายคู่ จะต้องใช้ดีเอ็นเอโพลีเมอเรส "ผู้เล่นวิญญาณ" ในปฏิกิริยา PCR เพื่อปรากฏตัว โดยการโพลีเมอไรเซชันดีออกซีไรโบนิวคลีโอไทด์อิสระเพื่อขยายสายดีเอ็นเอ และดีเอ็นเอเทมเพลตจำนวนมากจะถูกขยายในหลอดทดลองเพื่อให้บรรลุวัตถุประสงค์ในการตรวจจับกรดนิวคลีอิกของไวรัส
DNA โพลิเมอเรสที่ใช้กันทั่วไปในปฏิกิริยา RT-qPCR คือ Taq DNA โพลิเมอเรสแบบฮอทสตาร์ต เอนไซม์ประเภทนี้จะไม่ทำงานที่อุณหภูมิห้องมีกิจกรรมพอลิเมอไรเซชันเฉพาะหลังจากการสตาร์ทร้อนเท่านั้น ซึ่งสามารถลดการสร้างสัญญาณพื้นหลังได้ ช่วยแก้ปัญหาการขยายแบบไม่จำเพาะที่เกิดจากการสร้างไพรเมอร์-ไดเมอร์หรือการไม่ตรงกันในปฏิกิริยา PCR ทั่วไป ปัจจุบัน วิธีปรับเปลี่ยนการสตาร์ทร้อนของดีเอ็นเอโพลีเมอเรสที่ใช้กันทั่วไป ได้แก่ การดัดแปลงทางเคมี การดัดแปลงลิแกนด์ และการดัดแปลงแอนติบอดี หลักการของวิธีการปรับเปลี่ยนการสตาร์ทร้อนที่แตกต่างกันแสดงไว้ในรูปที่ 5
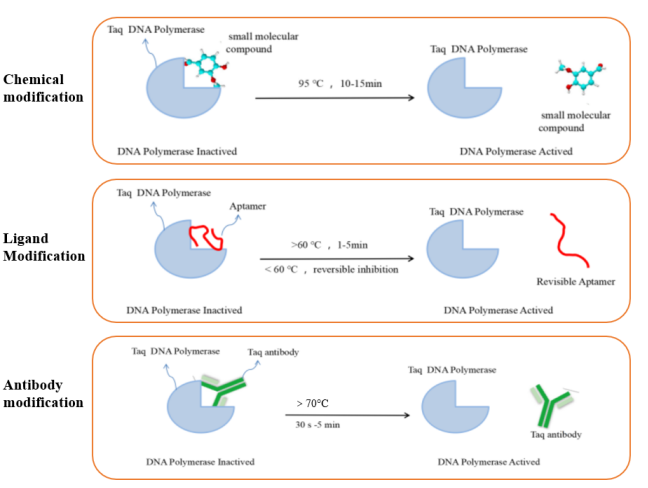
รูปที่ 5 แผนผังของเอนไซม์ฮอทสตาร์ทดัดแปลงชนิดต่างๆ
3.3 ยูราซิลดีเอ็นเอไกลโคซิเลส
ในกระบวนการตรวจจับกรดนิวคลีอิกของไวรัสโคโรนาสายพันธุ์ใหม่ มลพิษจากละอองลอยในสภาพแวดล้อมการทำงานเป็นปัจจัยที่พบบ่อยที่สุดที่ทำให้เกิดผลบวกปลอมของ PCR การเติมเอนไซม์ UDG (Uracil DNA Glycosylase, uracil DNA glycosylase) ลงในระบบขยายพันธุ์สามารถกำจัดมลพิษตกค้างจากการขยายพันธุ์ (ส่วนใหญ่อยู่ในรูปของละอองลอย) ที่ผสมอยู่ในระบบ PCR ได้อย่างมีประสิทธิภาพเพื่อให้แน่ใจถึงความแม่นยำของผลการขยายพันธุ์ หลักการต่อต้านมลพิษของเอนไซม์ UDG แสดงไว้ในรูปที่ 6
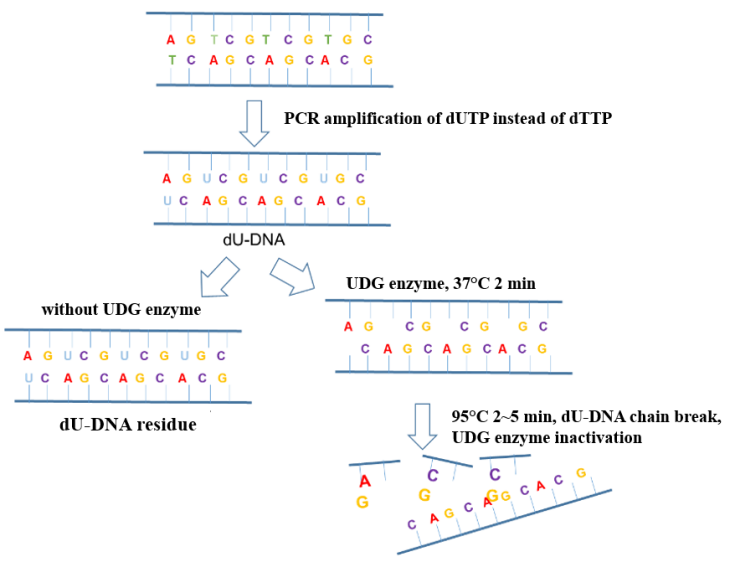
รูปที่ 6 แผนผังหลักการต่อต้านมลพิษของเอนไซม์ UDG
4.เอนไซม์หลักของการตรวจจับกรดนิวคลีอิกของ SARS-CoV-2 จาก Yeasen
| กระบวนการ | คำอธิบาย | ชื่อสินค้า | รหัสสินค้า |
| การประมวลผลตัวอย่าง | การย่อยโปรตีน | 10401ES | |
| การสกัด RNA | รีคอมบิแนนท์ ดีเอ็นเอส ไอ (ปลอด RNase) (สอบถาม) | 10325ES | |
| การยับยั้ง RNase | สารยับยั้ง RNase ของหนู (40U/ ไมโครลิตร) | 10603ES | |
| การถอดความย้อนกลับ | เหมาะสำหรับ RT-qPCR | ไฮแฟร์™ วี รีเวิร์สทรานสคริปเทส (200U/ ไมโครลิตร) | 11300ES |
| HifairTM V Reverse Transcriptase (600U/ μL) ปราศจาก GlyceroL (สอบถาม) | 11301ES | ||
| การขยาย PCR | ดีเอ็นเอโพลีเมอเรสแบบฮอตสตาร์ท | 10726ES | |
| UDG ความร้อน | ยูราซิลดีเอ็นเอไกลโคซิเลส (UDG/UNG) ไม่เสถียรต่อความร้อน 1 U/μL | 10303ES |
ในส่วนของการอ่าน:
การคัดเลือกทรานสคริปเทสย้อนกลับ
สารยับยั้ง RNase ในหนู - กำจัดการปนเปื้อนของ RNase และรักษา RNA ได้สำเร็จ
