Restriktionsendonukleaser fra Yeasen FuniCut™-serien, 5 minutters fordøjelse med universalbuffer
Molekylær kloning bruges i næsten alle laboratorier. Restriktionsendonukleaser er en afgørende komponent i molekylære kloningseksperimenter, men folk er også ofte generet af forskellige problemer med restriktionsendonuklease-eksperimenter. Almindelige problemer er som følger:
- Der er så mange typer restriktionsendonukleaser, at det er svært at vælge.
- Fejlspaltning, tilfældig spaltning eller ufuldstændig enzymatisk spaltning.
- Fordøjelseshastigheden er træg; det kan tage en time eller endda natten over.
- Derudover skal flere enzymfordøjelseseksperimenter også vælge en række forskellige enzymfordøjelseseksperimenter bTo
For med ide at bevise en mere grundig forståelse af restriktionsendonukleaser, vil det følgende kort forklare, hvad restriktionsendonukleaser er, og hvordan forskellige endonukleaser klassificeres som svar på det første spørgsmål. Yeasen FuniCut™-serien af endonukleaser med hurtig restriktion kan let løse det andet til det fjerde problem gennem belastningsmodulering og en forbedret procedure!
1. Hvad er restriktionsendonuklease?
2. Restriktioner endonukleasenomenklatur
3. Restriktioner for endonukleaseklassificering
4. Yeasen FuniCut™ hurtige restriktionsendonukleaser
1. Hvad er restriktionsendonuklease?
Restriktionsendonukleaser er en klasse af enzymer, der kan genkende specifikke nukleotidsekvenser i dobbeltstrengede DNA-molekyler og skære phosphodiester-binding i DNA-kæder på specifikke steder.
Forskellige restriktionsendonukleaser vil genkende forskellige DNA-sekvenser, De kan skære DNA inde i genkendelsessekvensen eller et sted ikke langt fra genkendelsessekvensen, hvilket resulterer i forskellige produkter, som illustreret i figur 1. BamHI danner klæbrige ender, KpnI danner 3'ticky ender, og EcoRV danner stumpe ender.
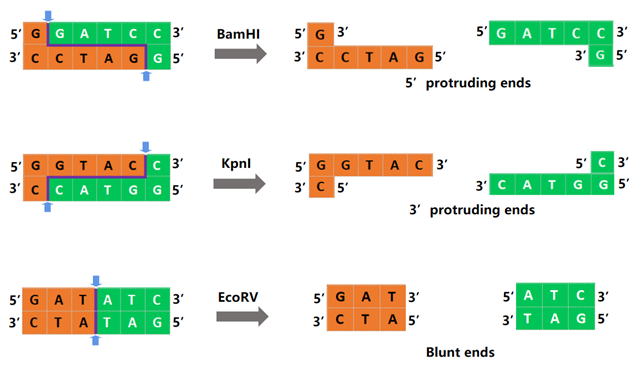
Figur 1. Skematisk diagram af BamHI, KpnI, EcoRV fordøjelse
2. Restriktioner endonukleasenomenklatur
Slægtsnavnet, artsnavnet, bakteriestammen eller serotypen og rækkefølgen af opdagelse er de primære faktorer, der bruges til at navngive restriktionsendonukleaser. Navneretningslinjerne er vist i tabel 1 nedenfor med BstEII som eksempel:
Tabel 1. Begrænsninger endonukleasenomenklatur
| Forkortelse | Fuldt navn | Implikation |
| B | Bacillus | Første bogstav i slægtsnavnet |
| st | stearothermophilus | De to første bogstaver i artsnavnet |
| E | ET | Det første bogstav i stammenavnet |
| II | Anden opdagelse | Den rækkefølge, der findes i sådanne bakterier |
3.Begrænsninger for endonukleaseklassificering
I henhold til kompleksiteten af strukturen, virkemåden og forskellen mellem cofaktorerne kan restriktionsenzymer opdeles i fire kategorier. Den følgende tabel 2 opsummerer egenskaberne og typiske enzymer for hver kategori.
Tabel 2. Variationerne mellem flere restriktionsendonukleaser
| Endonuklease klasse | Funktioner | Typiske enzymarter |
| Type I | 1. Genkendelse og modifikation spaltning. 2. Det kan genkende specifikke DNA-sekvenser, og fordøjelsesstedet er på ubestemt tid væk fra genkendelsesstedet, op til tusindvis af baser. 3. Handling nødvendiggør ATP. | EcoB, EcoK osv. |
| Type II | 1. Har kun funktionen at identificere skæring. 2. Genkendelsessekvensen er ofte en kort palindromisk sekvens (ca. 4-8 bp), og det specifikke fordøjelsessted for restriktionsendonukleasen er typisk den genkendte sekvens. 3. Handling nødvendiggør Mg2+. 4. Den type restriktionsenzymer, der oftest anvendes i molekylær kloning. | HindIII, NotI osv. |
| Type III | 1. Genkendelse og modifikation fordøjelse. 2. Separer om at adskille genkendelsesstedet fra fordøjelsesstedet. 3. Handling nødvendiggør ATP. | HinfIII et al. |
| Type IV | 1. Skærer kun methylerede DNA-sekvenser. 2. Ca. 30 bp adskiller genkendelsesstedet fra fordøjelsesstedet. | McrA, McrBC osv. |
Som angivet i tabel 3 nedenfor kan den også opdeles i tre grupper baseret på lighederne og forskellene mellem genkendelses- og fordøjelsesstederne.
Tabel 3.Sammenlignende analyse af Isoschizomer, Neoschizomer og Isocaudarner
|
| Isoskizomer | Neoskizomer | Isocaudarner |
| genkendelsessekvens | samme | samme | anderledes |
| fordøjelsessteder | samme | anderledes | samme |
| typisk repræsentant | AgeI og BshTI: både genkender og spalter 5′-A↓CCGGT-3′ | SmaI (5'-CCC↓GGG-3′) og XmaI(5'-C↓CCGGG-3′) | BamHI (5'-G↓GATCC-3') og BglII (5'-A↓GATCT-3') |
I lyset af det faktum, at nogle restriktionsendonukleaser på markedet stadig kræver fordøjelse natten over, og produkterne er tilbøjelige til ukorrekt fordøjelse, fuldfører Yeasen Fast Restriction Endoa nuklease, en universal buffer, nøjagtig fordøjelse på 5 minutter, hvilket tillader dine fordøjelseseksperimenter ikke flere problemer!
4. Yeasen FuniCut™ Hurtige restriktionsendonukleaser
Figur 2. Yeasen FuniCut™ endonukleaser med hurtig restriktion
4.1 Funktioner
- Hurtig fordøjelse: fordøjelsen kan afsluttes på 5-15 minutter, hvilket kan reduceres med mere end 1-2 timer sammenlignet med konventionelle fordøjelseseksperimenter.
- Universal buffer: Multi-enzym spaltningsreaktionen gøres enklere ved, at enhver kombination af endonukleaser kan bruge den samme buffer.
- Effekt af enzymfordøjelse: sammenlignelig med N* og passende for dets buffer.
- Direkte elektroforese gør processen enklere ved at give en rød farvebuffer og tillade, at fordøjelsesprodukterne leveres direkte til elektroforese.
- Præcis fordøjelse: Selv med fordøjelse natten over er der meget lidt stjerneaktivitet.
4.2 Udstilling af forestilling
4.2.1 I FuniCut™ Buffer finder enkelt, dobbelt og tredobbelt fordøjelse sted på 5 minutter.

Figur 3. Enkelt-, dobbelt- og tredobbelt fordøjelse ved hjælp af FuniCut™ Buffer kan typisk være færdig på 5 minutter, for eksempel den tredobbelte fordøjelse ved hjælp af EcoRI+KpnI+SmaI.
4.2.2 Virkningen af enzymfordøjelsen er sammenlignelig med virkningen af N* og T*, og den fungerer godt med deres buffere.

Figur 4. Sammenlignet med lignende N*, T* varer er Yeasen FuniCut™ BamHI enzymfordøjelseseffekt kompatibel med N*, T* mærkebuffere.
4.2.3 Fordøjelse natten over med meget lav stjerneaktivitet
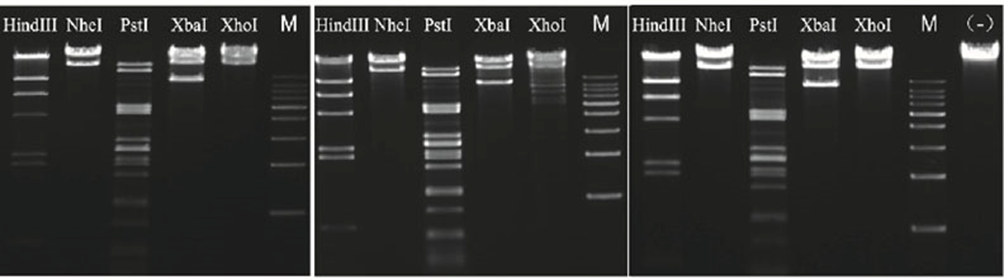
Figur 5. Brug de hurtige endonukleaser HindIII, NheI, PstI, Xbal og XhoI fra Yeasen, Tbydance med den eksperimentelle protokol, der anbefales af hvert mærke.Udfør en fordøjelse natten over (16 timer) og analyser de fordøjede produkter ved hjælp af agarosegelelektroforese.
4.2.4 Meget overflødig og i stand til at håndtere noget overskydende substrat.
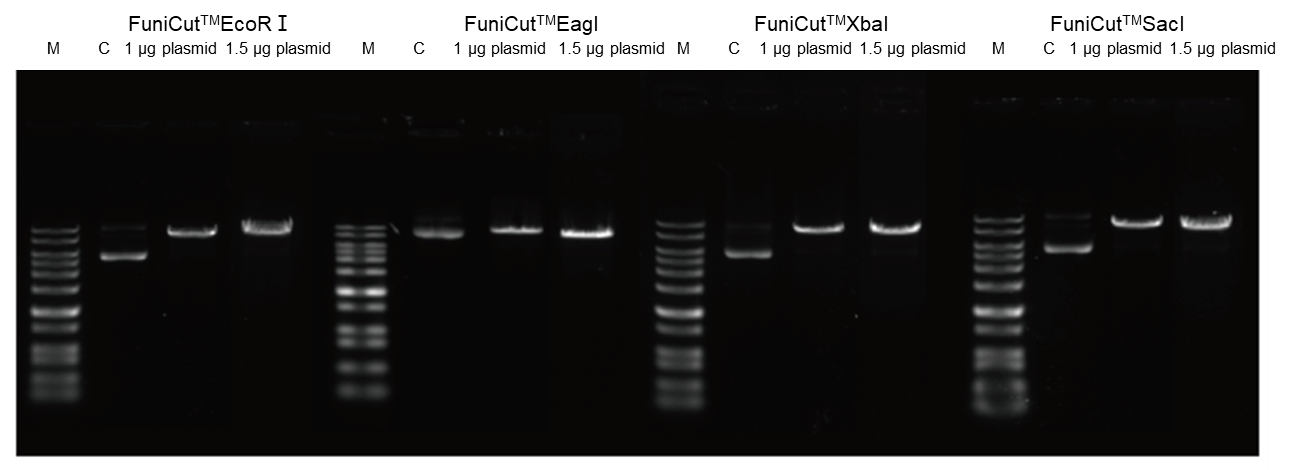
Figur 6. Ved brug af FuniCut™ Fast Restriction Endonucleases EcoR I, Eag I, XbaI og Sac I blev henholdsvis 1 μg og 1,5 μg plasmid brugt som substrater og fordøjet i 5 minutter, og de fordøjede produkter blev udsat for agarosegelelektroforese.
Tabel 4.Produkter og reaktionsbufferkompatibilitet
| Produktnavn | Kat# | Genkendelsessekvens | Temperatur | Termisk deaktiveringstemperatur |
| T* buffer | N* buffer | Ta* Buffer | Beskyttet base (bp) | Svar |
| FuniCut™ AscI (spørge) | 15001ES50 | GG/CGCGCC | 37 | 80 | 100 | 100 | 100 | 100 | 1 | 50T |
| FuniCut™ AvrII (spørge) | 15002ES25 | C/CTAGG | 37 | 80 | 100 | 100 | 100 | 100 | 3 | 25T |
| FuniCut™ BamHI (spørge) | 15003ES76 | G/GATCC | 37 | 80 | 100 | 100 | 100 | 100 | 3 | 500T |
| FuniCut™ BclI (spørge) | 15004ES62 | T/GATCA | 37 | 80 | 100 | 100 | 100 | 100 | 3 | 125T |
| FuniCut™ BsaI (spørge) | 15005ES50 | GGTCTC(1/5) | 37 | 80 | 100 | 100 | 100 | 100 | Ikke | 50T |
| FuniCut™ BstEII (spørge) | 15006ES60 | G/GTNACC | 37 | 80 | 100 | 75 | 100 | 75 | Ikke | 100T |
| FuniCut™ ClaI (spørge) | 15007ES50 | AT/CGAT | 37 | 80 | 100 | 100 | 100 | 100 | 4 | 50T |
| FuniCut™ DpnII (spørge) | 15008ES50 | /GATC | 37 | 80 | 100 | 75 | 100 | 75 | Ikke | 50T |
| FuniCut™ EagI (spørge) | 15009ES25 | C/GGCCG | 37 | 80 | 100 | 100 | 100 | 100 | 2 | 25T |
| FuniCut™ EcoRI (spørge) | 15010ES78 | G/AATTC | 37 | 80 | 100 | 100 | 100 | 100 | 5 | 600T |
| FuniCut™ EcoRV (spørge) | 15011ES70 | GAT/ATC | 37 | 80 | 100 | 100 | 100 | 100 | 5 | 200T |
| FuniCut™ HindIII (spørge) | 15012ES76 | A/AGCTT | 37 | 80 | 100 | 75 | 100 | 100 | 3 | 500T |
| FuniCut™ HpaI (spørge) | 15013ES50 | GTT/AAC | 37 | 80 | 100 | 100 | 100 | 50 | 1 | 50T |
| FuniCut™ KpnI (spørge) | 15015ES70 | GGTAC/C | 37 | 80 | 100 | 100 | 100 | 100 | 2 | 200T |
| FuniCut™ MluI (spørge) | 15016ES60 | A/CGCGT | 37 | 80 | 100 | 75 | 100 | 100 | 3 | 100T |
| FuniCut™ NcoI (spørge) | 15018ES30 | C/CATGG | 37 | 80 | 100 | 100 | 100 | 100 | 3 | 30T |
| FuniCut™ NdeI (spørge) | 15019ES70 | CA/TATG | 37 | 80 | 100 | 100 | 100 | 100 | 3 | 200T |
| FuniCut™ NheI (spørge) | 15020ES30 | G/CTAGC | 37 | 80 | 100 | 100 | 100 | 100 | 3 | 30T |
| FuniCut™ NotI (spørge) | 15021ES50 | GC/GGCCGC | 37 | 80 | 100 | 100 | 100 | 100 | 2 | 50T |
| FuniCut™ PstI (spørge) | 15022ES76 | CTGCA/G | 37 | Ingen | 100 | 100 | 100 | 100 | 2 | 500T |
| FuniCut™ SacI (spørge) | 15023ES60 | GAGCT/C | 37 | 80 | 100 | 100 | 100 | 100 | 3 | 100T |
| FuniCut™ SalI (spørge) | 15024ES70 | G/TCGAC | 37 | 80 | 100 | 100 | 100 | 100 | 3 | 200T |
| FuniCut™ SbfI (spørge) | 15025ES25 | CCTGCA/GG | 37 | 80 | 100 | 100 | 100 | 100 | 2 | 25T |
| FuniCut™ SmaI (spørge) | 15027ES60 | CCC/GGG | 37 | 80 | 100 | 100 | 100 | 100 | 1 | 100T |
| FuniCut™ Spil (spørge) | 15028ES50 | A/CTAGT | 37 | 80 | 100 | 100 | 100 | 100 | 2 | 50T |
| FuniCut™ SphI (spørge) | 15029ES50 | GCATG/C | 37 | 80 | 100 | 100 | 100 | 100 | 1 | 50T |
| FuniCut™ SspI (spørge) | 15030ES56 | AAT/ATT | 37 | 80 | 100 | 50 | 100 | 100 | 2 | 60T |
| FuniCut™ StuI (spørge) | 15031ES60 | AGG/CCT | 37 | 80 | 100 | 100 | 100 | 100 | 1 | 100T |
| FuniCut™ TaqI (spørge) | 15032ES70 | T/CGA | 65 | Ingen | 100 | 100 | 100 | 100 | 4 | 200T |
| FuniCut™ XbaI (spørge) | 15033ES76 | T/CTAGA | 37 | 80 | 100 | 50 | 100 | 100 | 2 | 500T |
| FuniCut™ XhoI (spørge) | 15034ES76 | C/TCGAG | 37 | 80 | 100 | 100 | 100 | 100 | 4 | 500T |
| FuniCut™ FspI (spørge) | 15036ES50 | TGC/GCA | 37 | 80 | 100 | 100 | 100 | 100 | 3 | 50T |
| FuniCut™ HinfI (spørge) | 15038ES76 | G/ANTC | 37 | 80 | 100 | 100 | 100 | 50 | 2 | 500T |
| FuniCut™ ApaLI (spørge) | 15039ES70 | G/TGCAC | 37 | 80 | 100 | 75 | 100 | 100 | 4 | 200T |
| FuniCut™ NruI (spørge) | 15040ES50 | TCG/CGA | 37 | Ingen | 100 | 100 | 100 | 100 | 5 | 50T |
| FuniCut™ PacI (spørge) | 15041ES25 | TTAAT/TAA | 37 | 80 | 100 | 100 | 100 | 100 | 2 | 25T |
| FuniCut™ PvuII (spørge) | 15042ES70 | CAG/CTG | 37 | Ingen | 100 | 100 | 100 | 100 | 4 | 200T |
| FuniCut™ SacII (spørge) | 15043ES50 | CCGC/GG | 37 | 80 | 100 | 100 | 100 | 100 | 4 | 50T |
| FuniCut™ NsiI (spørge) | 15044ES25 | ATGCA/T | 37 | 80 | 100 | 100 | 100 | 100 | 3 | 25T |
| FuniCut™ Esp3I (BsmBI) (spørge) | 15048ES30 | CGTCTC(1/5) | 37 | 80 | 100 | 100 | 100 | 100 | 5 | 30T |
| FuniCut™ BglII (spørge) | 15049ES60 | A/GATCT | 37 | 80 | 100 | 100 | 100 | 50 | 2 | 100T |
| FuniCut™ BstBI (spørge) | 15050ES60 | TT/CGAA | 37 | 80 | 100 | 100 | 100 | 100 | Ikke | 100T |
| FuniCut™ MnlI (spørge) | 15051ES50 | CCTC(7/6) | 37 | 80 | 100 | 100 | 100 | 100 | 2 | 50T |
CpG: Påvirket af CpG-methylering.
EK: Påvirket af EcoKI-methylering.
EB: Påvirket af EcoBI-methylering.
Dcm: Påvirket af Dcm-methylering.
Dam: Påvirket af Dam-methylering.

