Hvad skal man gøre, når qPCR-resultaterne er mærkelige?
qPCR-eksperimenter kan føles nemme og nogle gange svære. Efter at primerdesignet er afsluttet, tilføjes qPCR-prøver trin for trin som PCR, og eksperimentet kan fuldføres på maskinen. Der er flere almindelige problemer i qPCR-eksperimenter efter farvestofmetode, og givet de mulige årsager og løsninger, håber vi, at du kan bruge dem til at løse problemerne i qPCR-eksperimenter.
1. Hvad er qPCR?
2. Unormal qPCR-amplifikationskurve
3. Unormal qPCR-smeltekurve
4. Bestillingsoplysninger
5. Relaterede produkter
6. Vedrørende læsning
1. Hvad er qPCR?
Siden polymerasekædereaktionsteknikken (PCR) blev opfundet, er PCR sandsynligvis den mest udbredte teknik i molekylærbiologi på grund af dens enkelhed, billighed, pålidelighed, hurtighed og høje følsomhed. qPCR er en teknologi udviklet af PCR-teknologi. Under DNA-amplifikationsprocessen har metoden til påvisning af den samlede mængde af produkter efter hver PCR-cyklus med fluorescerende farvestoffer ikke kun PCR's hurtighed og følsomhed, men har også mere specificitet og høj, realtidsovervågning, repeterbar og nøjagtig kvantificering og andre fordele. qPCR er en teknologi, der bruger en kvantitativ realtids-PCR-amplifikationsenhed, nemlig et qPCR-instrument, til at overvåge nukleinsyreamplifikationsprodukter i realtid under PCR-processen. qPCR realiserer den kvantitative analyse af startskabelonen gennem analysen af Ct-værdien og standardkurven. I 1992 foreslog japanske Higuchi først "real-time fluorescerende kvantitativ PCR-teknologi". I 1996 lancerede American Biological Company verdens første fluorescerende kvantitative PCR-instrument, som er sammensat af et PCR-amplifikations termisk cyklussystem, optisk fluorescensdetektionssystem og computer- og applikationssoftware. Det kan overvåge nukleinsyreamplifikationsprodukter i realtid gennem fluorescerende farvestoffer eller fluorescerende prober. , gennem det matematiske funktionsforhold, kombineret med softwaren til resultatanalyse, realiseres beregningen af den oprindelige skabelonmængde af prøven, der skal testes. Derfor er real-time fluorescerende kvantitativ PCR-teknologi blevet brugt i vid udstrækning. Fluorescerende mærkningsmetoder til qPCR er opdelt i fluorescerende farvestofmosaikmetode baseret på SYBR Green I-farvemetode, fluorescerende probemetode baseret på Taqman-probemetode (Cycling Probe, Molecular Bracon, etc.), quencher-farvestofprimermetode.
qPCR-eksperimenter kan føles nemme og nogle gange svære. Efter at primerdesignet er afsluttet, tilføjes qPCR-prøver trin for trin som PCR, og eksperimentet kan fuldføres på maskinen. Mange små detaljer kræver dog opmærksomhed, såsom ingen væske hængende på rørvæggen, ingen bobler i røret og behovet for at minimere den direkte kontakt mellem rørvæggen, rørdækslet osv. for at reducere eksperimentelle fejl eller mærkelige forsøgsresultater. Xiaoyi har samlet flere almindelige problemer i qPCR-eksperimenter ved farvestofmetode og givet de mulige årsager og løsninger, som vi håber du kan bruge til at løse problemerne i qPCR-eksperimenter.
2. Unormal qPCR-amplifikationskurve
2.1 Abnormiteter i basislinjefasen af det logaritmiske plot af amplifikationskurven
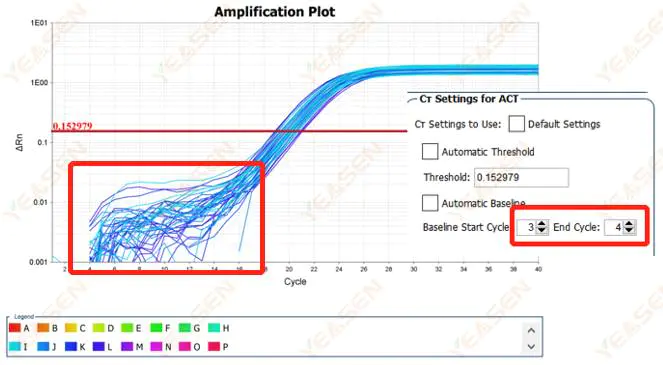
Figur 1. Amplifikationsplot
Mulige årsager:
Forkert grundlinjeindstilling.
Løsninger:
Det anbefales at øge endepunktsværdien for basislinjen.Justeringseksempeldiagrammet er som følger.

Figur 2. Amplifikationsplot
2.2 Amplifikationskurve log plot kurve segmentering
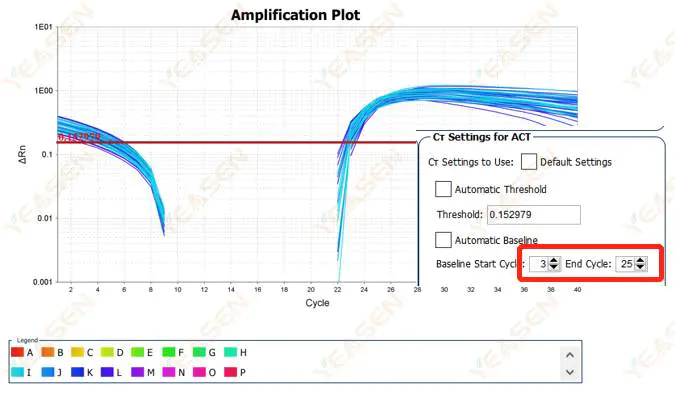
Figur 3. Amplifikationsplot
Mulige årsager:
Grundlinjen er sat for højt.
Løsninger:
Det anbefales at reducere endepunktsværdien for basislinjen. Justeringseksempeldiagrammet er som følger.
2.3 Ujævn amplifikationskurve linearitetsplot
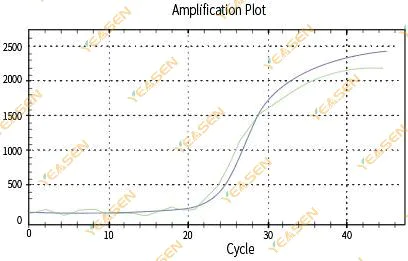
Figur 4. Amplifikationsplot
Mulige årsager:
1) PCR-reaktionsrøret var ikke lukket tæt, og reaktionsopløsningen lækkede.
2)PCR-reaktionsopløsning med hængende væg.
3) Instrumentet er ikke kalibreret (inklusive automatisk kalibrering eller ROX-kalibrering).
4) Systemet har mange inhibitorer, hvilket fører til ustabil fluorescens.
5) Overdreven brug af instrumentet resulterer i ustabil fluorescensopsamling.
Løsninger:
1) Tryk rørdækslet hårdt.
2) Bland reagenset grundigt, og sæt det forsigtigt i det kvantitative instrument efter fuld centrifugering.
3) Kalibrer instrumentet.
4) Forbedre RNA-renheden og vælg passende revers transkriptionsreagenser.
2.4 Forstærket kurve lineær graf zigzag på et plateau
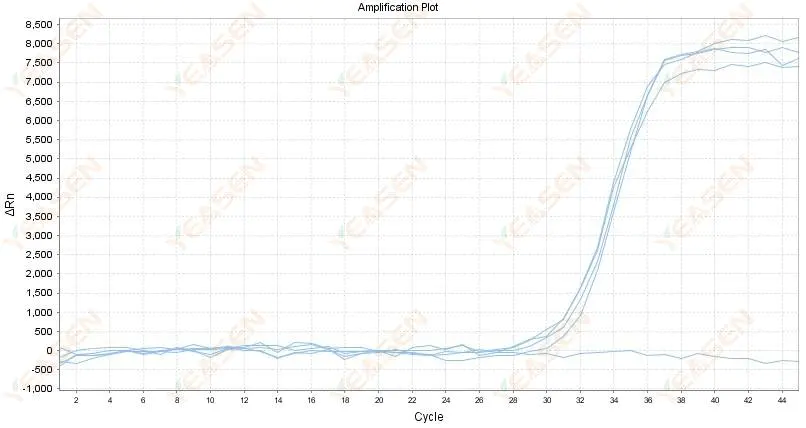
Figur 5. Amplifikationsplot
Mulige årsager:
1) Renheden af RNA er dårlig, og der er mange urenheder.
2) Instrumentet er brugt for længe.
Løsninger:
1) Genudtræk RNA af høj kvalitet.
2) Fortynd RNA-skabelonen for at reducere koncentrationen af urenheder.
3) Kalibrer instrumentet.
2.5 Lineær plot af amplifikationskurven når ikke et plateau
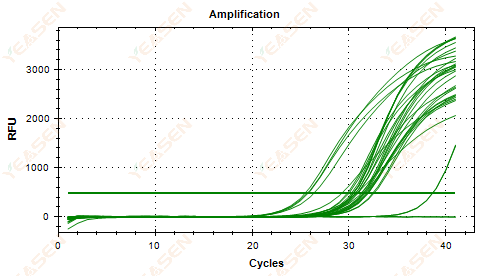
Figur 6. Amplifikationsplot
Mulige årsager:
1) Lav skabelonkoncentration (Ct-værdi omkring 35).
2) For få forstærkningscyklusser.
3) Lav effektivitet af reagensforstærkning (lille Ct, men også ude af stand til at nå plateau).
Løsninger:
1) Øg koncentrationen af skabelonen.
2) Øg antallet af cyklusser.
3) Stigende Mg2+ koncentration.
2.6 Amplifikationskurve lineært plot plateau faldende
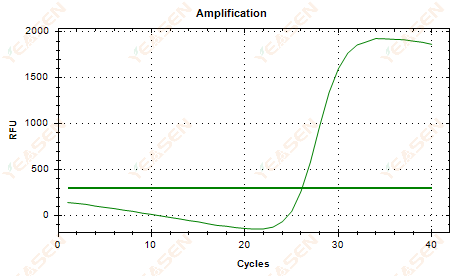
Figur 7. Amplifikationsplot
Mulige årsager:
1) Tilstedeværelse af nedbrydning (forstærkningsproduktnedbrydning, SYBR-nedbrydning).
2) Rørets hætte er ikke dækket korrekt, og reagenset er fordampet.
3) cDNA-koncentrationen er for høj (hvis Ct-værdien er for lille, trækkes fluorescens-tærsklen højt, og sagning er mere alvorlig).
4) Luftbobler i røret, så forsvinder de.
Løsninger:
1) Forbedre systemets renhed.
2) Reducer cDNA-mængden (fortyndet skabelon).
3) Reducer slutpunktværdien for basislinjen.
2.7 Stor værdi af Ct i amplifikationskurven
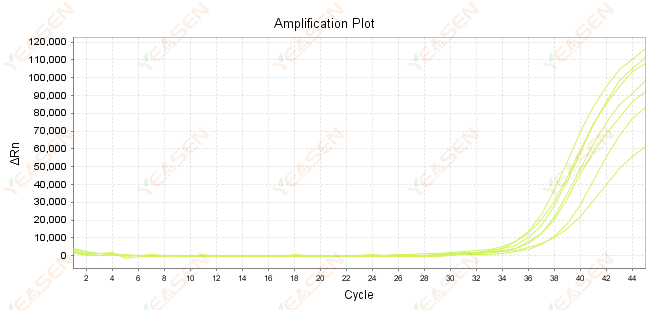
Figur 8. Amplifikationsplot
Mulige årsager:
1) Lavt antal skabeloner.
2) Lav forstærkningseffektivitet.
3) PCR-fragmentet er for langt.
4) Tilstedeværelse af inhibitorer i reaktionssystemet.
Løsninger:
1) Reducer fortyndingstiderne eller øg mængden af skabelon, så Ct-værdien falder mellem 15 og 30 så meget som muligt.
2) Optimer reaktionsbetingelserne, prøv en tre-trins amplifikationsprocedure, eller redesign primere.
3) PCR-produktlængden er designet til at være inden for 100 -150 bp, anbefales ikke at overstige 300 bp.
4) Reproduktion af skabeloner med højere renhed.
2.8 Dårlig repeterbarhed af amplifikationskurver
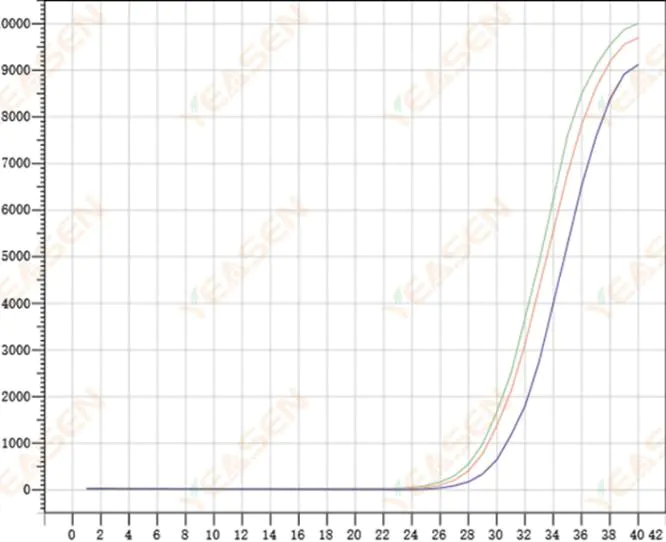
Figur 9. Amplifikationsplot
Mulige årsager:
1) Store spidsfejl.
2) Reagenser og systemer er ikke godt blandet.
3) Lavt kopiantal af cDNA.
4) Der blev ikke brugt nogen Rox-kalibrering.
Løsninger:
1) Kalibrering af pipetter.
2) Grundig blanding af reaktionssystemet.
3) Lav skabelonkoncentration, dårlig repeterbarhed, 4-6 sammensatte huller og 1-2 afvigelser af større værdier kan kasseres passende.
4) Det er bedst at bruge Rox-kalibrering. Hvis det anvendte reagens ikke indeholder Rox, skal referencefarvestoffet vælges som Ingen.
2.9 Tilfældige amplifikationskurver
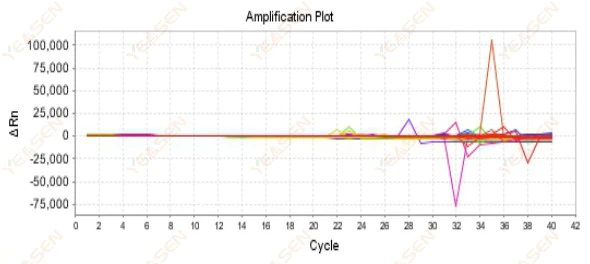
Figur 10. Amplifikationsplot
Mulige årsager:
Misforholdet mellem Rox-koncentration og modellen.
Løsninger:
Skift indstillingen for referencefarvestof på instrumentet fra ROX til INGEN, og se om amplifikationskurven vender tilbage til normal.
2.10 NTC Peaking
2.10.1 Ct>35, smeltekurve Tm-værdi <80℃
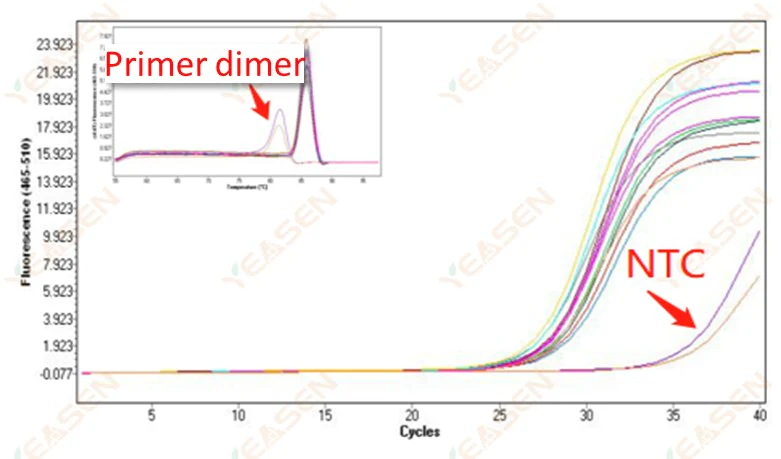
Figur 11. Amplifikationsplot
Mulige årsager:
Primer dimerisering resultater.
Løsninger:
Optimerede primere.
2.10.2 Ct-værdi <35, NTC-smeltekurve og gensmeltekurvespidsform overlapper hinanden
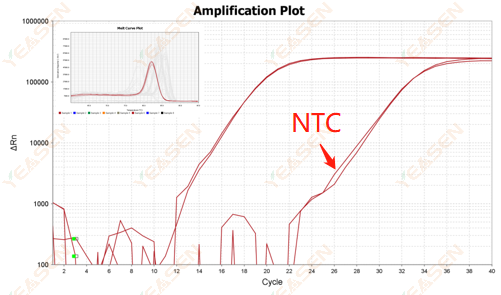
Figur 12. Amplifikationsplot
Mulige årsager:
Forurening af reaktionssystem.
Løsninger:
Tjek forureningskilderne én efter én.
3. Unormal qPCR-smeltekurve
3.1 Enkeltspidssmeltekurve, men ikke skarp
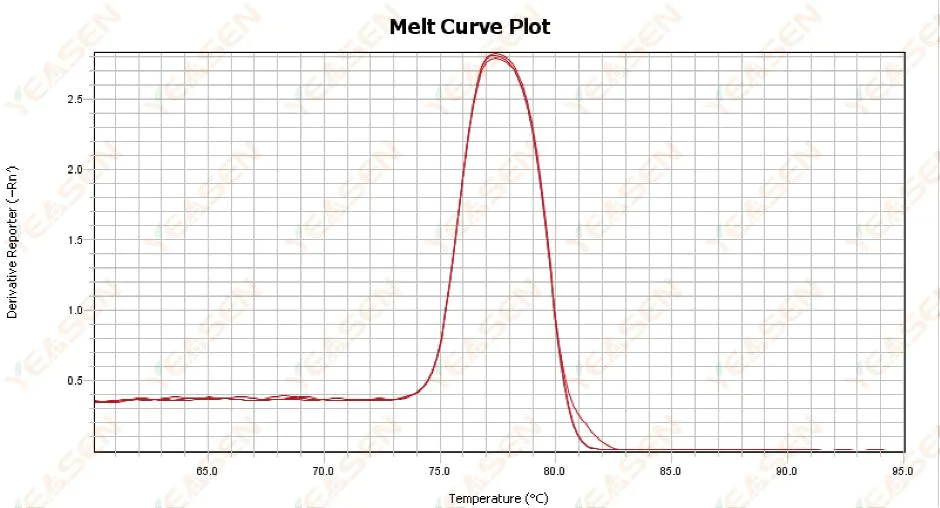
Figur 13. Smeltekurveplot
Mulige årsager:
1) Det er relateret til reagenssammensætning og instrumentmodel.
2) Tilstedeværelse af ikke-specifik amplifikation af fragmenter af samme størrelse.
Løsninger:
1) Temperaturspændet fra peak start til peak off er ikke højere end 7°C. Resultaterne anses for brugbare, dvs. enkelt peak.
2) Højkoncentration agaroseelektroforese (f.eks. 3 % agarose) blev udført for at hjælpe med bestemmelsen.
3.2 Smeltekurven er dobbelt toppet, og den nederste top Tm er før 80°C
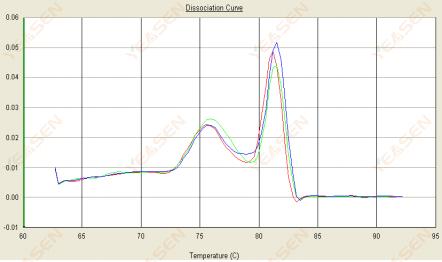
Figur 14. Dissociationskurve
Mulige årsager:
Tilstedeværelse af primer dimerer.
Løsninger:
Forøg annealingstemperaturen, sænk primerkoncentrationen, eller redesign primere.
3.3 Smeltekurven er dobbelt toppet, og den nederste top Tm er efter 80°C
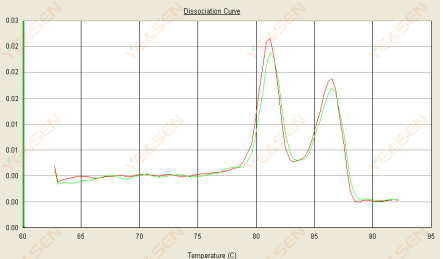
Figur 15. Dissociationskurve
Mulige årsager:
1) Dårlig primerspecificitet fører til amplifikation af ikke-specifikke produkter.
2) gDNA-kontamination.
Løsninger:
1) Blast check primer specificitet, redesign hvis dårlig.
2) Bekræftelse af NRC negativ kontrol, hvis nogen, skal genforberede skabelonen.
3.4 Rodet smeltekurve toppe
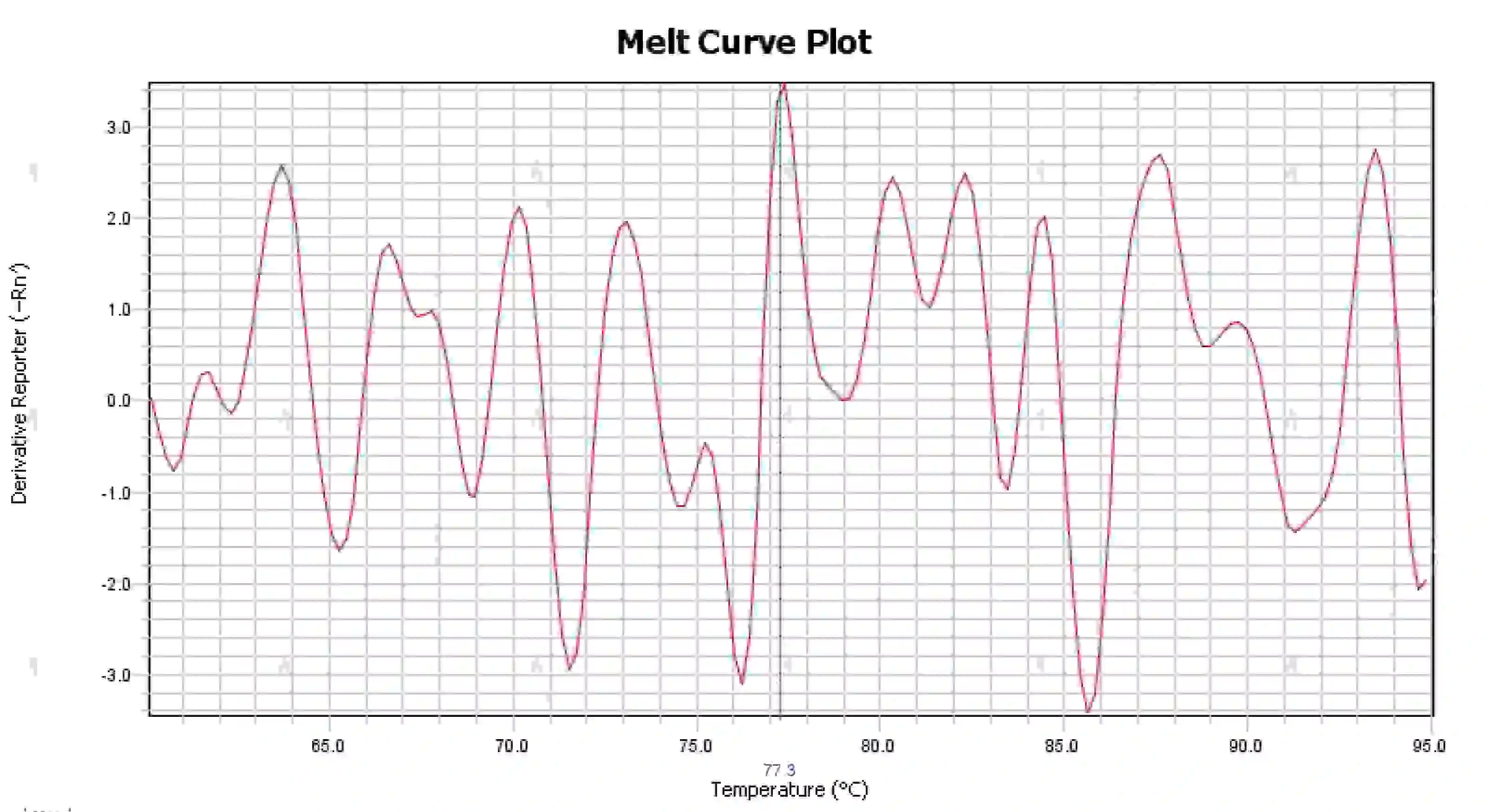
Figur 16. Smeltekurveplot
Mulige årsager:
1) Forurening af reaktionssystemet.
2) Udsættelse af reagenser for stærkt lys eller høj temperatur fører til reagensfejl.
3) Instrumentet har ikke været kalibreret i lang tid.
4) Uoverensstemmelse mellem forbrugsvarer og instrumenter.
Løsninger:
1) Ved at kombinere NTC- og NRC-resultater for at bekræfte kontaminering, anbefales det at udelukke kontaminering fra vand, primere, enzymer og miljøet én efter én.
2) Det anbefales at lave sammenligningsforsøg med nye reagenser.
3) Regelmæssig vedligeholdelse af instrumentkalibrering anbefales.
4) Bekræft kravene til det tilsvarende instrument til forbrugsstoffer.
3.5 Falske toppe i den forreste ende af smeltekurven
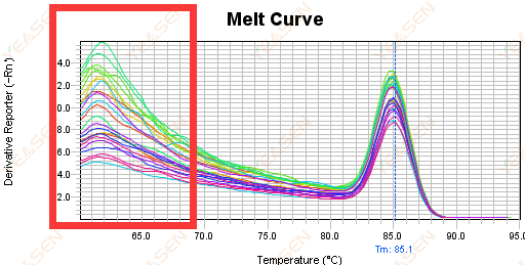
Figur 17. Smeltekurveplot
Mulige årsager:
Rox-koncentrationen matcher ikke modellen.
Løsninger:
Det anbefales at annullere Rox-korrektionen for at se, om smeltekurven er normal.
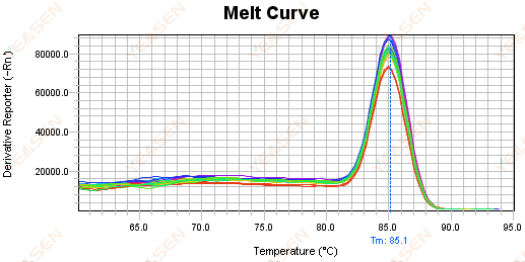
Figur 18. Smeltekurveplot
4. Bestillingsoplysninger
Tabel 1. Bestillingsoplysninger
| Produktnavn | Produktkode | Specifikation |
| Hieff Unicon™ Universal Blue qPCR Master Mix (farvebaseret) | 11184ES03 | 1 ml |
| 11184ES08 | 5×1 ml | |
| 11184ES50 | 50×1 ml | |
| 11184ES60 | 100×1 ml |
5. Relaterede produkter
Produkterne leveret af Yeasen er som følger.
Tabel 2. Relaterede produkter
| Produktpositionering | Produktnavn | Produktkode |
| Et-trins gDNA-fjernelse og revers transkription | Hifair™V et-trins RT-gDNA-fordøjelse SuperMix til qPCR (Forespørgsel) | 11142ES |
| High Sensitivity Master Mix (med gDNA-fjernelsestrin) | Hifair™ III 1st Strand cDNA Synthesis SuperMix til qPCR (gDNA digester plus) | 11141ES |
| High Sensitivity Kit (med gDNA-fjernelsestrin) | Hifair™ III 1st Strand cDNA Synthesis Kit (gDNA digester plus) (Forespørgsel) | 11139ES |
| Almindelig kvantitativ mastermix (sondemetode) | Hieff™ qPCR TaqMan Probe Master Mix (Forespørgsel) | 11205ES |
6. Vedrørende læsning
DNase I og deres anvendelser i biomedicin
Retningslinjen for udvælgelse af omvendt transkriptase
