DNase I og deres anvendelser i biomedicin
Deoxyribonuclease I (DNase I) er en endonuklease, dens anvendelse er ikke kun til at opretholde RNA-integritet, men også til DNA-fodaftryksanalyse, generering af tilfældige DNA-biblioteker, reduktion af klæbrighed i cellelysater eller proteinekstrakter osv. Kort sagt, DNase I kan bruges i næsten enhver applikation, der kræver enzymspaltning af DNA. Det følgende er en detaljeret introduktion til DNase I og dets specifikke anvendelse.
1. Hvad er DNase I?
2. DNase I til fremstilling af DNA-fri RNA-ekstraktion
3. DNase I til in vitro-transkription for at fjerne template-DNA
4. DNase I til rRNA-fjernelse
5. DNase I til DNA-mærkning
6. Andre anvendelser
7. DNase I produktvalgsguide
1. Hvad er DNase I?
Deoxyribonuklease I (DNase I) er en uspecifik endonuklease, der kan fordøje enkelt- eller dobbeltstrenget DNA, som findes i forskellige væv og kropsvæsker. Det kan hydrolysere phosphodiesterbindinger for at producere mono- og oligodeoxynukleotider, der indeholder en 5'-phosphatgruppe og en 3'-OH-gruppe. Det optimale arbejds-pH-område for DNase I er 7-8, dets aktivitet afhænger af Ca2+ og kan aktiveres af divalente metalioner såsom Mn2+, Mg2+, Zn2+ osv. I nærvær af Mg2+ forskyder DNase I tilfældigt ethvert sted af dobbeltstrenget DNA; i nærvær af Mn2+ kan DNase I forskyde DNA dobbeltstrenget på det samme sted for at danne en stump ende eller en 1-2 nukleotid klæbrige ender udhænger.
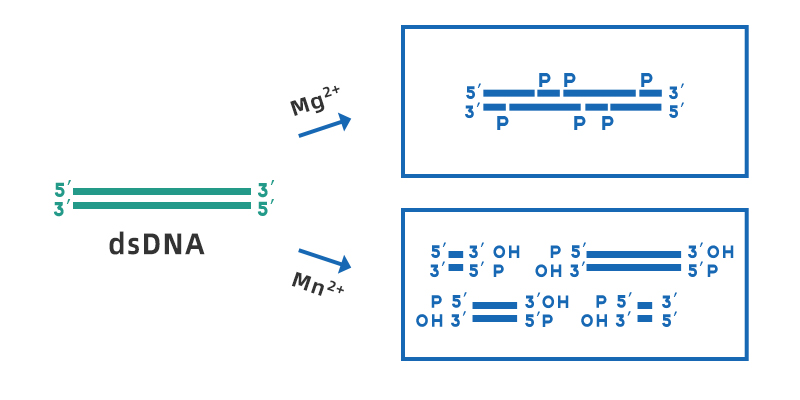
Figur 1. Skematisk diagram af spaltningen af dsDNA med DNase I i nærvær af Mg2+ og Mn2+.
Selvom DNase I-spaltninger generelt anses for at være ikke-specifikke spaltninger, er det mere sandsynligt, at DNase I virker på visse sekvensfragmenter, såsom minor groove-regionen, og er mere tilbøjelig til spaltninger af purin-pyrimidinsekvenser. Men når DNase I virker på heterogent dsDNA, vil alle fire baser blive spaltet, og virkningen på en specifik base vil ikke være mere end 3 gange større end andre basers.
2. DNase I til fremstilling af DNA-fri RNA-ekstraktion
I biologiske eksperimenter er det første trin at forberede nukleinsyre til at studere forskellige funktioner af RNA. Men da DNA og RNA ofte frigives sammen under cellelyseprocessen, kan interferensen i de to ikke undgås, uanset hvilken ekstraktionsopløsning der bruges, så specifikke enzymer skal bruges til at fjerne interferensen. Til højkvalitets RNA-ekstraktion bruges DNase I til at fjerne resterende DNA fra prøven.
DNase I kan nedbryde dobbeltstrenget og enkeltstrenget DNA til oligonukleotider og enkelte nukleotider, og DNA'et i RNA-præparationsproduktet kan effektivt nedbrydes. DNase I inaktiveres derefter ved opvarmning med en stopbuffer. Under opvarmningsprocessen kan hårnålestrukturen af RNA-molekylet åbnes, hvilket letter RNA'ets direkte indtræden i omvendt transkriptionsprocessen.
Kvaliteten af RNA vil direkte påvirke de eksperimentelle data i høj grad. Generelt kan gDNA-rester ikke fuldstændigt undgås under RNA-ekstraktion, derfor anbefales det generelt at behandle RNA-prøver med DNase I for at fordøje resterende gDNA, før man udfordrer nedstrømsapplikationer (f.eks. mRNA-ekspressionsanalyse, transkriptomanalyse osv.). Trinnet med at fordøje gDNA kan udføres under RNA-ekstraktion, efter RNA-ekstraktion eller før RNA-revers transkription.I henhold til produktpositioneringen er produkterne leveret af Yeasen som følger:
Tabel 1: Liste over produkter relateret til DNA-fjernelse af RNA-ekstraktion eller før revers transkription
| Produktpositionering | Produktnavn | Kat # |
| RNA-ekstraktion | TRIeasy™ Total RNA-ekstraktionsreagens [spørge] | 10606ES |
| 19221ES | ||
| MolPure™ Plant Plus RNA Kit [spørge] | 19292ES | |
| MolPure™ viralt DNA/RNA-kit [spørge] | 19321ES | |
| gDNA fjernelse | 10325ES | |
| Omvendt transskription | Hifair™Ⅲ1st Strand cDNA Synthesis SuperMix til qPCR (gDNA digester plus) | 11141ES |
| qPCR | 11184ES |
3. DNase I til in vitro-transkription for at fjerne template-DNA
In vitro transkription (IVT) bruger hovedsageligt DNA som skabelon plus tilsvarende substrater og buffere til at opnå RNA gennem in vitro transkription. I in vitro-transkriptionseksperimenter bruges RNA-polymeraser såsom T7, T3 og SP6 almindeligvis til RNA-syntese. Det syntetiserede RNA kan have DNA-rester. Eliminering af DNA-resterne er gavnligt for udviklingen af downstream-eksperimenter. For eksempel i mRNA-vaccineudviklingsstadiet er fjernelse af rester et kritisk trin, som kan reducere vanskeligheden ved nedstrøms oprensning og øge produktets renhed. DNA-skabelonen fjernes typisk under anvendelse af rekombinant DNase I (RNase-fri).Ifølge mRNA-synteseprocessen er produkterne leveret af Yeasen som følger:
| mRNA synteseproces | Produktnavn | Kat# |
| Skabelon forberedelse | Hieff Canace™ Plus High-Fidelity DNA-polymerase [spørge] | 10148ES |
| 10922ES | ||
| 10125ES | ||
| FuniCut™BsaI [spørge] | 15005ES | |
| FuniCut™ XbaI [spørge] | 15033ES | |
| BspQI[spørg] [spørge] | 16215ES | |
| In vitro transkription | 10623ES | |
| 10624ES | ||
| T7 RNA-polymerase (50 U/μL)[spørge] | 10618ES | |
| 10133ES | ||
| 10620ES | ||
| 10621ES | ||
| Fjern skabelon-DNA | 10611ES | |
| mRNA modifikation | 10614ES | |
| 10612ES | ||
| 10132ES | ||
| 10619ES | ||
| mRNA oprensning | 12602ES |
4. DNase I til rRNA-fjernelse
In vivo er rRNA meget rigeligt og meget konservativt, hvilket er af ringe betydning for opnåelse af biologisk information, så rRNA fjernes ofte først i RNA-bibliotekskonstruktion og -sekventering.På nuværende tidspunkt er fjernelsesmetoden for rRNA hovedsageligt RNase H-fordøjelse. De vigtigste trin i enzymatisk-baseret rRNA-udtømning er vist i figur 2: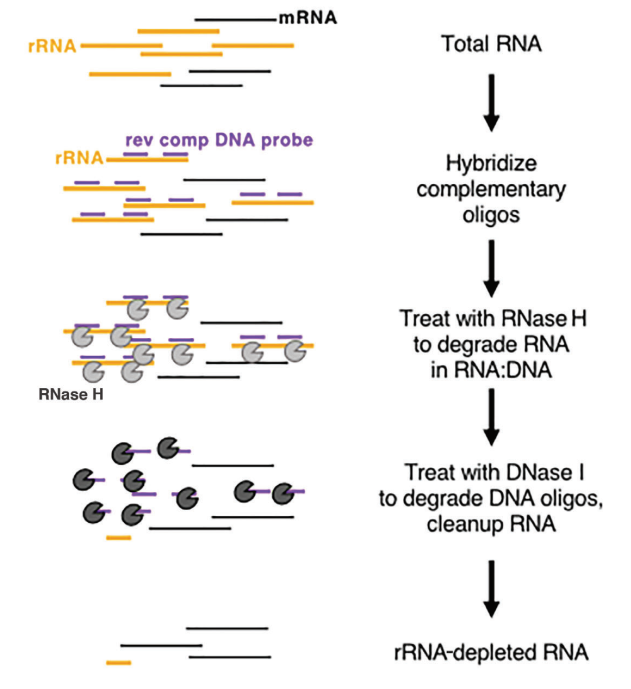
Figur 2: Skematisk diagram af princippet om enzymatisk-baseret rRNA-udtømning (Baldwin, A. et al. 2021, Current Protocols)
Først ekstraheres totalt RNA, hybridiser derefter enkeltstrenget DNA-probe med rRNA, design og syntetiser rRNA-specifik enkeltstrenget DNA-probe, brug derefter RNase H til at nedbryde det hybridiserede rRNA og brug DNase I til at nedbryde DNA-proben. Til sidst forlader ikke-rRNA RNA skabelonen. Produkterne relateret til rRNA-fjernelse leveret af Yeasen er som følger:
| mRNA synteseproces | Produktnavn | Kat# |
| Menneske/mus/rotte rRNA-depletering | Hieff NGS™ MaxUp rRNA Depletion Kit (menneske/mus/rotte) MaxUp [spørge] | 12253ES |
| Plante rRNA udtømning | 12254ES | |
| Fjernelse af ribosomalt RNA og 45S ITS/ETS-regioner fra humant totalt RNA | 12257ES | |
| Nedbrydning af rRNA | 12906ES | |
| Nedbrydning af DNA-sonde | 10325ES |
5. DNase I til DNA-mærkning
Nick-oversættelsen er en af laboratoriets mest almindeligt anvendte deoxyribonukleinsyre-probemærkningsmetoder. Denne metode anvender forskellige enzymatiske aktiviteter af DNA-polymerase I til at inkorporere mærkede deoxyribonukleosidtriphosphater i nyligt syntetiserede DNA-kæder. Ensartet mærkede DNA-prober til høj specifik aktivitet syntetiseres således. Karakteristikaene ved nick-translation er hurtige, enkle, bevidste, høj specificitet og ensartet mærkede prober, som er egnede til længere dobbeltstrenget DNA.Metoden realiseres ved den kombinerede virkning af DNase I og E. coli DNA-polymerase I. De vigtigste trin i DNA-mærkning ved nick-translation er vist i figur 3:
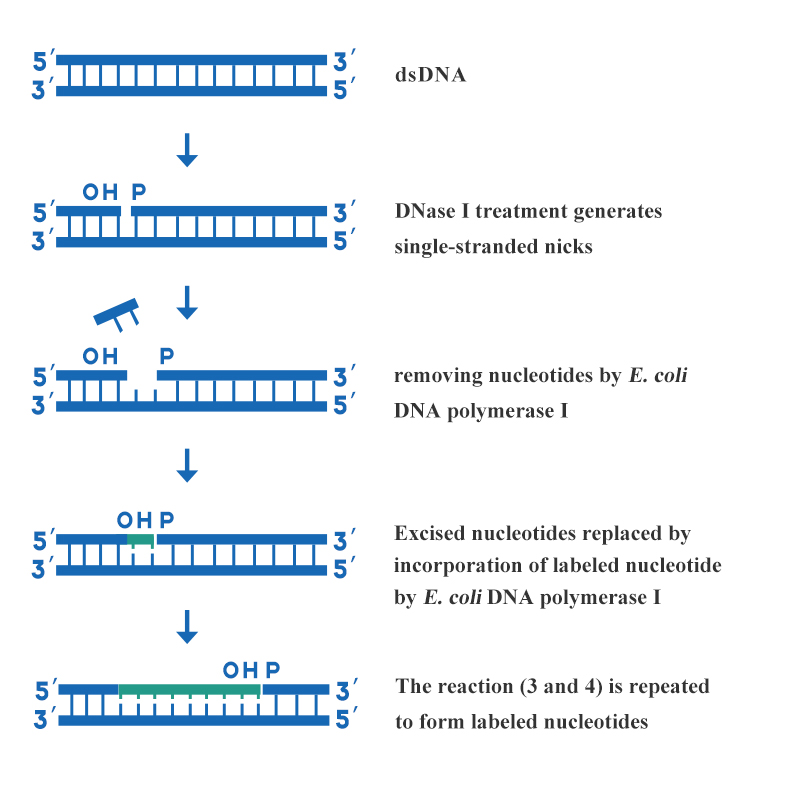
Figur 3: Skematisk diagram af DNA-mærkning ved nick-translation
En passende koncentration af DNase I bruges til at skabe flere enkeltstrengede huller på hver streng af det dobbeltstrengede DNA, der skal mærkes, og 3'-hydroxylterminalen dannes ved mellemrummet. Brug 5'→3'-exonukleaseaktiviteten af E. coli DNA-polymerase I til at skære et nukleotid fra 5'-enden af nicket, og på samme tid introducerer 5'→3'-polymeraseaktiviteten af E. coli DNA-polymerase I et nukleotid mærket med 3'-enden af mellemrummet for at reparere mellemrummet. Når mellemrummet bevæger sig langs DNA-strengen, inkorporeres de mærkede nukleotider i den nyligt syntetiserede streng.Produkterne relateret til DNA-mærkning leveret af Yeasen er som følger:
| Produktpositionering | Produktnavn | Kat# |
| Almindelig | Deoxyribonuclease I (DNase I) fra bovin bugspytkirtel [spørge] | 10607ES/10608ES |
| RNase fri | 10325ES | |
| E. coli kilde | 12903ES |
6. Andre anvendelser
Ovenstående er flere almindeligt anvendte applikationer. Flere anvendelser af DNase I inkluderer følgende, såsom DNase I footprinting assay og DNase I hypersensitive sites. DNase I footprinting assay er en detektionsmetode, der nøjagtigt kan identificere bindingsstederne for DNA-bindende proteiner på DNA. Når et protein binder til et DNA-fragment, kan det beskytte bindingsstedet mod at blive beskadiget af DNase I, og DNA-fragmenterne vil blive efterladt efter enzymfordøjelse ("fodaftryk"), og dets sekvens kan bestemmes. På gelbilledet er der ingen bånd, hvor DNA'et binder til proteinet. For at læse mere klik på linket.DNase I hypersensitive steder henviser til spaltninger på et lille antal specifikke steder, når kromatin behandles med lav DNase I, og disse specifikke steder kaldes DNase I hypersensitive sites. Princippet er, at når et gen er i en transkriptionelt aktiv tilstand, er kromatinet, der indeholder genet, væsentligt mere følsomt over for DNase-nedbrydning end det inaktive område. For at læse mere klik på linket.
7. DNase I produktvalgsguide
Yeasen Biotechnology (Shanghai) Co., Ltd., grundlagt i 2014, er en højteknologisk virksomhed, der beskæftiger sig med forskning og udvikling og produktion af værktøjsenzymråmaterialer og antigenantistoffer. Dets produkter omfatter molekylære diagnostiske enzymer, proteiner og antistoffer, der bruges i lægemidler, fødevaresikkerhedstest, avl, retfærdighed og andre industrier. Vi er forpligtet til at give kunder inden for life science produkter og tjenester af høj kvalitet. Retningslinjerne for produktkøb for DNase I er som følger:| Produktnavn (kat #) | Produktpositionering | Anbefalede applikationer |
| DNase I fra bovin bugspytkirtel (CAT#10607,10608)[spørge] | RNase fjernet, ikke fundet | Anvendes hovedsageligt i proteinforskning: DNA-fjernelse fra proteinpræparater. |
| Rekombinant DNase I (RNase-fri)(KAT#10325) | RNase-fri, til forskning | Ideel til en række anvendelser: DNA-fjernelse fra RNA- og proteinpræparater såsom RNase-følsomme cDNA-biblioteker eller prøveforberedelse til RT-PCR-eksperimenter. |
| UCF.ME™ Deoxyribonuclease I (DNase I) GMP-kvalitet(KAT#10611) | RNase-fri, GMP farmaceutisk kvalitet. | Ideel til en række anvendelser: DNA-fjernelse fra RNA- og proteinpræparater såsom RNase-følsomme cDNA-biblioteker eller prøveforberedelse til RT-PCR-eksperimenter. |
Angående læsning:
GMP-grade reagenser til mRNA in vitro syntese
Principperne for DNase I-fodaftryk og dets biomedicinske anvendelser
Referencer
1. Baldwin A, Morris AR, Mukherjee N. En nem, omkostningseffektiv og skalerbar metode til at udtømme humant ribosomalt RNA for RNA-seq[J]. Nuværende protokoller, 2021.
2. Song C, Zhang S, Huang H. Valg af en passende metode til identifikation af replikationsoprindelse i mikrobielle genomer[J]. Frontiers in Microbiology, 2015, 6:1049.
