Arbejd langs begge linjer, miRNA-ekspressionsanalyse gjort lettere
Længden af modent miRNA er kun omkring 20 nt, normalt vil den forreste primer dække sin fulde længde eller endda have en overskydende del, og den omvendte primer vil ikke have nogen steder at placere. Den nuværende markedsløsning er at øge længden af det omvendte transkript under omvendt transkription. Almindelige metoder omfatter halemetoden og stammeløkkemetoden.
1. Introduktion
2. Anbefalede produkter
3. Relateret produktliste
1. Introduktion
MikroRNA'er (miRNA'er) er enkeltstrengede små RNA'er med en størrelse på omkring 21-23 baser, som er genereret fra enkeltstrengede RNA-precursorer med en hårnålestruktur på omkring 70-90 baser efter Dicer-enzymbehandling. miRNA binder hovedsageligt specifikt til 3'-endens ikke-kodende region af målgen-mRNA og regulerer translationsprocessen af post-transkriptionelt målgen-mRNA, og blokerer derved mRNA-translation og fremmer mRNA-nedbrydning, hvilket resulterer i nedsat proteinekspression. MiRNAs rolle er involveret i forekomsten og udviklingen af forskellige hjerte-kar-sygdomme såsom ontogeni, vævsdifferentiering, celleproliferation og apoptose.
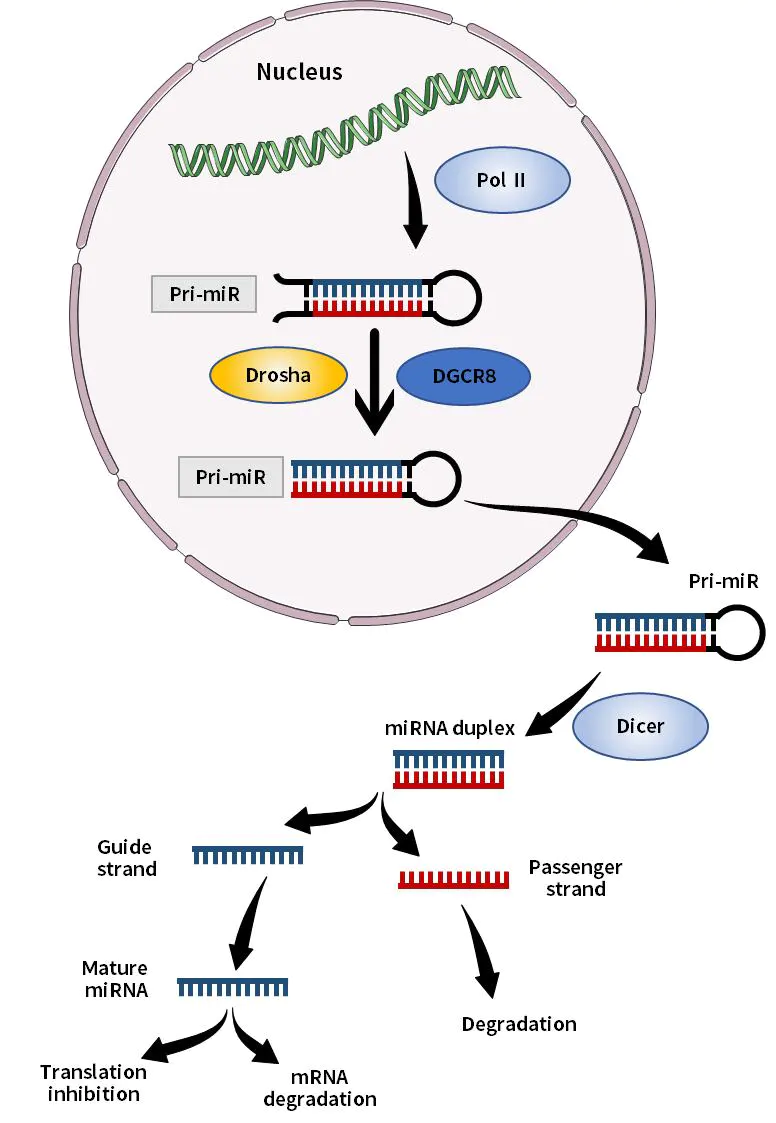
Figur 1. Skematisk diagram af miRNA-syntese in vivo
Længden af modent miRNA er kun omkring 20 nt, normalt vil den forreste primer dække sin fulde længde eller endda have en overskydende del, og den omvendte primer vil ikke have nogen steder at placere. Den nuværende markedsløsning er at øge længden af det omvendte transkript under omvendt transkription. Almindelige metoder omfatter halemetoden og stammeløkkemetoden.
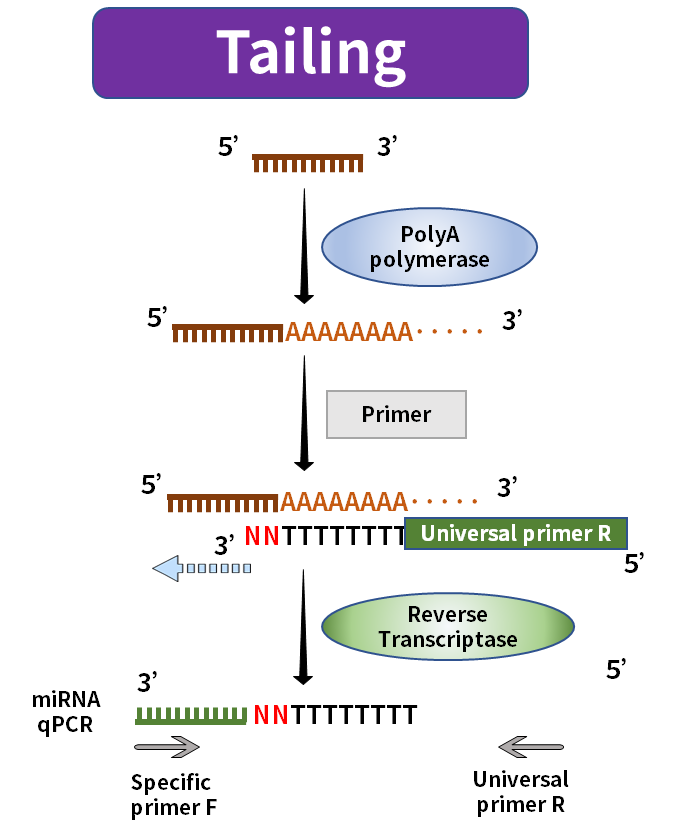
Figur 2. miRNA-syntese ved tailing-metode
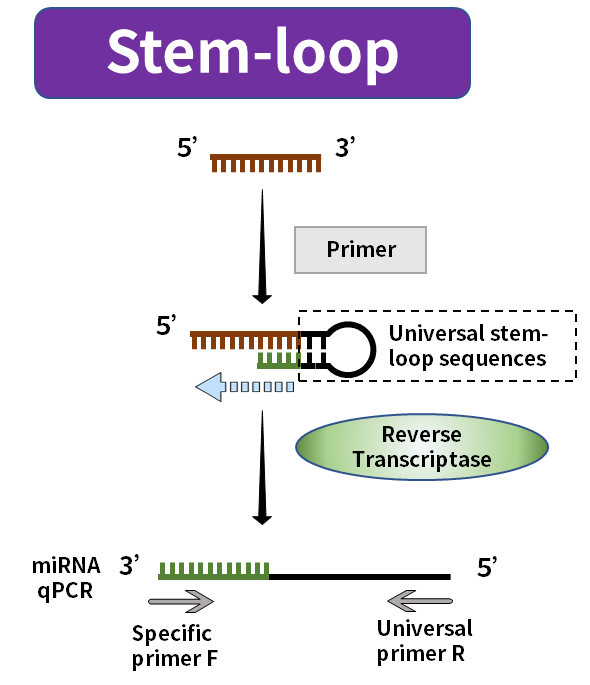
Figur 3. miRNA-syntese ved stam-loop-metoden
Halemetoden bruger PolyA-polymerase til at tilføje Poly(A)-hale til modent miRNA og bruger Oligo-dT med den universelle sekvens som en revers transkriptionsprimer til at syntetisere det forlængede miRNA svarende til den første streng af cDNA. Stam-loop-metoden bruger den universelle sekvens foldet ind i stam-loop-strukturen som en primer til at syntetisere den første streng af cDNA'et svarende til det aflange miRNA. Stængelløkkestrukturen vil blive åbnet ved en passende temperatur under den efterfølgende amplifikationsproces og kombineret med de relevante amplifikationsprimere.
Tabel 1.Sammenligning af Tailing og Stem-loop
| Kategori | Tailing | Stængel-løkke |
| Anvendelighed | Velegnet til at studere flere forskellige miRNA'er samtidigt | Velegnet til nøjagtig kvantitativ påvisning af nogle få miRNA'er i en prøve |
| Fordel | En enkelt revers transkription kan detektere flere miRNA'er med høj gennemstrømning | Kun ét miRNA kan påvises ved omvendt transkription ad gangen med stærk specificitet |
| System | Sammen med det involverede miRNA, revers transkription i det samme reaktionssystem med høj nøjagtighed | Internt involverede miRNA'er adskilles og omvendt transskriberet i de to systemer, hvilket er tilbøjeligt til fejl |
| Specificitet | ★★★★☆ | ★★★★★ |
| Følsomhed | ★★★★★ | ★★★★☆ |
| Tid | ★★★★☆ | ★★★★★ |
2. Anbefalede produkter
2.1 Yeasen miRNA tailed inversion kvantitativ kombination (11148ES&11171ES)
Figur 4. 11148ES-Hifair™ miRNA 1st Strand cDNA Synthesis Kit
Sættet anvender tailing-metoden til at reversere transkription af miRNA første-strengs cDNA. 2×Hifair™ miRNA RT-bufferen i produktet indeholder alle råmaterialer og primere til miRNA-halereaktion og omvendt transkriptionsreaktion og er omhyggeligt optimeret. Det kan sikre, at poly(A)-modifikations- og revers transkriptionsprocesserne af miRNA 3'-enden udføres effektivt samtidigt.
Figur 5. 11171ES -Hieff™ miRNA Universal qPCR SYBR Master Mix
En ny generation af forblandede fluorescens kvantitative PCR detektionsreagenser specielt udviklet til miRNA kvantitativ detektion, indeholdende speciel ROX Passive Reference Dye, velegnet til alle qPCR instrumenter, ingen grund til at justere koncentrationen af ROX på forskellige instrumenter, kun i forberedelsesreaktionen Amplifikation kan udføres ved at tilføje primere og skabeloner til systemet. DNA-polymerasen anvender kemisk modificeret hot-start polymerase og samarbejder med et særligt buffersystem, som gør reaktionsspecificiteten og sensitiviteten højere og kan kvantificere præcist i et bredere område.
2.2 Produktets ydeevne
2.2.1 Kompatibel med qPCR-instrumenter på alle platforme

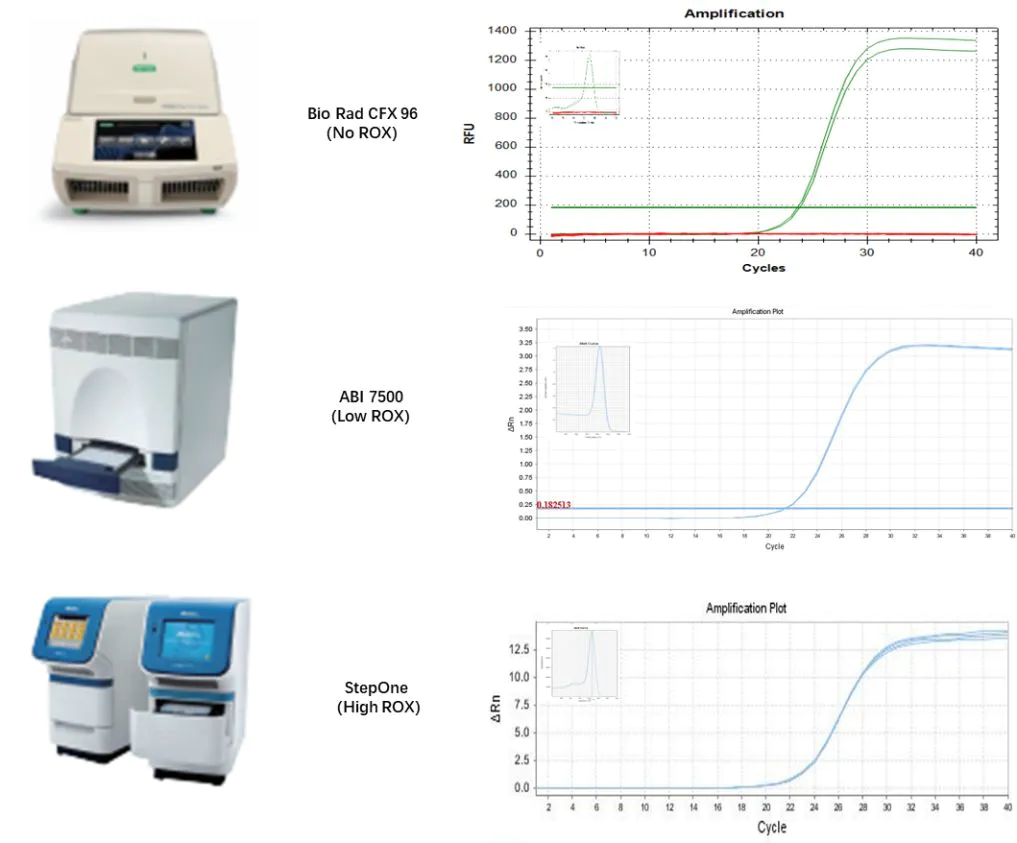
Figur 6. Kompatible resultater med forskellige qPCR-instrumenter
Ved at bruge det totale RNA fra 293T-celler som skabelon blev revers transkription udført med Hifair™ miRNA 1st Strand cDNA Synthesis Kit (Cat # 11148ES).Det opnåede cDNA blev fortyndet 10 gange og brugte derefter Hieff™ miRNA Universal qPCR SYBR Master Mix (Cat # 11171ES) til at detektere de interne referencegener af hsa-miR-let-7b-5p, hsa-miR-let-7c-5p og U6, henholdsvis.
2.2.2 Høj detektionshastighed, op til 10 pg
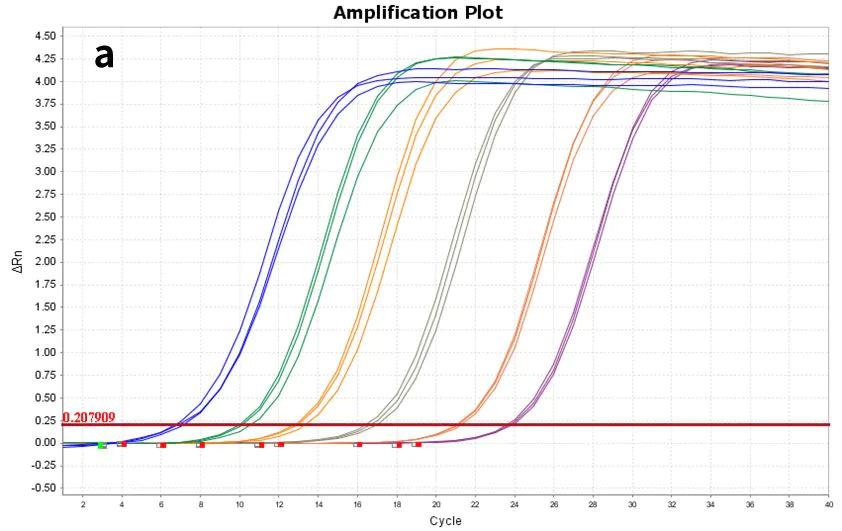
Figur 7. Amplifikationsplot
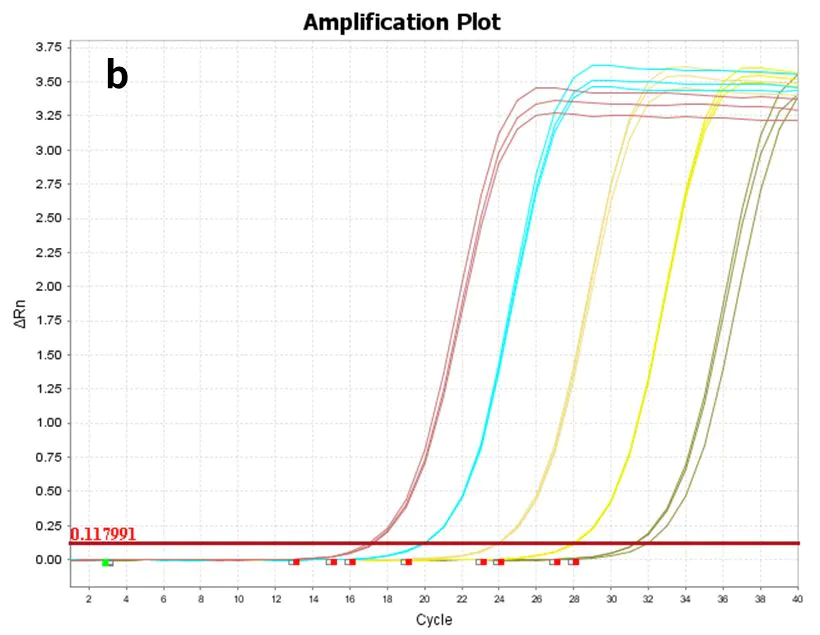
Figur 8. Amplifikationsplot
Ved at bruge syntetisk hsa-miR-let-7e-5p(a) og 293T celle Total RNA(b) som skabeloner, fortyndet til henholdsvis følgende gradienter: 60 kopier til 606 kopier (6 gradienter) og 10 pg-100 ng (5 gradienter), omvendt transkriberet Strand-kit med Synthest Strand™ miDNA (Cat # 11148ES), og det resulterende cDNA blev detekteret med Hieff™ miRNA Universal qPCR SYBR Master Mix (Cat # 11171ES) til hsa-miR-let-7e -5p genekspression.
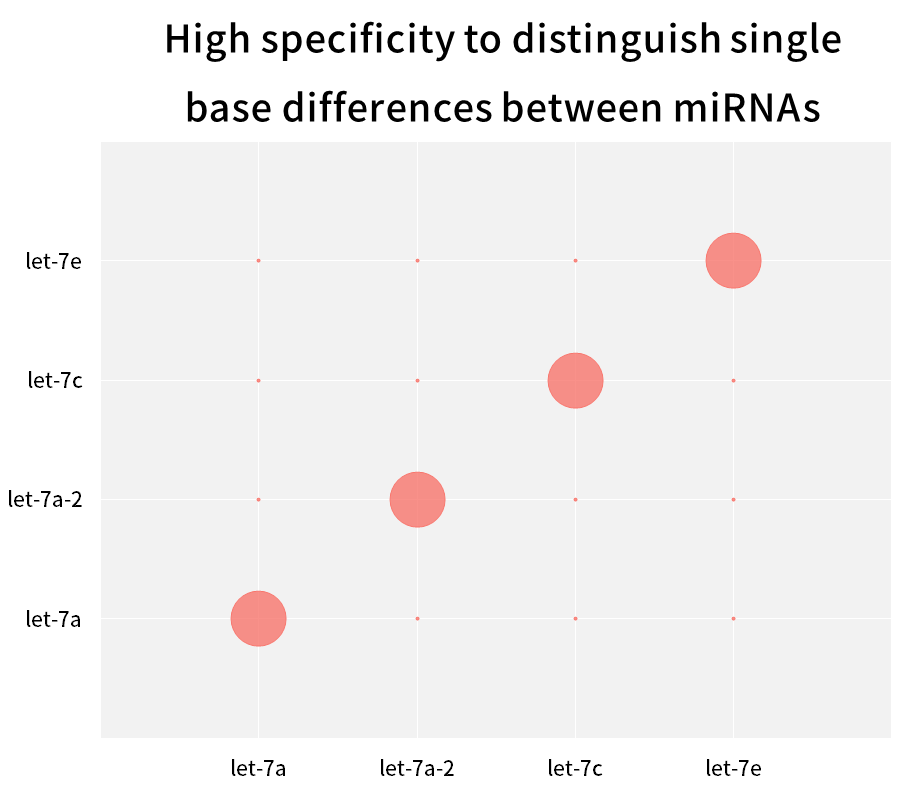
2.2.3 Høj specificitet, i stand til at skelne enkeltbaseforskelle mellem miRNA'er i samme familie
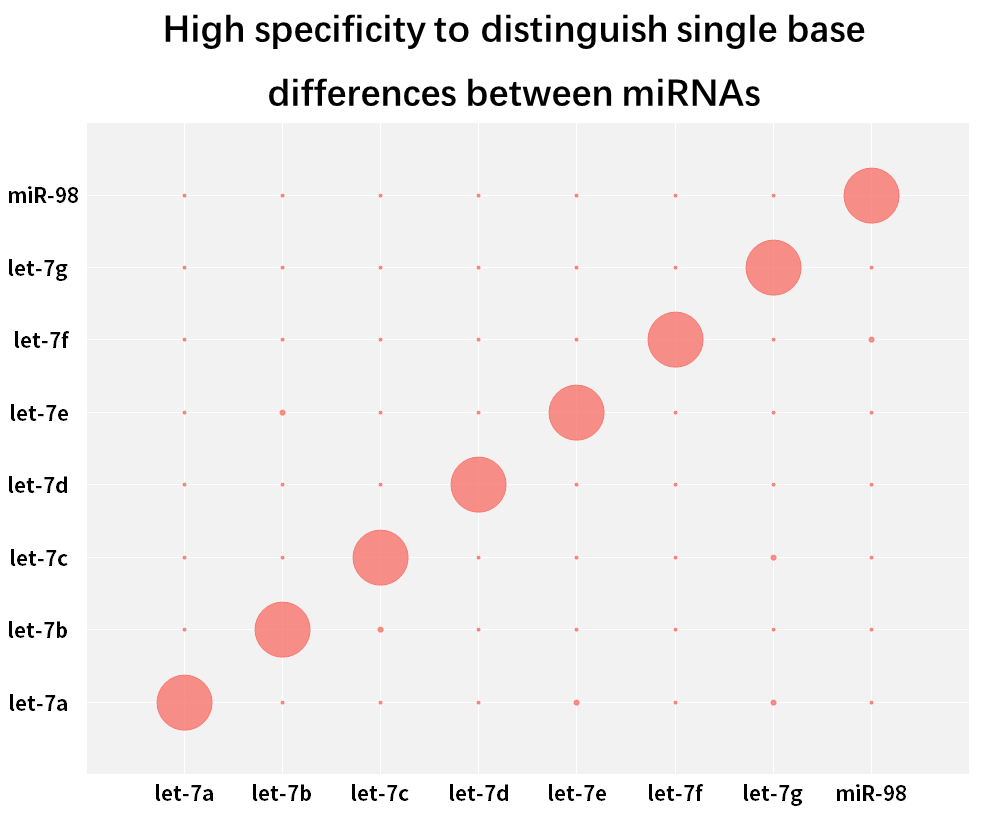
Figur 9. Skelne enkeltbaseforskelle mellem miRNA'er
Den humane hsa-let-7-familie har flere miRNA'er med få baser, der adskiller sig fra hinanden, eller endda kun en enkelt baseforskel. Syntetiske miRNA'er for hver Let-7 isoform (hsa-miR-let-7a~Let-7i, has-miR-98) blev omvendt transskriberet til cDNA ved hjælp af Hifair™ miRNA 1st Strand cDNA Synthesis Kit (Cat # 11148ES) , ved at bruge Hieff™ miRNA Universal qPCR 1C1BR amifyES (1C1Atify) miRNA'er separat med forskellige primere, og beregne matchhastigheden ved hjælp af ligning 2-ΔCT x 100%. Resultaterne viser, at nært beslægtede subtype familiemedlemmer effektivt kan skelnes.
2.2.4 Fremragende forstærkningseffektivitet
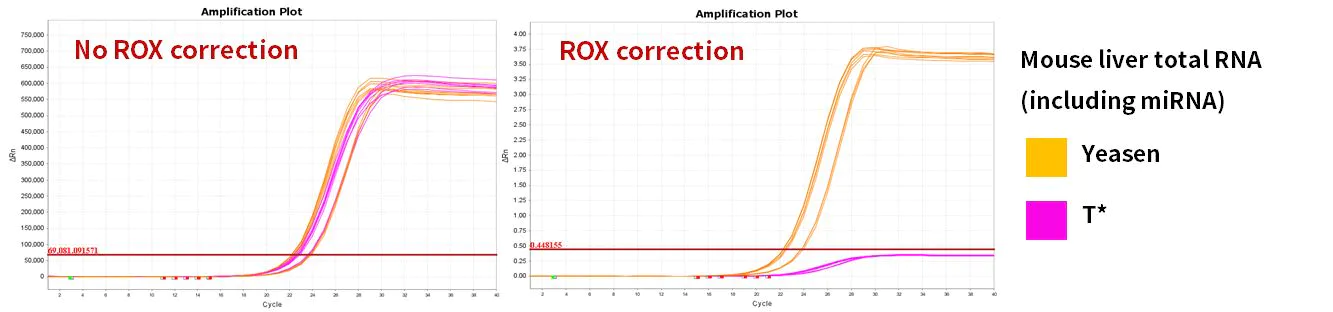
Figur 10. Amplifikationseffektivitet
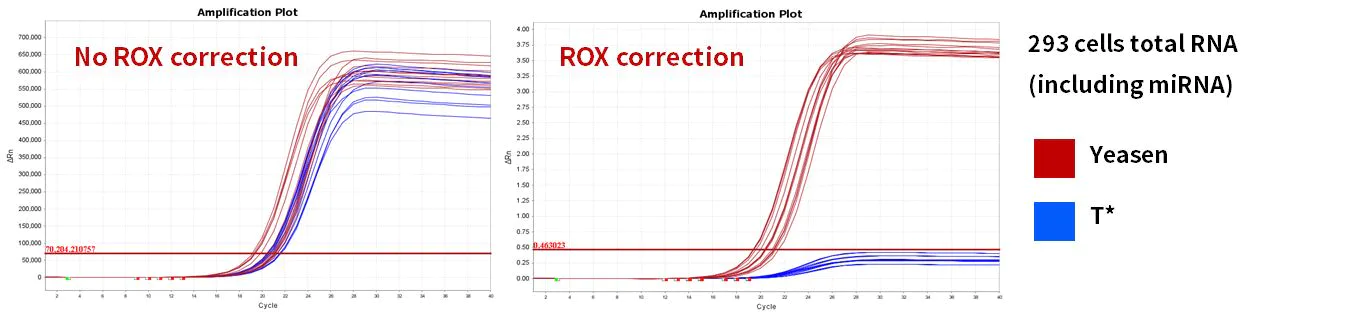
Figur 11. Amplifikationseffektivitet
miR-let-7b-5p, miR-let-7c-5p, miR-let-7e-5p, miR-let-7f-5p gener blev amplificeret ved hjælp af humane 293T-celler og muselever Total RNA som skabeloner. Resultaterne viste, at sammenlignet med lignende produkter matchede Hifair™ miRNA 1st Strand cDNA Synthesis Kit (Cat# 11148ES) reaktionssystemet i Hieff™ miRNA Universal qPCR SYBR Master Mix (Cat# 11171ES) med fremragende ydeevne.
2.2.5 Fremragende stabilitet

Figur 12. Produktstabilitetsanalyse ved 37℃ i 7 dage

Figur 13. Analyse af resultater efter 50 fryse-tø-cyklusser
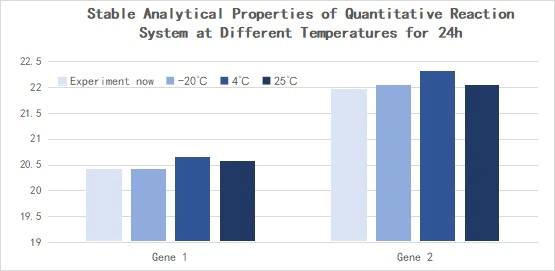
Figur 14. Stabile analytiske egenskaber af det kvantitative reaktionssystem ved forskellige temperaturer i 24 timer
Omvendt transkription blev udført med Hifair™ miRNA 1st Strand cDNA Synthesis Kit (Cat # 11148ES) under anvendelse af 293T celle Total RNA som skabelon, og det opnåede cDNA blev fortyndet 10 gange og detekteret med Hieff™ miRNA Universal qPCR SYBR Master Mix (Cat # 11171ES). Ekspression af hsa-miR-let-7b-5p, hsa-miR-let-7c-5p, hsa-miR-let-7e-5p. △Ct<0,5.
2.3 Yeasen miRNA stam-loop inversion kvantitativ kombination (11139ES&11170ES)
Figur 15. 11139ES-Hifair™ III 1st Strand cDNA Synthesis Kit (gDNA digester plus)
Sættet er udviklet baseret på Hifair™ III Reverse Transcriptase.Enzymets cDNA-syntesehastighed er hurtig, og den termiske stabilitet er væsentligt forbedret. Samtidig øger enzymet affiniteten med templaten og er velegnet til revers transkription af en lille mængde templates og lavkopierede gener. Hifair™ III Reverse Transcriptases evne til at syntetisere fuldlængde cDNA er også blevet forbedret, hvilket muliggør syntese af cDNA op til 19,8 kb.

Figur 16. 11170ES-Hieff™ miRNA Universal qPCR SYBR Master Mix
Dette produkt er en speciel mastermix til miRNA-kvantificering ved hjælp af SYBR Green I kimærisk fluorescensmetode. Fordi miRNA-sekvenser er korte, og miRNA-sekvenser i samme familie ofte er meget ens, er specificitet ekstremt påkrævet under kvantificering. Dette produkt er baseret på hot-start DNA polymerase, med optimeret buffer, som i høj grad kan reducere uspecifik amplifikation; samtidig gør det specielle ROX Reference Dye mastermixet velegnet til alle qPCR-instrumenter, og der er ingen grund til at justere ROX på forskellige instrumenter Amplifikation kan udføres ved at tilføje primere og skabeloner ved klargøring af reaktionssystemet.
2.3.1 Kompatibel med qPCR-instrumenter på alle platforme
Figur 17. Kompatible resultater med forskellige qPCR-instrumenter
Ved at bruge totalt RNA fra museleverceller som skabelon blev revers transkription udført med Hifair™ III 1. Strand
cDNA Synthesis Kit (gDNA digester plus) (Cat # 11139ES), og det opnåede cDNA blev fortyndet 40 gange og brugt Hieff™ miRNA Universal qPCR SYBR Master Mix (Cat # 11170ES) blev brugt til at amplificere det humane mmu-miR-125a-gen.
2.3.2 Høj følsomhed, op til 10 pg

Figur 18. Følsomhedsdetektion
Ved at bruge totalt RNA fra museleverceller som skabelon blev revers transkription udført med Hifair™ III 1st Strand cDNA Synthesis Kit (gDNA digester plus) (Cat # 11139ES), og det opnåede cDNA blev seriefortyndet 10 gange (interval 10pg/μL). -0,1 μg/μL), amplificere generne mmu-miR-125a, mmu-miR-132 og mmu-miR-351 af murin oprindelse.
2.3.3 Høj specificitet, i stand til at skelne enkeltbaseforskelle mellem miRNA'er i samme familie
Figur 19. Skelne enkeltbaseforskelle mellem miRNA'er
Den humane hsa-let-7-familie har flere miRNA'er med få baser, der adskiller sig fra hinanden, eller endda kun en enkelt baseforskel. Disse miRNA'er blev amplificeret separat med forskellige primere ved hjælp af Hieff™ miRNA Universal qPCR SYBR Master Mix (Cat# 11170ES), og matchningshastigheden blev beregnet ved hjælp af formlen 2^-Δct x 100%.
2.3.4 God repeterbarhed

Figur 20. Gentagelsesdetektion
Ved at bruge det totale RNA fra humane 293T-celler som en skabelon for revers transkription blev revers transkription udført med Hifair™ III 1st Strand cDNA Synthesis Kit (gDNA digester plus) (Cat # 11139ES), og det opnåede cDNA blev fortyndet 50 gange og brugt Hieff™ miRNASYBR1CR qP1C Universal Mix at amplificere det humane hsa-let-7a-gen i et 90-brønds replikateksperiment.
2.3.5 God stabilitet

Figur 21.Stabilitetsanalyse af 11170ES ved forskellige temperaturer i 14 dage
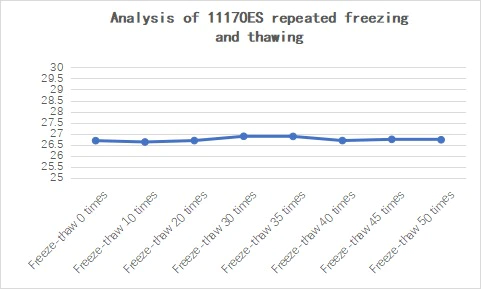
Figur 22. Analyse af 11170ES gentagen frysning og optøning

Figur 23. Stabile analyseegenskaber af det kvantitative reaktionssystem ved forskellige temperaturer i 24 timer
Det totale RNA fra museleverceller blev brugt som en skabelon til revers transkription med Hifair™ III 1st Strand cDNA Synthesis Kit (gDNA digester plus) (Cat # 11139ES). Det opnåede cDNA blev fortyndet 100 gange for at amplificere det murine mmumiR-132-gen.
3. Relateret produktliste
Yeasen Biotechnology (Shanghai) Co., Ltd., grundlagt i 2014, er en højteknologisk virksomhed, der beskæftiger sig med forskning og udvikling og produktion af værktøjsenzymråmaterialer og antigenantistoffer. Dets produkter omfatter molekylære diagnostiske enzymer, proteiner og antistoffer, der bruges i lægemidler, fødevaresikkerhedstest, avl, retfærdighed og andre industrier. Vi er forpligtet til at give kunder inden for life science produkter og tjenester af høj kvalitet. De relaterede produkter leveret af Yeasen er som følger:
Tabel 2. Relateret produktliste
| Metode Type | Eksperimentel procedure | Produktnavn | Produktkode |
| Tailing | Omvendt transskription | 11148ES | |
| qPCR | 11171ES | ||
| Stængel-løkke | Omvendt transskription | Hifair™ III 1st Strand cDNA Synthesis Kit (gDNA digester plus) (Forespørgsel) | 11139ES |
| qPCR | Hieff™ miRNA Universal qPCR SYBR Master Mix (Forespørgsel) | 11170ES |