Hvordan vælger du dine NGS-adaptere?
NGS Adapter, en væsentlig del af næste generations sekventeringsbibliotek, spiller en rolle ved at forbinde det testede DNA-fragment og Flow-cellen (Sequencing-chip). Effektiviteten af samlingen er en vigtig faktor for at bestemme kvaliteten og udbyttet af biblioteket. Så hvad er en NGS-adapter? Hvad er de almindelige typer af NGS-adaptere? Og hvordan vælger du de rigtige NGS-adaptere til dine sekventeringsplatforme?
1. Hvad er NGS Adapter?
2. Hvilke faktorer bør du overveje, når du vælger NGS-indekset?
3. Hvad er de almindelige typer af indekser?
4. Hvad er de almindelige typer af NGS-adaptere?
- UMI adapter
- Komplet adaptere
- Ufuldstændige adaptere
- Tn5 adaptere
5. Hvordan vælger du de rigtige NGS-adaptere til dine sekventeringsplatforme?
6. Angående læsning
1. Hvad er NGS Adapter?
NGS Adapter, en serie af adaptere i sekventering, er en kort nukleotidsekvens med en kendt sekvens. Det ligeres til begge ender af målnukleinsyrefragmentet. Under sekventering starter den sekventering ved at hybridisere med den kendte sekvens på Flow-cellen for at kombinere biblioteket med chippen. Så hvad er strukturen af NGS-adapteren?
Tager man Illumina-platformen som et eksempel, kan en NGS-adapter opdeles i tre dele:
P5 og P7: Sekvens kombineret med P5 og P7 ender på Flow-cellen for at fiksere biblioteket på sekventeringschippen, hvilket letter cluster-reaktion gennem Bridge-PCR.
Rd1 SP og Rd2 SP (Read1/Read2 sekventeringsprimer): Bindingsområder af sekventeringsprimere, der angiver den position, hvor sekvensen begynder at blive læst.
Indeks (også kendt som stregkode): en kendt syntetisk sekvens, der bruges til at skelne mellem forskellige prøver i sekventeringen af det blandede bibliotek.
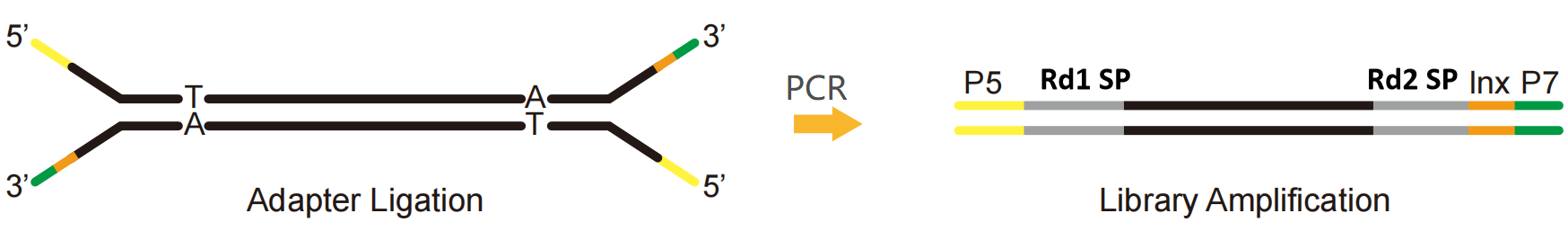
Fig. 1 Illumina platform enkelt indeks bibliotek

Fig. 2 MGI platform single-end indeks bibliotek
Med stigningen i sekventeringsgennemløb kan flere prøver sekventeres på samme tid. Så det er særligt vigtigt, hvordan man skelner forskellige prøver. Som nævnt før bruges indekssekvenser af NGS-adapter til at differentiere forskellige prøver i næste generations sekventering (NGS). Hvilke faktorer skal du overveje, når du vælger indeks? Fortsæt venligst med at læse...
2. Hvilke faktorer bør du overveje, når du vælger indeks?
Indekset er generelt i længden 6nt-18nt og er opdelt i et enkelt indeks og et dobbelt indeks i henhold til antallet af indekser. Dobbeltindekset er placeret i begge ender af det fragment, der skal testes. Basisbalance og fluorescensbalance bør tages i betragtning ved valg af en indekskombination.
Basisbalance refererer til balancen mellem flere indekser, snarere end basisbalancen i et enkelt indeks. Det skal overvejes ud fra både basistyper og basisfordeling. Kombinationsprincippet er, at de fire baser A/T/C/G i samme gruppe af indekser skal medtages, og andelen af disse fire baser er tæt, og udgør henholdsvis ca. 25%.
Fluorescenssignalbalance refererer til valget om at sikre balancen af fluorescerende signaler, når basisbalancen ikke kan garanteres. I 4-kanals sequencer i Illumina platformen er dG/dT mærket med grøn fluorescens, og dC/dA er mærket med rød fluorescens. Under sekventering skal både grønne og røde fluorescenssignaler eksistere i hver cyklus for at sikre vellykket sekventering. Derfor bør balancen mellem det grønne signal og det røde signal tages i betragtning ved valg af indeks.
3. Hvad er de almindelige typer af indekser?
De almindelige dobbeltindekser inkluderer normalt Unique Dual Index (UDI), Unique Dual Barcode (UDB) og Combined Dual Index (CDI), som reducerer indekshopping og fejltildeling betydeligt.
UDI&UDB: indekserne i begge ender er en-til-en-korresponderende, designet i grupper og kan krydsverificeres i begge ender;
Et Stubby UDI Primer Kit til Illumina leveret af Yeasen (Cat#12404ES/12405ES)>>
CDI: indekserne i begge ender kan kombineres i henhold til visse krav for at danne et dobbeltendet indeksbibliotek;
En 384 CDI Primer til Illumina,Set1-Set2 leveret af Yeasen (Cat#12412ES/12413ES)>>
For at forbedre gennemløbs- og amplifikationseffektiviteten og reducere sekventeringsomkostningerne introducerede Illumina array flow cell (PFCT) og eksklusive amplification (ExAmp) klyngeteknologi til Novaseq og andre high-throughput sequencere, men utilsigtet forstærkede prøvelabel mismatch fænomenet og indekshopping.

Fig. 3 Illumina forskellige instrumentmodeller anvender ikke-mønstrede flowceller eller mønstrede flowceller
For at kompensere for indeksering-problemet, der fremhæves af sekventeringsplatforme såsom HiSeq3000/4000, HiSeq X Series og NovaSeq, foreslog Illumina strategien med at placere indekset i begge ender af biblioteket, hvilket kan udføre bilateral verifikation og eliminere de mismatchede adaptere. Ved brug af de unikke indekser i begge ender, vil indeksfejlallokeringsraten blive reduceret til 0,01 %. Sammenlignet med den tidligere konventionelle indekspermutationsgruppekombinationsmetode vil indekshop blive reduceret med to størrelsesordener.
I konstruktionen af et PCR-frit bibliotek er en single-end indeksadapter tilgængelig. Etiketmismatch er hovedsageligt forårsaget af sekventeringsfejl. Samlet set er mærkningsmismatch-raten lav (gennemsnitlig 0,0004 %, op til 0,001 %). Men i konstruktionen af et målrettet capture-bibliotek forstærkes krydstaleproblemet, fordi flere trin vil føre til etiketmismatch, og UDI/UDB/CDI-adaptere bruges normalt.
4.Hvad er de almindelige typer af NGS-adaptere?
Med udviklingen af sekventeringsteknologi er der flere og flere typer adaptere, såsom enkeltindeks/dobbeltindeksadaptere (som nævnt i afsnit 3), UMI-adaptere, transposase-adaptere, komplette/ufuldstændige adaptere osv., som er velegnede til en række forskellige anvendelsesscenarier. Denne del sorterer systematisk disse adaptere fra for at give dig grundlaget for adaptervalg.
4.1 UMI-adapter
Unique molecular identifier (UMI) adapter er et kantværktøj til lavfrekvent mutationsdetektion og absolut kvantificering. UMI er en tilfældig syntetisk sekvens med en kendt sekvens. Den kan udformes som en fuldstændig tilfældig nukleotidkæde, delvis degenereret nukleotidkæde eller fast nukleotidkæde.Længden er normalt 10nt (single-ended UMI) eller 5-8nt (double-ended UMI). Dens funktion er at fryse DNA-fragmenternes tilstand før amplifikation, og hvert DNA-molekyle svarer til et UMI. Under analysen af bioinformatik kan den derfor skelne DNA-skabeloner fra forskellige kilder, skelne mellem, hvilke er falsk-positive mutationer forårsaget af tilfældige fejl i processen med PCR-amplifikation og sekventering, og som faktisk udføres af patienter, for at filtrere baggrundsstøjen, realisere den nøjagtige påvisning af lavfrekvente og ekstremt lavfrekvente mutationer af DNA-mutationer og ekstremt lavfrekvente molekyler, der udføres. Det er meget udbredt i lavfrekvent mutationsdetektion, især inden for tumorforskning.

Fig. 4 Skematisk diagram af UMI-adapter strukturen af Illumina platformen
4.2 Komplet adaptere
Komplette adaptere, et nødvendigt produkt til et PCR-frit bibliotek, indeholder alle de sekvenser, der kræves til sekventeringer, såsom P5, P7, RdS1 og RdS2 i Illumina-platformen, også indekssekvenser og UMI-sekvenser i henhold til kravene til sekventering. Med de komplette adaptere kan den sekventeres direkte uden at introducere andre adaptere gennem PCR. Så komplette adaptere kan bruges til at bygge et PCR-frit bibliotek. De PCR-frie biblioteker kan reducere PCR-amplifikationsbias, fejlrate og sekvensduplikation, hvilket øger dækningen af nogle regioner med høj GC eller høj AT, som er meget brugt i populationsgenomforskning.
Et komplet adapterprodukt til Illumina-platformen leveret af Yeasen (Cat#13519ES/13520ES)>>
Et komplet adapterprodukt til MGI platform leveret af Yeasen(Cat#13360ES/13361ES)>>
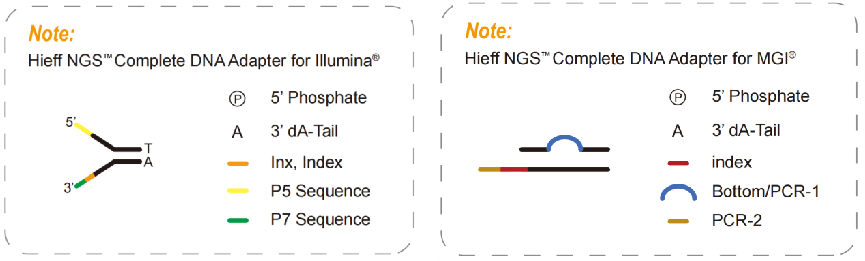
Fig. 5 Komplet adapterdiagram
4.3 Ufuldstændige adaptere
Ufuldstændige adaptere skal introducere andre sekvenser ved PCR efter adapterligeringen for at danne en komplet adapter. De er kendetegnet ved høj forbindelseseffektivitet og høj effektiv bibliotekshastighed. PCR-processen er en berigelseseffekt for hele biblioteket for at sikre koncentrationen af det effektive bibliotek og kan også introducere dobbeltendede indekser og UMI-sekvenser.
4.4 Tn5 adaptere
Tn5-adaptere forbinder en del af adaptorsekvensen til begge ender af DNA-fragmenterne gennem restriktionsendonukleaseaktiviteten af Tn5. De laver fragmentering og adapterligering udført samtidigt for at spare tid og prøver. Til sidst indføres resten af linkersekvensen, indekset, UMI og andre sekvenser ved PCR for at danne et komplet bibliotek. Det kan bruges til at bygge et Cut&tag-bibliotek.
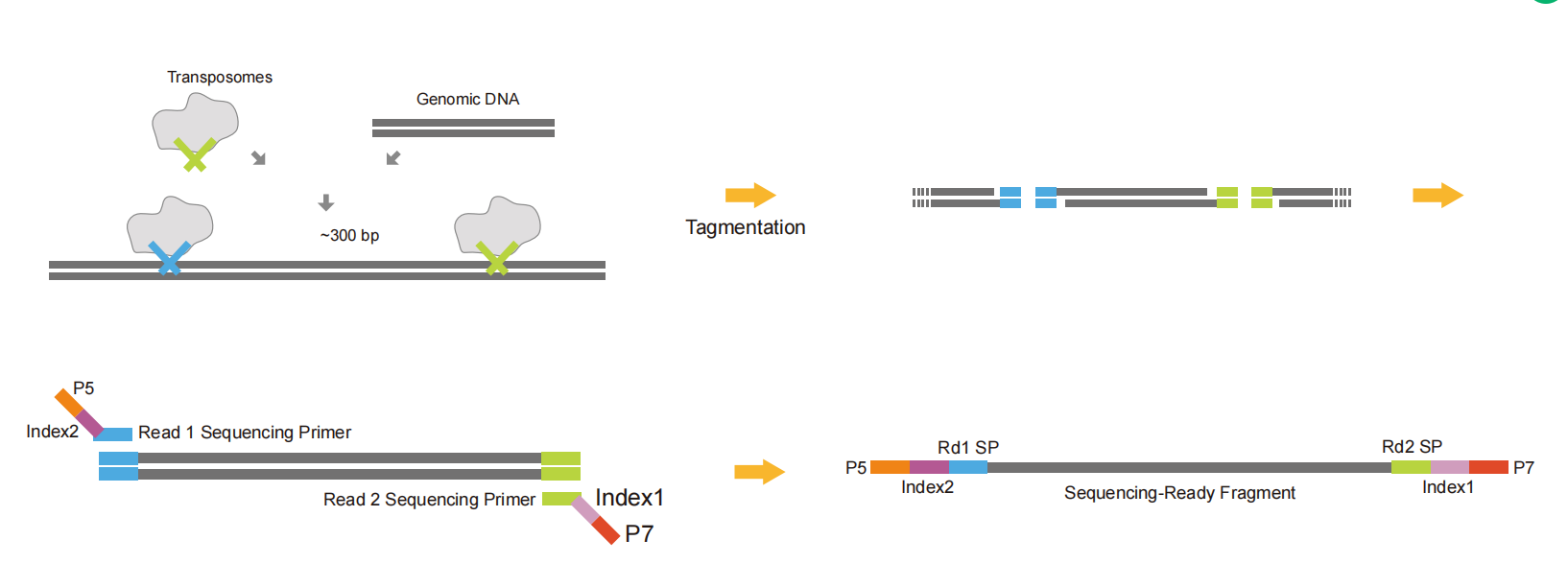
Fig.6 Skematisk diagram af Tn5-adapterbibliotekets konstruktion
5. Hvordan vælger du de rigtige NGS-adaptere til dine sekventeringsplatforme?
I øjeblikket er der to mainstream sekventeringsplatforme, inklusive Illumina og MGI. Yeasen, en komplet løsning til NGS-udbyderen, har udviklet flere NGS-adaptere, der passer til Illumina- eller MGI-platforme.
Med hensyn til Illumina-platformen indeholder Illumina NGS-adaptere leveret af Yeasen tre typer, herunder UDI, CDI og et enkelt indeks. Med hensyn til MGI-platformen har MGI NGS-adaptere, der tilbydes af Yeasen, to typer, der involverer Dual UMI -UDB og Single Index.Vi har angivet produktinformationen i følgende tabel, inklusive typer af adaptere, tilgængelige størrelser og koncentration af henholdsvis adapter og primer.
Komplet og UDI NGS adaptere behøver ikke at bekymre sig om koblingsproblemer, velegnet til kunder, der ønsker let at bruge; CDI NGS-adaptere har færre rør og lille størrelse, hvilket er velegnet til kunder, der ønsker at opbevare og bære let. PCR-fri kræver brug af komplette NGS-adaptere.
Til Illumina
| InTube | Hieff NGS® DNA Lib Prep 384 CDI Primer til Illumina, sæt 1 (8*12, 96 indeks) | 12412ES |
| Hieff NGS® DNA Lib Prep 384 CDI Primer til Illumina, sæt 2 (8*12, 96 indeks) | 12413ES | |
| Hieff NGS® RNA Lib Prep 384 CDI Primer til Illumina, sæt 1 (96 indeks) | 12414ES | |
| Hieff NGS® RNA Lib Prep 384 CDI Primer til Illumina, sæt 1 (96 indeks) | 12415ES | |
| I plade | Hieff NGS® Stubby UDI Primer Kit til Illumina (1-384 Index) Sæt 1-4 | 12407ES |
| Hieff NGS® Stubby UDI Primer Kit til Illumina Sæt 1(96 brønds plade, 1-96 Indeks) Sæt 1 | 12327ES | |
| Hieff NGS® Stubby UDI Primer Kit til Illumina Sæt 2(96 brønds plade, 97-192 Indeks) Sæt 2 | 12328ES | |
| Hieff NGS® Stubby UDI Primer Kit til Illumina Sæt 3(96 brønds plade, 193-288 Indeks) Sæt 3 | 12329ES | |
| Hieff NGS® Stubby UDI Primer Kit til Illumina Sæt 4(96 brønds plade, 289-384 Indeks) Sæt 4 | 12330ES |
Til MGI
| Hieff NGS™ Dual UMI UDB Adapter Kit til MGI, Set1/Set2 | 96 slags indeks | |
| Hieff NGS™ Komplet adaptersæt til MGI, Sæt1/Sæt2/Sæt3(Forespørg) | 13360ES | 8 slags indeks, 41-48 |
| 13361ES | 16 slags indeks, 57-72 | |
| 13362ES | 96 slags indeks, 1-96 |
Angående læsning
Nøgleenzymer involveret i NGS bibliotekskonstruktion
Hvor meget ved du om NGS-relateret teknologi?
Forskellige typer af magnetiske perler i NGS: DNA\RNA\mRNA magnetiske perler
NGS bibliotek kvantificering: Hurtig og nøjagtig Qubit eller Præcis qPCR?Alt nødvendigt!
