Endonucléases de restriction de la série Yeasen FuniCut™, digestion en 5 minutes avec tampon universel
Le clonage moléculaire est utilisé dans presque tous les laboratoires. Les endonucléases de restriction sont un élément crucial des expériences de clonage moléculaire, mais les expériences avec les endonucléases de restriction posent souvent divers problèmes. Les problèmes courants sont les suivants :
- Il existe tellement de types d’endonucléases de restriction qu’il est difficile de choisir.
- Mauvais clivage, clivage aléatoire ou clivage enzymatique incomplet.
- Le rythme de digestion est lent ; cela peut prendre une heure ou même toute la nuit.
- De plus, les expériences de digestion enzymatique multiple doivent également choisir une variété de digestion enzymatique bTo
Pour vous permettre de mieux comprendre les endonucléases de restriction, nous allons brièvement expliquer ce que sont les endonucléases de restriction et comment les différentes endonucléases sont classées en réponse à la première question. La série Yeasen FuniCut™ d'endonucléases de restriction rapides peut facilement résoudre les deuxième à quatrième problèmes grâce à la modulation de la contrainte et à une procédure améliorée !
1. Qu’est-ce qu’une endonucléase de restriction ?
2. Restrictions relatives à la nomenclature des endonucléases
3. Restrictions sur la classification des endonucléases
4. Endonucléases de restriction rapides Yeasen FuniCut™
1. Qu’est-ce qu’une endonucléase de restriction ?
Les endonucléases de restriction sont une classe d'enzymes capables de reconnaître des séquences nucléotidiques spécifiques dans les molécules d'ADN double brin et de couper la liaison phosphodiester dans les chaînes d'ADN à des sites spécifiques.
Différentes endonucléases de restriction reconnaîtront différentes séquences d'ADN. Elles peuvent couper l'ADN à l'intérieur de la séquence de reconnaissance ou à un endroit non loin de la séquence de reconnaissance, ce qui donne divers produits, comme illustré dans la figure 1. BamHI forme des extrémités collantes, KpnI forme des extrémités 3'ticky et EcoRV forme des extrémités franches.
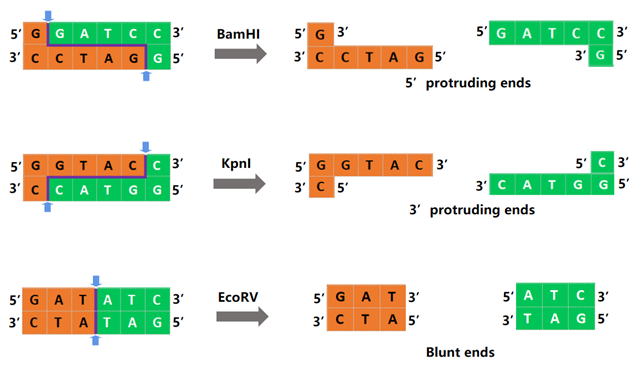
Figure 1. Diagramme schématique de la digestion par BamHI, KpnI, EcoRV
2. Restrictions relatives à la nomenclature des endonucléases
Le nom du genre, le nom de l'espèce, la souche bactérienne ou le sérotype et l'ordre de découverte sont les principaux facteurs utilisés pour nommer les endonucléases de restriction. Les directives de dénomination sont présentées dans le tableau 1 ci-dessous en utilisant BstEII comme exemple :
Tableau 1. Nomenclature des endonucléases restrictives
| Abréviation | Nom et prénom | Implication |
| B | Bacille | Première lettre du nom du genre |
| St | stearothermophilus | Les deux premières lettres du nom de l'espèce |
| E | ET | La première lettre du nom de la souche |
| II | Deuxième découverte | L'ordre trouvé dans ces bactéries |
3.Restrictions sur la classification des endonucléases
Selon la complexité de la structure, le mode d'action et la différence entre les cofacteurs, les enzymes de restriction peuvent être divisées en quatre catégories. Le tableau 2 suivant résume les caractéristiques et les enzymes typiques de chaque catégorie.
Tableau 2. Variations entre plusieurs endonucléases de restriction
| Classe d'endonucléases | Caractéristiques | Espèces d'enzymes typiques |
| Type I | 1. Reconnaissance et modification du clivage. 2. Il peut reconnaître des séquences d’ADN spécifiques, et le site de digestion est indéfiniment éloigné du site de reconnaissance, jusqu’à des milliers de bases. 3. L’action nécessite l’ATP. | EcoB, EcoK, etc. |
| Type II | 1. A uniquement pour fonction d'identifier la coupe. 2. La séquence de reconnaissance est souvent une courte séquence palindromique (environ 4 à 8 pb) et le site de digestion spécifique de l'endonucléase de restriction est généralement la séquence reconnue. 3. L’action nécessite Mg2+. 4. Le type d’enzymes de restriction le plus fréquemment utilisé dans le clonage moléculaire. | HindIII, NotI, etc. |
| Type III | 1. Reconnaissance et modification de la digestion. 2. À propos de séparez le site de reconnaissance du site de digestion. 3. L’action nécessite l’ATP. | HinfIII et al. |
| Type IV | 1. Coupe uniquement les séquences d’ADN méthylées. 2. Environ 30 pb séparent le site de reconnaissance du site de digestion. | McrA, McrBC, etc. |
Comme indiqué dans le tableau 3 ci-dessous, il peut également être divisé en trois groupes en fonction des similitudes et des différences entre les sites de reconnaissance et de digestion.
Tableau 3.Analyse comparative de l'isoschizomère, du néoschizomère et de l'isocaudarner
|
| Isoschizomère | Néoschizomère | Isocaudarner |
| séquence de reconnaissance | même | même | différent |
| sites de digestion | même | différent | même |
| représentant typique | AgeI et BshTI : tous deux reconnaissent et clivent 5′-A↓CCGGT-3′ | SmaI (5′-CCC↓GGG-3′) et XmaI (5′-C↓CCGGG-3′) | BamHI (5'-G↓GATCC-3') et BglII (5'-A↓GATCT-3') |
Étant donné que certaines endonucléases de restriction sur le marché nécessitent encore une digestion nocturne et que les produits sont sujets à une digestion incorrecte, la nucléase Yeasen Fast Restriction Endoa, un tampon universel, effectue une digestion précise en 5 minutes, permettant à vos expériences de digestion de ne plus avoir de problèmes !
4. Yeasen FuniCut™ Endonucléases de restriction rapides
Figure 2. Endonucléases de restriction rapides Yeasen FuniCut™
4.1 Caractéristiques
- Digestion rapide : la digestion peut être terminée en 5 à 15 minutes, ce qui peut être réduit de plus de 1 à 2 heures par rapport aux expériences de digestion conventionnelles.
- Tampon universel : La réaction de clivage multi-enzymatique est simplifiée par le fait que n'importe quelle combinaison d'endonucléases peut utiliser le même tampon.
- Effet de la digestion enzymatique : comparable à N* et approprié à son tampon.
- L'électrophorèse directe simplifie le processus en fournissant un tampon de couleur rouge et en permettant aux produits de digestion d'être délivrés directement pour l'électrophorèse.
- Digestion précise : même avec une digestion nocturne, il y a très peu d'activité des étoiles.
4.2 Présentation des performances
4.2.1 Dans FuniCut™ Buffer, la digestion simple, double et triple a lieu en 5 minutes.

Figure 3. Digestion simple, double et triple à l'aide du FuniCut™ Le tampon peut généralement être terminé en 5 minutes, par exemple, la triple digestion utilisant EcoRI+KpnI+SmaI.
4.2.2 L’impact de la digestion enzymatique est comparable à celui de N* et T*, et elle fonctionne bien avec leurs tampons.

Figure 4. Comparé à des produits N*, T* similaires, l'effet de digestion enzymatique Yeasen FuniCut™ BamHI est compatible avec les tampons de marque N*, T*.
4.2.3 Digestion nocturne avec une très faible activité stellaire
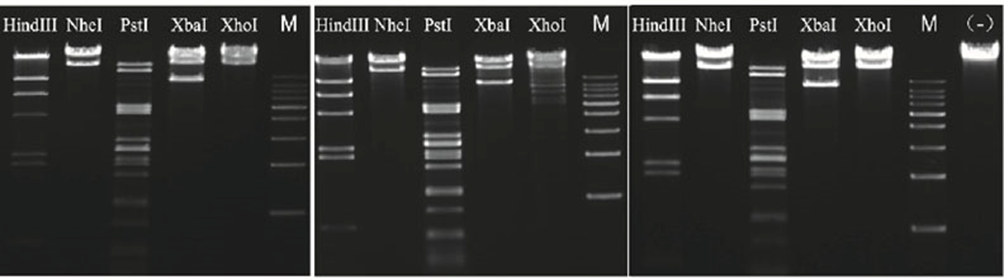
Figure 5. Utiliser les endonucléases rapides HindIII, NheI, PstI, XbaI et XhoI de Yeasen, Tbydance avec le protocole expérimental conseillé par chaque marque.Effectuer une digestion nocturne (16 h) et analyser les produits digérés par électrophorèse sur gel d'agarose.
4.2.4 Hautement redondant et capable de gérer un surplus de substrat.
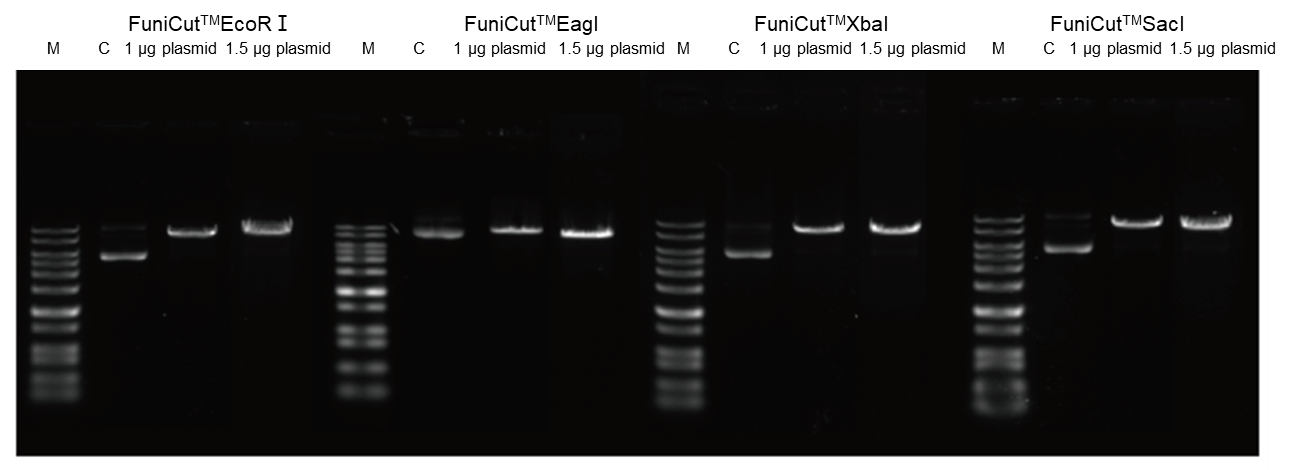
Figure 6. En utilisant les endonucléases de restriction rapides FuniCut™ EcoR I, Eag I, XbaI et Sac I, 1 μg et 1,5 μg de plasmide ont été utilisés comme substrats, respectivement, et digérés pendant 5 min, et les produits digérés ont été soumis à une électrophorèse sur gel d'agarose.
Tableau 4.Produits et compatibilité des tampons de réaction
| Nom du produit | Chat# | Séquence de reconnaissance | Température | Température de désactivation thermique |
| Tampon T* | Tampon N* | Tampon Ta* | Base protégée (pb) | Réponses |
| FuniCut™ AscI (renseigner) | 15001ES50 | GG/CGCGCC | 37 | 80 | 100 | 100 | 100 | 100 | 1 | 50T |
| FuniCut™ AvrII (renseigner) | 15002ES25 | C/CTAGG | 37 | 80 | 100 | 100 | 100 | 100 | 3 | 25T |
| FuniCut™ BamHI (renseigner) | 15003ES76 | G/GATCC | 37 | 80 | 100 | 100 | 100 | 100 | 3 | 500T |
| FuniCut™ BclI (renseigner) | 15004ES62 | T/GATCA | 37 | 80 | 100 | 100 | 100 | 100 | 3 | 125T |
| FuniCut™ BsaI (renseigner) | 15005ES50 | GGTCTC(1/5) | 37 | 80 | 100 | 100 | 100 | 100 | Pas | 50T |
| FuniCut™ BstEII (renseigner) | 15006ES60 | G/GTNACC | 37 | 80 | 100 | 75 | 100 | 75 | Pas | 100T |
| ClaI FuniCut™ (renseigner) | 15007ES50 | AT/CGAT | 37 | 80 | 100 | 100 | 100 | 100 | 4 | 50T |
| FuniCut™ DpnII (renseigner) | 15008ES50 | /GATC | 37 | 80 | 100 | 75 | 100 | 75 | Pas | 50T |
| FuniCut™ EagI (renseigner) | 15009ES25 | C/GGCCG | 37 | 80 | 100 | 100 | 100 | 100 | 2 | 25T |
| FuniCut™ EcoRI (renseigner) | 15010ES78 | G/AATTC | 37 | 80 | 100 | 100 | 100 | 100 | 5 | 600T |
| FuniCut™ EcoRV (renseigner) | 15011ES70 | GAT/ATC | 37 | 80 | 100 | 100 | 100 | 100 | 5 | 200T |
| FuniCut™ HindIII (renseigner) | 15012ES76 | A/AGCTT | 37 | 80 | 100 | 75 | 100 | 100 | 3 | 500T |
| FuniCut™ HpaI (renseigner) | 15013ES50 | GTT/AAC | 37 | 80 | 100 | 100 | 100 | 50 | 1 | 50T |
| FuniCut™ KpnI (renseigner) | 15015ES70 | GGTAC/C | 37 | 80 | 100 | 100 | 100 | 100 | 2 | 200T |
| FuniCut™ MluI (renseigner) | 15016ES60 | A/CGCGT | 37 | 80 | 100 | 75 | 100 | 100 | 3 | 100T |
| FuniCut™ NcoI (renseigner) | 15018ES30 | C/CATGG | 37 | 80 | 100 | 100 | 100 | 100 | 3 | 30T |
| FuniCut™ NdeI (renseigner) | 15019ES70 | CA/TATG | 37 | 80 | 100 | 100 | 100 | 100 | 3 | 200T |
| FuniCut™ NheI (renseigner) | 15020ES30 | G/CTAGC | 37 | 80 | 100 | 100 | 100 | 100 | 3 | 30T |
| FuniCut™ NotI (renseigner) | 15021ES50 | GC/GGCCGC | 37 | 80 | 100 | 100 | 100 | 100 | 2 | 50T |
| FuniCut™ PstI (renseigner) | 15022ES76 | CTGCA/G | 37 | Non | 100 | 100 | 100 | 100 | 2 | 500T |
| Sac FuniCut™ (renseigner) | 15023ES60 | GAGCT/C | 37 | 80 | 100 | 100 | 100 | 100 | 3 | 100T |
| FuniCut™ SalI (renseigner) | 15024ES70 | G/TCGAC | 37 | 80 | 100 | 100 | 100 | 100 | 3 | 200T |
| FuniCut™ SbfI (renseigner) | 15025ES25 | CCTGCA/GG | 37 | 80 | 100 | 100 | 100 | 100 | 2 | 25T |
| FuniCut™ Petit (renseigner) | 15027ES60 | CCC/GGG | 37 | 80 | 100 | 100 | 100 | 100 | 1 | 100T |
| Jeu FuniCut™ (renseigner) | 15028ES50 | A/CTAGT | 37 | 80 | 100 | 100 | 100 | 100 | 2 | 50T |
| FuniCut™ SphI (renseigner) | 15029ES50 | GCATG/C | 37 | 80 | 100 | 100 | 100 | 100 | 1 | 50T |
| FuniCut™ SspI (renseigner) | 15030ES56 | AAT/ATT | 37 | 80 | 100 | 50 | 100 | 100 | 2 | 60T |
| FuniCut™ StuI (renseigner) | 15031ES60 | AGG/CCT | 37 | 80 | 100 | 100 | 100 | 100 | 1 | 100T |
| FuniCut™ TaqI (renseigner) | 15032ES70 | T/CGA | 65 | Non | 100 | 100 | 100 | 100 | 4 | 200T |
| FuniCut™ XbaI (renseigner) | 15033ES76 | T/CTAGA | 37 | 80 | 100 | 50 | 100 | 100 | 2 | 500T |
| FuniCut™ XhoI (renseigner) | 15034ES76 | C/TCGAG | 37 | 80 | 100 | 100 | 100 | 100 | 4 | 500T |
| FuniCut™ FspI (renseigner) | 15036ES50 | TGC/GCA | 37 | 80 | 100 | 100 | 100 | 100 | 3 | 50T |
| FuniCut™ HinfI (renseigner) | 15038ES76 | G/ANTC | 37 | 80 | 100 | 100 | 100 | 50 | 2 | 500T |
| FuniCut™ ApaLI (renseigner) | 15039ES70 | G/TGCAC | 37 | 80 | 100 | 75 | 100 | 100 | 4 | 200T |
| FuniCut™ NruI (renseigner) | 15040ES50 | TCG/CGA | 37 | Non | 100 | 100 | 100 | 100 | 5 | 50T |
| FuniCut™ PacI (renseigner) | 15041ES25 | TTAAT/TAA | 37 | 80 | 100 | 100 | 100 | 100 | 2 | 25T |
| FuniCut™ PvuII (renseigner) | 15042ES70 | CAG/CTG | 37 | Non | 100 | 100 | 100 | 100 | 4 | 200T |
| FuniCut™ SacII (renseigner) | 15043ES50 | CCGC/GG | 37 | 80 | 100 | 100 | 100 | 100 | 4 | 50T |
| FuniCut™ NsiI (renseigner) | 15044ES25 | ATGCA/T | 37 | 80 | 100 | 100 | 100 | 100 | 3 | 25T |
| FuniCut™ Esp3I (BsmBI) (renseigner) | 15048ES30 | CGTCTC(1/5) | 37 | 80 | 100 | 100 | 100 | 100 | 5 | 30T |
| FuniCut™ BglII (renseigner) | 15049ES60 | A/GATCT | 37 | 80 | 100 | 100 | 100 | 50 | 2 | 100T |
| FuniCut™ BstBI (renseigner) | 15050ES60 | TT/CGAA | 37 | 80 | 100 | 100 | 100 | 100 | Pas | 100T |
| FuniCut™ MnlI (renseigner) | 15051ES50 | CCTC (7/6) | 37 | 80 | 100 | 100 | 100 | 100 | 2 | 50T |
CpG : Affecté par la méthylation du CpG.
EK : Affecté par la méthylation d'EcoKI.
EB : Affecté par la méthylation EcoBI.
Dcm : Affecté par la méthylation Dcm.
Barrage : Affecté par la méthylation du barrage.

