Avez-vous appris à créer une bibliothèque sans PCR ?
Dans le séquençage à haut débit, la construction de bibliothèques conventionnelles nécessite une amplification par PCR. D'une part, il s'agit d'amplifier les échantillons d'ADN traces et d'augmenter le rendement de la bibliothèque. D'autre part, il peut amplifier les signaux fluorescents, ce qui permet aux séquenceurs de capturer et d'identifier plus facilement les signaux fluorescents pour améliorer la précision du séquençage. Cependant, la PCR est comme une « épée à double tranchant ». Tout en résolvant le problème du faible volume d'échantillon initial et en amplifiant le signal fluorescent, elle introduit également des erreurs et des biais d'amplification et ne peut pas présenter parfaitement la séquence du génome « True Face ». De plus, la PCR polymérase présente un certain biais d'amplification. Certaines régions, en particulier les régions à GC élevé ou faible, la structure secondaire et d'autres régions, ont une faible efficacité d'amplification et sont difficiles à couvrir. Cela introduira également un grand nombre d'InDels erronés et entraînera un grand nombre de duplications, ce qui entraînera un gaspillage de volume de données ADN et augmentera le coût du séquençage. Par conséquent, l'introduction de la technologie sans PCR présente non seulement les avantages de la PCR, mais surmonte également les défauts de la PCR. Elle peut non seulement fournir des bibliothèques de haute qualité avec une sensibilité élevée et réaliser la détection d'échantillons traces, mais également avoir une grande précision et réduire la charge de travail de validation de suivi pour les études de variantes. Alors, qu'est-ce que la bibliothèque PCR-Free exactement et quels sont ses avantages ?
1. Qu'est-ce qu'une bibliothèque sans PCR ?
2. Quels sont les avantages de la technologie PCR-Free ?
3. Affichage des données de PCR-Free
4. FAQ
5. Informations sur le produit
6. Concernant la lecture
1. Qu'est-ce qu'une bibliothèque sans PCR ?
La technologie de séquençage de nouvelle génération est la technologie la plus couramment utilisée dans la recherche de séquençage à haut débit en biologie moléculaire moderne. Le séquençage traditionnel de première génération ne peut plus répondre pleinement aux besoins des chercheurs. Le reséquençage du génome d'organismes modèles et le séquençage du génome d'organismes non modèles nécessitent des coûts plus élevés. Une technologie de séquençage plus rapide et à faible débit a ainsi donné naissance à la technologie de séquençage de deuxième génération. Le principe de base de la technologie de séquençage de deuxième génération est le séquençage par synthèse, c'est-à-dire la détermination de la séquence d'ADN en capturant les informations de base marquées à l'extrémité nouvellement synthétisées. Le principe principal de la technologie de séquençage de deuxième génération pour le séquençage de l'ADN est de fragmenter d'abord l'ADN, de réparer l'extrémité de l'ADN fragmenté, puis d'ajouter des adaptateurs spécifiques des deux côtés, puis d'utiliser différentes méthodes pour générer des millions de PCR spatialement fixées. Pour les matrices clonales, les données de séquençage sont obtenues à l'aide d'une hybridation d'amorces et de réactions d'extension enzymatique pour imager les marqueurs fluorescents incorporés par chaque réaction d'extension.
Le séquençage de l'ADN par séquençage de nouvelle génération comprend principalement deux processus : la préparation de la bibliothèque et le séquençage sur machine. Pendant la préparation de la bibliothèque, les fragments génomiques interrompus de manière aléatoire sont généralement amplifiés par PCR standard. Cependant, pour certains modèles spéciaux, il existe des facteurs tels qu'une structure secondaire complexe ou une faible stabilité thermique, qui affectent la préférence d'amplification de la PCR modèle, de sorte que toutes les séquences génomiques ne peuvent pas être reflétées de manière égale dans la bibliothèque d'amplification PCR. En particulier pour certains modèles à teneur élevée en GC ou en AT, il est parfois difficile d'utiliser la méthode PCR pour amplifier en vue de la construction de la bibliothèque. Il n'y a aucune différence entre la bibliothèque sans PCR et la bibliothèque PCR conventionnelle lors du séquençage sur machine, sauf que la PCR n'est pas effectuée pendant le processus de construction de la bibliothèque. La bibliothèque sans PCR peut théoriquement améliorer la distribution des lectures de données et générer une couverture génomique plus uniforme.La bibliothèque sans PCR est un complément à la méthode de construction de bibliothèque. Puisqu'elle est directement préparée dans une bibliothèque embarquée sans amplification PCR, elle peut améliorer la couverture de certaines régions à GC élevé ou à AT élevé, réduisant ainsi les bases d'erreur introduites par la PCR, le biais de données et la répétition de séquences.
Les étapes principales de la construction d'une bibliothèque NGS conventionnelle sont la fragmentation du génome, la réparation des extrémités, l'ajout d'adaptateurs, la PCR et l'amplification du signal avant le séquençage. Alors, qu'en est-il de la construction d'une bibliothèque sans PCR ? Comme son nom l'indique, il s'agit d'un processus de construction de bibliothèque qui ne nécessite pas de PCR. Les étapes principales de la construction d'une bibliothèque sont la fragmentation du génome, la modification des extrémités, l'ajout de lieurs et le contrôle qualité de la bibliothèque. Mais en fait, cela ne peut être considéré comme sans PCR que dans le processus de construction de la bibliothèque. Le véritable sans PCR devrait être un processus allant de la construction de la bibliothèque au séquençage sans amplification de la bibliothèque (comme la technologie de séquençage à molécule unique) ou une technologie de séquençage sans accumulation d'erreurs d'amplification PCR (comme le DNBseq de MGI).

Figure 1. Organigramme de la construction d'une bibliothèque conventionnelle et de la construction d'une bibliothèque sans PCR
2. Quels sont les avantages de la technologie PCR-Free ?
Dans le processus de construction de bibliothèque de routine, l'enzyme d'amplification peut introduire des erreurs. Par amplification cyclique, ces erreurs seront accumulées et amplifiées, réduisant la fidélité de la réplication de la séquence d'ADN. De plus, l'ADN polymérase présente également un certain biais d'amplification, en particulier pour les régions présentant de grandes différences de contenu en GC ou de structures secondaires, ce qui entraîne une faible efficacité d'amplification et une mauvaise uniformité de la couverture ; tout en introduisant des InDels erronés, elle entraînera également un grand nombre de duplications, ce qui entraîne un gaspillage de volume de données ADN et augmente les coûts de séquençage. La technologie sans PCR peut bien éviter les problèmes mentionnés ci-dessus introduits par l'amplification PCR dans la construction de bibliothèque conventionnelle. Dans le processus de construction et de séquençage de la bibliothèque, s'il existe un processus d'amplification PCR, il aura un impact plus important sur l'uniformité de la couverture du génome. Pour les modèles d'ADN, certains ont des structures secondaires complexes et certains ont de grandes différences de stabilité thermique. Ces facteurs affecteront l'efficacité de l'amplification PCR. Par conséquent, dans le processus d'amplification par PCR, il n'est pas possible de garantir que tous les fragments génomiques puissent obtenir la même efficacité d'amplification, mais il existe un biais d'amplification évident, tel qu'une faible couverture dans les régions à GC élevé ou à GC faible du génome. Cependant, il n'y a pas de PCR dans l'ensemble du processus de séquençage sans PCR. Par rapport à la construction d'une bibliothèque PCR, la couverture des régions à GC élevé et des régions de répétition AT/TA du génome est améliorée. Les avantages spécifiques sont les suivants.
2.1 Processus de préparation de la bibliothèque simplifié, permettant de gagner du temps
La préparation de bibliothèque sans PCR élimine le besoin d'amplification PCR et d'autres étapes, simplifiant ainsi le processus de préparation de la bibliothèque et faisant gagner du temps.
2.2 Réduction des coûts de préparation de la bibliothèque
Dans le processus de préparation de bibliothèque classique, des enzymes coûteuses et de haute fidélité sont utilisées pour l'amplification par PCR afin de garantir l'authenticité de la séquence. En même temps, PCR-Free élimine l'étape d'amplification par PCR, réduisant ainsi les coûts de préparation de la bibliothèque.
2.3 Réduire le biais d’amplification
Les enzymes d'amplification de l'ADN présentent certains biais d'amplification, en particulier pour les modèles présentant des différences significatives dans la teneur en GC ou la structure secondaire. Par conséquent, PCR-Free peut éviter efficacement le biais d'amplification causé par la polymérase.
2.4 Pas de duplication PCR
PCR-Free peut réduire les lectures en double et augmenter les taux de mappage unique et l'utilisation efficace des données.
2.5 Réduction des erreurs
L'amplification PCR est plus susceptible d'introduire des erreurs de réplication d'insertion et de suppression, ce qui entraîne un taux d'erreur plus élevé de séquençage Indel, tandis que la PCR sans PCR peut éviter efficacement la génération et l'accumulation de telles erreurs.
3. Affichage des données de PCR-Free
Kit de préparation de bibliothèque d'ADN sans PCR Hieff NGS™ Ultima Pro V2 (Cat# 12196ES) a un meilleur taux de conversion de bibliothèque que les produits concurrents.
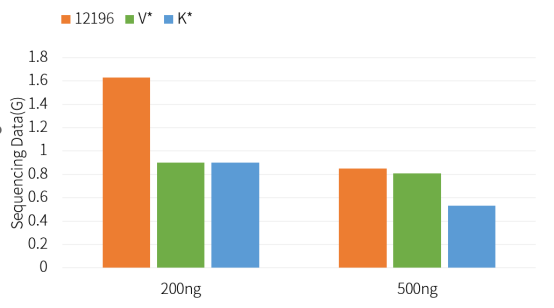
Figure 2. Quantité de données de séquençage hors machine pour la construction d'une bibliothèque sans PCR
4. FAQ
Q : Quels types d’échantillons conviennent aux bibliothèques sans PCR ?
R : Certains échantillons avec des structures secondaires complexes, une GC élevée et une préférence pour la PCR peuvent choisir une bibliothèque sans PCR à construire.
Q : Quelle est la taille générale des fragments de la bibliothèque sans PCR ?
R : La bibliothèque sans PCR elle-même n'a pas d'exigences particulières concernant la taille des fragments. La bibliothèque de produits PCR est construite en fonction de la taille du produit PCR. Pour la bibliothèque génomique, il est recommandé d'avoir une taille d'environ 350 pb, afin que la qualité des données de la bibliothèque soit relativement bonne.
5. Informations sur le produit
Le produit que Yeasen peut fournir est présenté dans le tableau 1.
Tableau 1. Informations sur le produit
| Produit Nom | N° de cat. | Taille |
| Kit de préparation de bibliothèque d'ADN sans PCR Hieff NGS™ Ultima Pro V2 | 12196ES24/96 | 24/96T |
6. Concernant la lecture
La solution pour la préparation de la bibliothèque NGS à partir d'échantillons d'ADN
Que savez-vous de la technologie liée au NGS ?
