Kit di sintesi di RNA ad alto rendimento T7: sintesi di RNA efficiente per la ricerca
Kit di sintesi di RNA ad alta resa T7 ottimizza il sistema di reazione di trascrizione. Il kit può sintetizzare l'RNA a singolo filamento in modo efficiente utilizzando la RNA polimerasi T7, il DNA lineare a doppio filamento con la sequenza del promotore T7 come stampo e gli NTP come substrato per controllare la sequenza del DNA a valle del promotore. Durante la trascrizione, i nucleotidi modificati possono essere aggiunti al substrato per preparare l'RNA marcato con biotina o colorante.
1. Introduzione del kit di sintesi di RNA ad alto rendimento T7
2. Principi di in vitro trascrizione
3. Vantaggi del kit di sintesi di RNA ad alta resa Yeasen T7
4. Prestazioni del prodotto
5. Come utilizzare questo kit
6. Domande frequenti
7. Informazioni per l'ordinazione
1. Introduzione del kit di sintesi di RNA ad alto rendimento T7
Il kit di sintesi di RNA ad alto rendimento T7 può sintetizzare sia trascrizioni lunghe che trascrizioni corte, e l'RNA può essere prodotto a 100-200 μg con 1 μg di input di modello di DNA. L'RNA sintetizzato può essere utilizzato per varie applicazioni downstream, come la ricerca sulla struttura e la funzione dell'RNA, la protezione RNasi, l'ibridazione della sonda, l'interferenza dell'RNA, la microiniezione e in vitro traduzione.
2. Principi di in vitro trascrizione
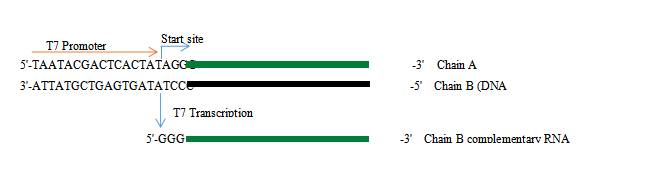
Figura 1. In vitro processo di trascrizione
3. Vantaggi del kit di sintesi di RNA ad alta resa Yeasen T7
Rese estremamente elevate: fino a 200 µg in 2 ore
Versatile: adatto per trascrizioni di RNA da 20 nt a 10000 nt
Usi multipli: genera RNA non etichettato, etichettato o tappato
4. Prestazioni del prodotto
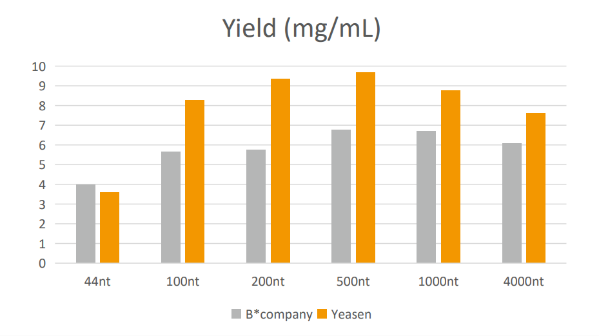
Figura 2. Confronto della resa IVT
Nota: tutti i reagenti di reazione provengono da Kit di sintesi dell'RNA T7 (Yeasen#10623 o società B*). Le rese della reazione IVT di Yeasen e della società B* sono mostrate sopra.
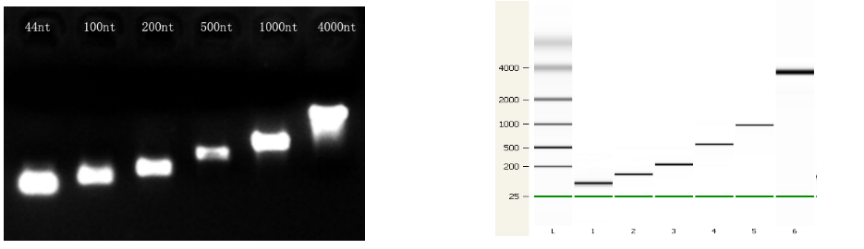
Figura 3. La qualità dei prodotti trascrizionali per diverse lunghezze
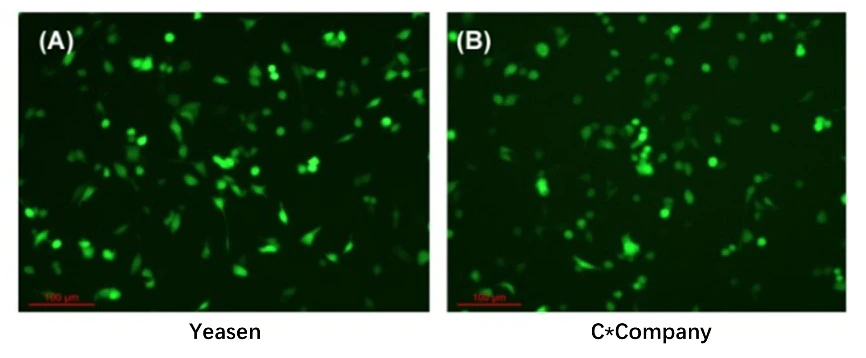
Figura 4. Il livello di espressione dell'mRNA trascritto nelle cellule HEK-293
Nota: l'mRNA è ricoperto dall'enzima Vaccinia Capping (Yeasen#10614/10615)
5. Come utilizzare questo kit
5.1 Scongelamento dei reagenti
Centrifugare brevemente la miscela di RNA polimerasi T7 e riporre sul ghiaccio. Scongelare il tampone di trascrizione 10× e i ribonucleotidi (ATP, CTP, GTP, UTP), mescolare e centrifugare sul fondo della provetta, posizionare il tampone di trascrizione 10× a temperatura ambiente e disporre quattro tipi di ribonucleotidi sul ghiaccio.
5.2 Preparare il sistema di reazione di trascrizione secondo la seguente tabella
Tabella 1.Metodo di preparazione del sistema di reazione di trascrizione
| Componenti | Volume(μL) | Concentrazione finale |
| H senza RNasi2Lo | Fino a 20 | - |
| 10×tampone di trascrizione | 2 | 1× |
| CTP / GTP / ATP / UTP (100 mM ciascuno) | 2 ciascuno | 10 mM ciascuno |
| Modello di DNA | 1 µg | - |
| Miscela di RNA polimerasi T7 | 2 | - |
Nota:
a) La reazione viene preparata a temperatura ambiente. Poiché il tampone di trascrizione 10× contiene spermidina, una concentrazione di spermidina troppo elevata a basse temperature causerà la precipitazione del modello di DNA.
b) Trascrizione breve (<100 nt), è possibile utilizzare un modello da 2 µg, il tempo di trascrizione aumenta a 4-8 ore.
c) Per trascrizioni lunghe (>1000 nt), si raccomanda l'uso di modelli plasmidici linearizzati per la trascrizione.
d) Eseguire la reazione nella macchina PCR con il coperchio caldo aperto per evitare che la soluzione di reazione evapori per lungo tempo.
e) Il prodotto di reazione può avere un precipitato bianco. Questo è pirofosfato di magnesio formato da pirofosfato libero e ioni di magnesio durante la reazione, che non influenzerà gli esperimenti successivi. Puoi aggiungere EDTA per rimuoverlo. Se sei preoccupato che l'aggiunta di EDTA influisca sugli esperimenti successivi, il surnatante può anche essere recuperato tramite centrifugazione.
f) I reagenti e i contenitori non devono essere contaminati da RNasi.
5.3 Incubare a 37°C per 2 ore
Miscelare la soluzione di reazione di cui sopra, centrifugare brevemente sul fondo della provetta e incubare a 37°C per 2 ore. Se la lunghezza del trascritto è inferiore a 100 nt, aumentare il tempo di reazione a 4-8 ore.
5.4 Trattamento con DNasi I (facoltativo)
Una volta completata la reazione, aggiungere 2 μL di DNasi I (RNasi-free) a ciascuna provetta e incubare a 37°C per 15 minuti per rimuovere il DNA stampo.
6. Domande frequenti
6.1 La resa della reazione IVT è bassa
La qualità del modello è strettamente correlata alla resa. Se la resa del gruppo sperimentale è significativamente inferiore a quella del gruppo di controllo positivo, le possibili ragioni possono essere.
① I modelli contengono componenti che inibiscono la reazione IVT.
② C'è qualcosa di sbagliato nei modelli.
6.2 Suggerimenti
① Ripulire il modello.
② Confermare la concentrazione e l'integrità dei modelli.
③ Prolunga il tempo di reazione.
④ Aumenta l'input del modello.
⑤Provare altre RNA polimerasi e i promotori corrispondenti.
6.3 La resa delle trascrizioni brevi è bassa
Se i trascritti target sono più corti di 100 nt, estendere il tempo di reazione a 4-8 ore o aumentare l'input di modelli a 2 ug in un sistema di reazione da 20 μL.
6.4 Ci sono trascrizioni più lunghe inaspettate
Se il risultato dell'elettroforesi su gel mostra che ci sono trascrizioni più lunghe inaspettate, le possibili ragioni possono essere:
① I modelli plasmidici potrebbero non essere completamente linearizzati.
②I modelli hanno estremità coesive con sporgenze di 3'.
③I trascritti hanno una struttura secondaria che non è completamente denaturata.
6.5 Suggerimenti
①Controllare se i modelli plasmidici sono completamente linearizzati. Se necessario, eseguire nuovamente la linearizzazione del plasmide.
②Selezionare enzimi di restrizione adatti per linearizzare i modelli plasmidici ed evitare di produrre estremità coesive con sporgenze 3'. È possibile utilizzare il frammento di Klenow o la DNA polimerasi T4 per produrre un'estremità smussata, se necessario.
③Utilizzare gel denaturato per rilevare le trascrizioni.
6.6 Ci sono trascrizioni inaspettatamente più brevi
Se il risultato dell'elettroforesi su gel mostra che ci sono trascrizioni più corte inaspettate, le possibili ragioni possono essere:
①Negli stampi è presente una sequenza analoga alla sequenza di terminazione della RNA polimerasi T7.
②Il contenuto GC dei modelli è elevato.
6.7 Suggerimenti
①Ridurre la temperatura di reazione (ad esempio 30℃), ma tenere presente che a volte la riduzione della temperatura di reazione può ridurre le rese.
②Provare altre RNA polimerasi per catalizzare la reazione IVT.
③Se il contenuto di GC dei modelli è elevato, impostare la temperatura di reazione IVT a 42℃ o aggiungere SSB nel sistema di reazione IVT per aumentare le rese e la lunghezza delle trascrizioni.
6.8 Si verifica una sbavatura dei trascritti durante l'elettroforesi su gel
Se si verifica una sbavatura dei trascritti durante l'elettroforesi su gel, le possibili cause possono essere:
①Si verifica contaminazione da RNasi durante l'operazione sperimentale.
②I modelli di DNA sono contaminati dalle RNasi.
6.9 Suggerimenti
①Assicurarsi che tutti i reagenti siano formulati con H2O priva di RNasi. Utilizzare puntali per pipette e provette Eppendorf prive di RNasi e indossare guanti e maschere in lattice monouso durante le operazioni sperimentali.
②Ripurificare i modelli di DNA.
7. Informazioni per l'ordinazione
Di seguito sono riportati i prodotti rappresentativi offerti da Yeasen. Sono disponibili altre dimensioni. I nostri prodotti sono altamente ottimizzati per funzionare in concerto, per aiutare a garantire prestazioni e riproducibilità superiori.
Possiamo anche fornire servizi personalizzati. Se sei interessato a un prodotto che non è mostrato, contattaci e lavoreremo con te per soddisfare le tue esigenze.
Tabella 2.Informazioni sul prodotto
Per quanto riguarda la lettura:
Reagenti di grado GMP per la sintesi in vitro di mRNA
Materie prime per la sintesi in vitro di mRNA di grado GMP di Yeasen Biotechnology
