NGS アダプターの選択方法は?
次世代シーケンシング ライブラリの重要な部分である NGS アダプタは、テストされた DNA フラグメントとフロー セル (シーケンシング チップ) を接続する役割を果たします。接続の効率は、ライブラリの品質と収量を決定する重要な要素です。では、NGS アダプタとは何でしょうか。NGS アダプタの一般的なタイプは何でしょうか。また、シーケンシング プラットフォームに適した NGS アダプタを選択するにはどうすればよいでしょうか。
1. NGS アダプターとは何ですか?
2. NGS インデックスを選択する際に考慮すべき要素は何ですか?
3. 一般的なインデックスの種類は何ですか?
4. NGS アダプターの一般的なタイプは何ですか?
- UMI アダプター
- 完全なアダプター
- 不完全なアダプタ
- Tn5 アダプター
5. シーケンス プラットフォームに適した NGS アダプターを選択するにはどうすればよいでしょうか?
6. 読書について
1. NGS アダプターとは何ですか?
NGS アダプターは、シーケンシングにおける一連のアダプターであり、既知の配列を持つ短いヌクレオチド配列です。これは、ターゲット核酸フラグメントの両端に連結されます。シーケンシング中、フローセル上の既知の配列とハイブリダイズして、ライブラリをチップに結合することでシーケンシングを開始します。では、NGS アダプターの構造はどのようなものでしょうか。
Illumina プラットフォームを例にとると、NGS アダプターは次の 3 つの部分に分けられます。
P5 および P7: フローセル上で P5 および P7 末端と結合した配列により、シーケンシング チップ上のライブラリが固定され、ブリッジ PCR によるクラスター反応が容易になります。
Rd1 SP および Rd2 SP (Read1/Read2 シーケンス プライマー): シーケンスの読み取りが開始される位置を示すシーケンス プライマーの結合領域。
インデックス (バーコードとも呼ばれる): 混合ライブラリのシーケンスで異なるサンプルを区別するために使用される既知の合成シーケンス。
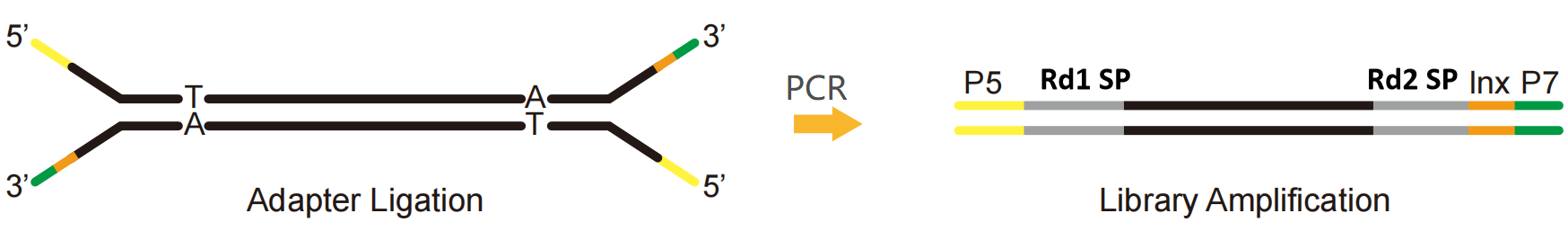
図1 イルミナプラットフォームシングルインデックスライブラリ

図2 MGIプラットフォームシングルエンドインデックスライブラリ
シーケンス処理能力の向上により、複数のサンプルを同時にシーケンスできるようになりました。そのため、多様なサンプルを区別する方法が特に重要になります。前述のように、NGS アダプターのインデックス シーケンスは、次世代シーケンス (NGS) でさまざまなサンプルを区別するために使用されます。インデックスを選択する際に考慮すべき要素は何でしょうか? 引き続きお読みください...
2. インデックスを選択する際に考慮すべき要素は何ですか?
インデックスは一般的に 6nt-18nt の長さで、インデックスの数に応じてシングルインデックスとダブルインデックスに分けられます。ダブルインデックスは、テストするフラグメントの両端にあります。インデックスの組み合わせを選択するときは、塩基バランスと蛍光バランスを考慮する必要があります。
ベースバランスとは、単一のインデックス内のベースバランスではなく、複数のインデックス間のバランスを指します。ベースタイプとベース分布の両方から考慮する必要があります。組み合わせの原則は、同じインデックスグループ内の4つのベースA / T / C / Gを含める必要があり、これら4つのベースの割合は近く、それぞれ約25%を占めることです。
蛍光信号バランスとは、塩基バランスが保証できない場合に蛍光信号のバランスを確保するための選択を指します。イルミナ プラットフォームの 4 チャネル シーケンサーでは、dG/dT は緑色蛍光でラベル付けされ、dC/dA は赤色蛍光でラベル付けされます。シーケンス中、シーケンスを成功させるには、各サイクルに緑色蛍光信号と赤色蛍光信号の両方が存在する必要があります。したがって、インデックスを選択するときは、緑色信号と赤色信号のバランスを考慮する必要があります。
3. 一般的なインデックスの種類は何ですか?
一般的なデュアル インデックスには、通常、ユニーク デュアル インデックス (UDI)、ユニーク デュアル バーコード (UDB)、および複合デュアル インデックス (CDI) が含まれ、これによりインデックス ホッピングと誤った割り当てが大幅に削減されます。
UDI&UDB: 両端のインデックスは 1 対 1 で対応し、グループで設計されており、両端で相互検証できます。
CDI: 両端のインデックスを特定の要件に従って組み合わせて、両端インデックス ライブラリを形成できます。
スループットと増幅効率を向上させ、シーケンシングコストを削減するために、イルミナはNovaseqやその他の高スループットシーケンサー向けにアレイフローセル(PFCT)と専用増幅(ExAmp)クラスタリング技術を導入しましたが、サンプルラベルの不一致現象とインデックスホッピングが意図せず増幅されてしまいました。

図3 イルミナの異なる機器モデルは、非パターン化フローセルまたはパターン化フローセルを採用している
HiSeq3000/4000、HiSeq Xシリーズ、NovaSeqなどのシーケンシングプラットフォームで浮き彫りになったインデックスホッピング問題を補うために、イルミナはライブラリの両端にインデックスを配置する戦略を提案しました。これにより、双方向検証を実行し、不一致のアダプターを排除できます。両端に固有のインデックスを使用すると、インデックスエラー割り当て率が0.01%に減少します。従来のインデックス順列グループの組み合わせ方法と比較して、インデックスホッピングは2桁減少します。
PCR フリー ライブラリの構築では、シングルエンド インデックス アダプターが利用できます。ラベルの不一致は主にシーケンス エラーによって発生します。全体的に、ラベルの不一致率は低いです (平均 0.0004%、最大 0.001%)。ただし、ターゲット キャプチャ ライブラリの構築では、複数のステップでラベルの不一致が発生するため、クロストークの問題が増幅され、UDI/UDB/CDI アダプターが通常使用されます。
4.NGS アダプターの一般的なタイプは何ですか?
シーケンシング技術の発展に伴い、シングルインデックス/ダブルインデックスアダプター(セクション3で説明)、UMIアダプター、トランスポザーゼアダプター、完全/不完全アダプターなど、さまざまなアプリケーションシナリオに適したアダプターの種類が増えています。この部分では、これらのアダプターを体系的に分類して、アダプター選択の基礎を提供します。
4.1 UMIアダプタ
ユニーク分子識別子(UMI)アダプターは、低頻度変異の検出と絶対定量化のためのエッジツールです。 UMIは、既知の配列を持つランダム合成配列です。 完全にランダムなヌクレオチド鎖、部分的に縮退したヌクレオチド鎖、または固定ヌクレオチド鎖として設計できます。 長さは通常10nt(シングルエンドUMI)または5〜8nt(ダブルエンドUMI)です。 その機能は、増幅前にDNA断片の状態を固定することであり、各DNA分子はUMIに対応しています。 したがって、バイオインフォマティクスの分析中に、異なるソースからのDNAテンプレートを区別し、PCR増幅および配列決定のプロセスでランダムエラーによって引き起こされる偽陽性変異と、実際に患者が持っている変異を区別して、バックグラウンドノイズを除去し、低頻度および極低頻度の変異を正確に検出し、さまざまなDNA分子の絶対定量化を実行できます。 特に腫瘍研究の分野で、低頻度変異の検出に広く使用されています。

図4 イルミナプラットフォームのUMIアダプタ構造の概略図
4.2 完全なアダプタ
PCRフリーライブラリに必要な製品である完全アダプターには、イルミナプラットフォームのP5、P7、RdS1、RdS2など、シーケンスに必要なすべての配列が含まれており、シーケンスの要件に応じたインデックス配列とUMI配列も含まれています。完全アダプターを使用すると、PCRを介して他のアダプターを導入することなく、直接シーケンスできます。そのため、完全アダプターを使用してPCRフリーライブラリを構築できます。PCRフリーライブラリは、PCR増幅バイアス、エラー率、および配列の重複を減らし、集団ゲノム研究で広く使用されている一部の高GCまたは高AT領域のカバレッジを拡大できます。
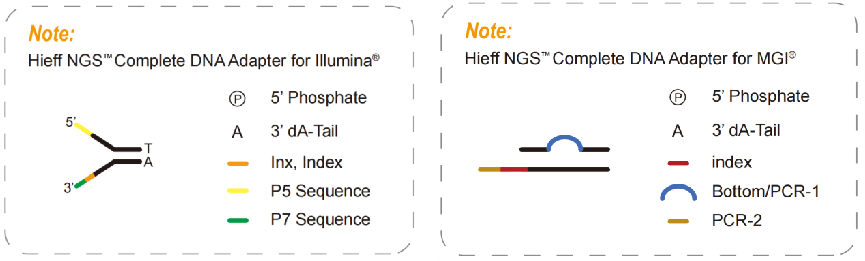
図5 完全なアダプタ図
4.3 不完全なアダプタ
不完全なアダプターは、アダプター連結後にPCRで他の配列を導入して完全なアダプターを形成する必要があります。これらは、高い連結効率と高い有効ライブラリ率を特徴としています。PCRプロセスは、完全なライブラリに対する濃縮効果であり、有効ライブラリの濃度を確保し、両端インデックスとUMI配列を導入することもできます。
4.4 Tn5アダプタ
Tn5 アダプターは、Tn5 の制限酵素活性により、アダプター配列の一部を DNA 断片の両端に接続します。断片化とアダプター連結を同時に実行することで、時間とサンプルを節約します。最後に、残りのリンカー配列、インデックス、UMI などの配列を PCR で導入して、完全なライブラリを形成します。これを使用して、Cut&tag ライブラリを構築できます。
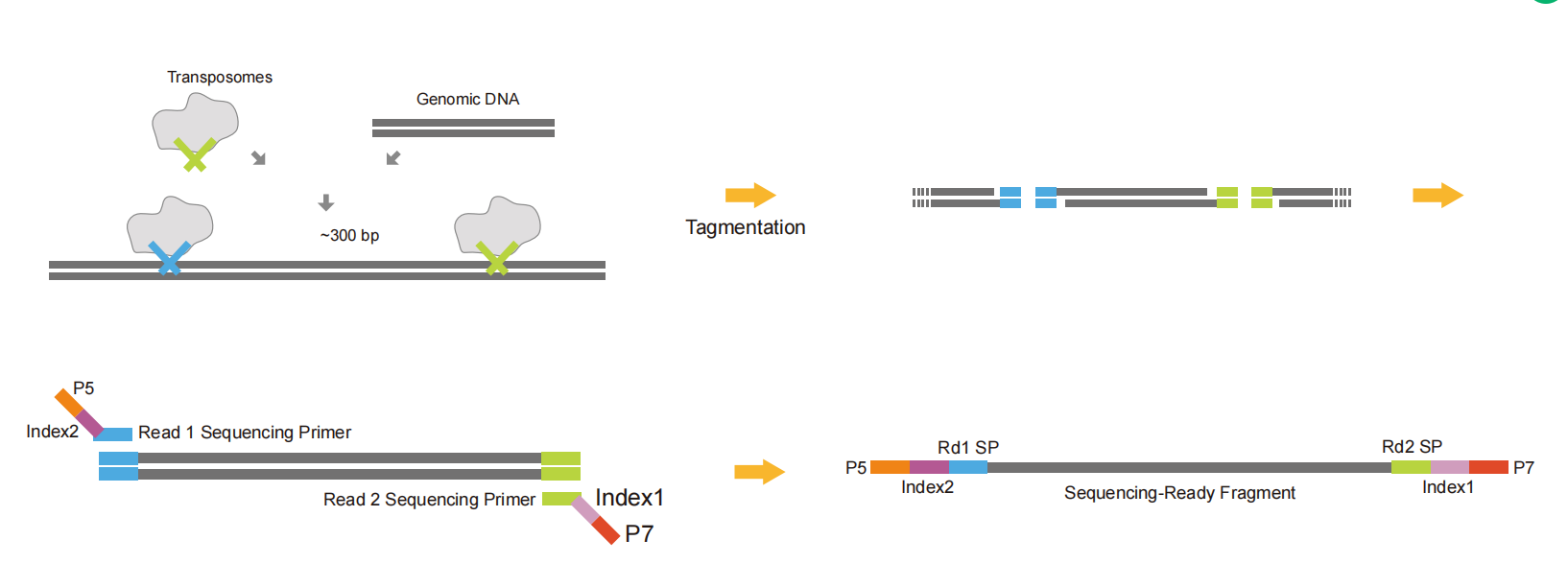
図6 Tn5アダプターライブラリ構築の概略図
5. シーケンス プラットフォームに適した NGS アダプターを選択するにはどうすればよいでしょうか?
現在、主流のシーケンシング プラットフォームは Illumina と MGI の 2 つです。NGS プロバイダー向けの完全なソリューションを提供する
Illumina プラットフォームに関しては、
完全および UDI NGS アダプターはカップリングの問題を心配する必要がないため、使いやすさを求めるお客様に適しています。CDI NGS アダプターはチューブが少なく、サイズが小さいため、簡単に保管および持ち運びしたいお客様に適しています。PCR フリーには、完全な NGS アダプターを使用する必要があります。
イルミナの場合
| インチューブ | Hieff NGS ® D NA Lib Prep 384 CDI プライマー(Illumina 用)、セット 1(8*12、96 インデックス) | 12412ES |
| Hieff NGS ® D NA Lib Prep 384 CDI プライマー(Illumina 用)、セット 2(8*12、96 インデックス) | 12413ES | |
| Hieff NGS® RNA Lib Prep 384 CDI プライマー(Illumina 用)、セット 1(96 インデックス) | 12414ES | |
| Hieff NGS® RNA Lib Prep 384 CDI プライマー(Illumina 用)、セット 1(96 インデックス) | 12415ES | |
| プレート内 | Hieff NGS® Stubby UDI プライマーキット (Illumina 用) (1-384 インデックス) セット 1-4 | 12407ES |
|
Hieff NGS® Stubby UDIプライマーキット(Illumina用) セット1 ( 96ウェルプレート、1-96インデックス)セット1 |
12327ES | |
|
Hieff NGS® Stubby UDIプライマーキット(Illumina用) セット2 ( 96ウェルプレート、97-192インデックス)セット2 |
12328ES | |
|
Hieff NGS® Stubby UDIプライマーキット(Illumina用) セット3 ( 96ウェルプレート、193-288インデックス)セット3 |
12329ES | |
|
Hieff NGS® Stubby UDIプライマーキット(Illumina用) セット4 ( 96ウェルプレート、289-384インデックス)セット4 |
12330ES |
MGIの場合
| Hieff NGS™ デュアル UMI UDB アダプタ キット (MGI 用)、Set1/Set2 | 96種類のインデックス | |
| Hieff NGS™ MGI 用完全アダプターキット、Set1/Set2/Set3(お問い合わせ) | 13360ES | 8種類の索引、41-48 |
| 13361ES | 16種類の索引、57-72 | |
| 13362ES | 96種類のインデックス、1~96 |
読書について
NGSライブラリ構築に関与する主要な酵素
NGS関連の技術について、どれくらいご存知ですか?
NGSにおけるさまざまな種類の磁気ビーズ: DNA\RNA\mRNA磁気ビーズ
NGS ライブラリの定量化: 迅速かつ正確な Qubit か、精密な qPCR か? すべて必要です!
