설명
Hieff NGS TM OnePot Pro DNA Library Prep Kit V3는 Illumina 및 MGI 시퀀싱 플랫폼을 위해 특별히 개발 및 설계된 차세대 효소 단편화 기반 라이브러리 준비 키트입니다 . 이 제품은 기존 라이브러리 구축 방법과 비교하여 고품질 단편화 효소를 사용하여 번거로운 초음파 프로세스를 제거합니다. 단편화 및 말단 복구 모듈을 하나로 결합하여 작업을 간소화합니다. 또한 라이게이션 모듈의 효소와 완충액은 미리 혼합되어 라이브러리 구축 시간과 비용을 크게 줄입니다. 따라서 자동화된 라이브러리 구축에 더 적합합니다. 이 라이브러리 준비 키트는 라이브러리 변환율이 뛰어나 모든 일반적인 동물, 식물, 미생물 등의 샘플과 FFPE 샘플에 적용할 수 있습니다. 이전 세대 라이브러리 구축 키트를 기반으로 이 제품은 이전 버전보다 단편화, 말단 복구, dA-테일링 및 어댑터 라이게이션에서 더 높은 효율성을 보입니다. 고충실도 효소는 증폭의 균일성과 충실도를 크게 향상시킵니다.
명세서
|
제품번호 |
12194ES08 / 12194ES24 / 12194ES96 |
|
크기 |
8톤 / 24톤 / 96톤 |
구성 요소
|
구성품 번호 |
이름 |
1 2194 ES08 |
1 2194 ES24 |
1 2194 ES96 |
|
12194-A |
스미어라 제TM 버퍼 3.0 |
80 μL |
240 μL |
960 μL |
|
12194-B |
스미어라 제TM 효소 3.0 |
80 μL |
240 μL |
960 μL |
|
12194-C |
라이게이션 레디 믹스 |
200 μL |
600 μL |
3 × 800 μL |
|
12194-D |
2× Ultima HF 증폭 믹스 |
200 μL |
600 μL |
3 × 800 μL |
[ 참고 ]: 키트 구성 요소는 전체 어댑터가 사용된 경우 Illumina 및 MGI 시퀀싱 플랫폼과 모두 호환됩니다. Hieff NGS TM 프라이머 믹스(Yeasen Cat#12190 또는 Cat#12191)가 필요합니다.
저장
본 제품은 -25~-15℃에서 1년간 보관하시기 바랍니다.
노트
1. 운영에 관하여
1. 안전을 위해 실험실 가운과 일회용 장갑 을 착용하고 작업해 주시기 바랍니다.
2. 실온에서 성분을 해동합니다. 해동 후, vortexing하여 완전히 섞고, 튜브를 잠깐 돌린 후 나중에 사용하기 위해 얼음 위에 두십시오.
3. 각 단계의 반응 용액을 제조할 때는 피펫을 사용하여 잘 섞거나 가볍게 흔드는 것이 좋습니다. 격렬하게 흔드는 경우 라이브러리 출력이 감소할 수 있습니다.
4. 교차 오염을 피하기 위해 여과된 피펫 팁을 사용하는 것이 좋습니다. 다른 샘플을 처리할 때는 피펫 팁을 교체해야 합니다.
5. 부적절한 조작은 에어로졸 오염을 일으킬 가능성이 매우 높으며, 결과의 정확도에 영향을 미칩니다. PCR 반응 혼합 영역과 PCR 생성물 정제 분석 영역의 의무적인 물리적 격리가 권장됩니다. 라이브러리 구축을 위한 특수 피펫과 같은 장비를 갖추고 있습니다. 0.5% 차아염소산나트륨 또는 10% 표백제로 표면을 닦아 각 영역에 대한 정기적인 세척을 수행합니다.
6. 본 제품은 연구용으로만 사용됩니다.
2. DNA 단편화
1. 이 키트는 100 pg - 1000 ng의 입력 DNA와 호환됩니다. A260/A280 = 1.8-2.0인 고품질 입력 DNA를 사용하는 것이 좋습니다.
2. 금속 킬레이트제와 같은 고농도의 염이 입력 DNA에 도입되면 다음 실험에 영향을 미칠 수 있습니다. DNA 샘플을 ddH 2 O에서 용출하여 단편화하는 것이 좋습니다.
3. 표준 DNA 샘플의 단편화 시간은 표 6을 참조하십시오. 이 키트는 단편화 편향이 낮고 다양한 GC 조성의 DNA 샘플에 대해 균일한 GC 적용 범위를 제공합니다. 실험 요구 사항에 따라 단편화 시간을 조정하십시오.
4. 정확한 분열을 위해 얼음 위에서 반응을 준비하세요.
3. 어댑터 결찰
1. Illumina 또는 MGI 긴 어댑터(바코드 어댑터) 키트와 짧은 어댑터 키트는 고객이 실험 요구 사항에 따라 선택할 수 있도록 제공됩니다.
2. 고품질의 상용 어댑터를 선택하는 것이 좋습니다. 자체 제작 어댑터를 선택하는 경우 NGS 프라이머 합성 경험이 있는 회사에 맡기고 엄격한 오염 관리가 필요하다는 점을 언급하세요. 또한 교차 오염을 방지하기 위해 클린 벤치에서 DNA 어닐링 용액을 준비하고 매번 한 가지 유형의 어댑터만 작동하는 것이 좋습니다.
3. 어댑터는 얼음 위나 4°C에서 해동하세요. 실온에서 작동할 경우, 어댑터가 변성되는 것을 방지하기 위해 실험실 온도는 25°C를 초과해서는 안 됩니다.
4. 어댑터의 품질과 농도는 라이게이션 효율과 라이브러리 수율에 직접적인 영향을 미칩니다. 어댑터 농도가 너무 높으면 어댑터 다이머 형성에 유리하고 어댑터가 너무 적으면 라이게이션 속도와 라이브러리 수율이 감소합니다. 어댑터를 사용할 때 입력 DNA 양에 따른 TE 버퍼로 희석합니다. 표 1-2는 Illumina 또는 MGI 시퀀싱 플랫폼에 이 키트를 사용하여 다양한 양의 입력 DNA에 대한 기존 및 UMI 어댑터에 권장되는 희석 방법을 나열합니다.
표 1 다양한 입력 DNA에 대한 권장 Illumina 어댑터 양
|
DNA 입력 |
C 기존 어댑터 희석 비율 |
집중 |
UMI 어댑터 희석 비율 |
집중 |
|
< 1 ng |
7.5배 |
2μM |
15배 |
1μM (마이크로미터) |
|
1ng ~ 10ng |
3-폴드 |
5μM(마이크로 미터) |
3-폴드 |
5μM(마이크로미터) |
|
10ng ~ 200ng |
1.5배 |
10μM(마이크로 미터) |
2-폴드 |
7.5μM |
|
> 200 ng |
0-폴드 |
15μM(마이크로미터) |
0-폴드 |
15μM(마이크로미터) |
표 2 다양한 입력 DNA에 대한 권장 MGI 어댑터 양
|
DNA 입력 |
C 기존 어댑터 희석 비율 |
집중 |
UMI 어댑터 희석 비율 |
집중 |
|
< 1 ng |
5배 |
2μM |
10배 |
1μM (마이크로미터) |
|
1ng ~ 10ng |
2-폴드 |
5μM(마이크로 미터) |
2-폴드 |
5μM(마이크로미터) |
|
10ng ~ 200ng |
0-폴드 |
10μM(마이크로 미터) |
1.25 배 |
8μM입니다 |
|
> 200 ng |
0-폴드 |
10μM(마이크로 미터) |
0-폴드 |
10μM(마이크로 미터) |
4. 비드 기반 DNA 정화 및 크기 선택
1. DNA 크기 선택은 말단 수리/dA-테일링 전, 어댑터 연결 후 또는 증폭 후에 수행할 수 있습니다.
2. 입력 DNA 양이 50ng 이상인 경우 어댑터 라이게이션 직후에 크기 선택을 수행하는 것이 좋습니다. 그렇지 않은 경우 증폭 후 크기 선택을 수행하세요.
3. 라이게이션 인핸서는 PEG의 고농도를 함유하고 있어 정확한 크기 선택에 상당한 영향을 미칠 수 있습니다. 따라서 어댑터 라이게이션 직후에 크기 선택을 수행해야 하는 경우 크기 선택 전에 비드 클린업 단계를 추가하는 것이 좋습니다. 크기 선택 단계는 엔드 리페어/dA-테일링 전이나 라이브러리 증폭 후에 수행하면 직접 수행할 수 있습니다.
4. 자기 비드는 사용 전 실온에서 평형을 맞춰야 합니다. 그렇지 않으면 수율이 감소하고 크기 선택 효과에 영향을 미칩니다.
5. 자석 비드는 사용 전에 소용돌이 치거나 피펫으로 잘 섞어야 합니다.
6. 상층액을 옮길 때 구슬을 흡인하지 마십시오. 구슬이 미량이라도 다음 반응에 영향을 미칠 수 있습니다.
7. 80% 에탄올은 신선하게 제조해야 합니다. 그렇지 않으면 회수 효율에 영향을 미칩니다.
8. 정확한 크기 선택을 위해 100 μL 이상의 용량으로 시작하는 것이 좋습니다. 그보다 적으면 초순수로 용량을 100 μL까지 늘리는 것이 좋습니다.
9. 자석 비드는 제품을 용출하기 전에 실온에서 건조해야 합니다. 건조가 충분하지 않으면 에탄올 잔류물이 후속 반응에 영향을 미치기 쉽고, 건조가 너무 심하면 자석 비드가 갈라지고 정제 수율이 감소합니다. 일반적으로 실온에서 3~5분 동안 건조하면 비드가 완전히 건조되기에 충분합니다.
10. 필요한 경우, 0.1× TE 완충액에서 용출된 정제 또는 크기 선택된 DNA 샘플은 4°C에서 1-2주 동안 또는 -20°C에서 1개월 동안 보관할 수 있습니다.
5. 라이브러리 증폭
1. 라이브러리 증폭을 수행할지 여부는 DNA 입력량, 어댑터 유형, 시퀀싱 데이터 응용 프로그램 등에 따라 달라집니다. 부분 어댑터를 사용하는 경우 증폭 단계가 필요합니다. 전체 길이 어댑터를 사용하는 경우 입력 DNA가 200ng 미만이면 증폭을 수행하는 것이 좋습니다. 그렇지 않으면 증폭이 필요하지 않습니다.
2. 증폭 사이클 수는 엄격하게 제어해야 합니다. 증폭이 충분하지 않으면 라이브러리 수율이 낮아질 수 있습니다. 과증폭은 편향, 오류, 중복된 판독 및 키메라 생성물을 증가시킬 수 있습니다. 표 3은 라이브러리 수율 1μg을 목표로 하는 권장 사이클 수를 나열합니다.
표 3 라이브러리 수율 1,000ng을 생성하기 위한 권장 사이클 수
|
DNA 입력 |
1μg의 라이브러리 수율을 생성하는 데 필요한 사이클 수 |
|
1000~2000ng |
2 - 4 |
|
500 ng |
2 - 4 |
|
250 ng |
4 - 6 |
|
100 ng |
5 - 7 |
|
50 ng |
7 - 9 |
|
10 ng |
9 - 11 |
|
5 ng |
10 - 12 |
|
1 ng |
12 - 15 |
|
100페이지 |
16 - 18 |
메모
1. 표 3은 200bp 정도의 고품질 Input DNA 검사를 이용하여 얻은 루프 매개변수의 수를 보여줍니다. FFPE DNA의 품질은 매우 다양하며, DNA 품질이 좋지 않거나 라이브러리 길이가 긴 경우 충분한 라이브러리를 얻기 위해 사이클 수를 적절히 늘려야 합니다.
2. 라이브러리 구축 과정에서 크기 선택이 필요한 경우, 라이브러리 증폭에 더 높은 사이클 번호를 권장합니다. 그렇지 않은 경우, 더 낮은 사이클 번호를 권장합니다.
3. 불완전한 어댑터를 사용하는 경우, 완전한 어댑터를 형성하려면 최소 2사이클을 증폭해야 합니다.
6. 도서관 품질 분석
1. 구축된 라이브러리의 품질은 일반적으로 농도와 크기 분포를 측정하여 분석됩니다.
2. 라이브러리의 농도는 Qubit, PicoGreen 또는 qPCR과 같은 형광 기반 방법을 통해 측정할 수 있습니다.
3. NanoDrop과 같은 흡광도 기반 정량화 방법을 사용하는 것은 권장되지 않습니다.
4. 라이브러리 정량화에는 qPCR 방법을 사용하는 것이 좋습니다. Qubit 및 PicoGreen과 같은 형광 기반 방법은 불완전한 dsDNA 구조(어댑터가 없거나 어댑터로 연결된 끝 중 하나만 있는 삽입물)와 완전한 라이브러리를 구별할 수 없습니다. qPCR 방법은 어댑터로 양쪽 끝이 연결된 완전한 라이브러리(시퀀싱 가능한 라이브러리)만 증폭하고 측정하므로 로딩을 위한 보다 정확한 측정을 제공합니다.
5. 라이브러리의 크기 분포는 모세관 전기영동이나 미세유체역학의 원리를 기반으로 하는 Agilent Bioanalyzer 또는 기타 장치를 사용하여 분석할 수 있습니다.
7. 기타 자료
1. DNA 정제용 자석 비드: Hieff NGS TM DNA 선택 비드(Yeasen Cat#12601) 또는 AMPure ® XP 비드(A63880) 또는 이와 동등한 다른 제품.
2. 어댑터: Illumina용 전체 어댑터: Yeasen Cat#13519-13520; 384 Dual CDI Primers: Yeasen Cat#12412~Cat#12413; 384 Unique Dual Index(UDI) Primers: Yeasen Cat#12312~Cat#12315; UMI UDI 어댑터: Yeasen Cat#13370~Cat#13371; MGI용 전체 어댑터: Yeasen Cat#13360-13362. DNA Primer Mix : Cat#12190 또는 Cat#12191.
3. 라이브러리 품질 분석: Agilent 2100 Bioanalyzer DNA 1000 Chip/ High Sensitivity Chip 또는 이와 동등한 제품, 라이브러리 정량 시약.
4. 기타 재료: 무수 에탄올, 멸균 초순수, 저유지 피펫 팁, PCR 튜브, 자기 스탠드, 열 순환기 등
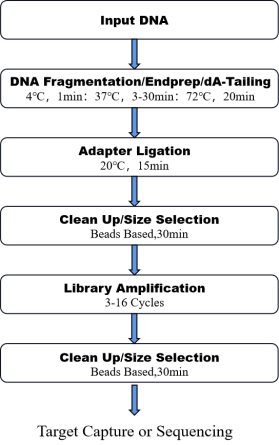
8. 작업 흐름
그림 1. OnePot Pro DNA Library Prep Kit 의 워크플로
그림
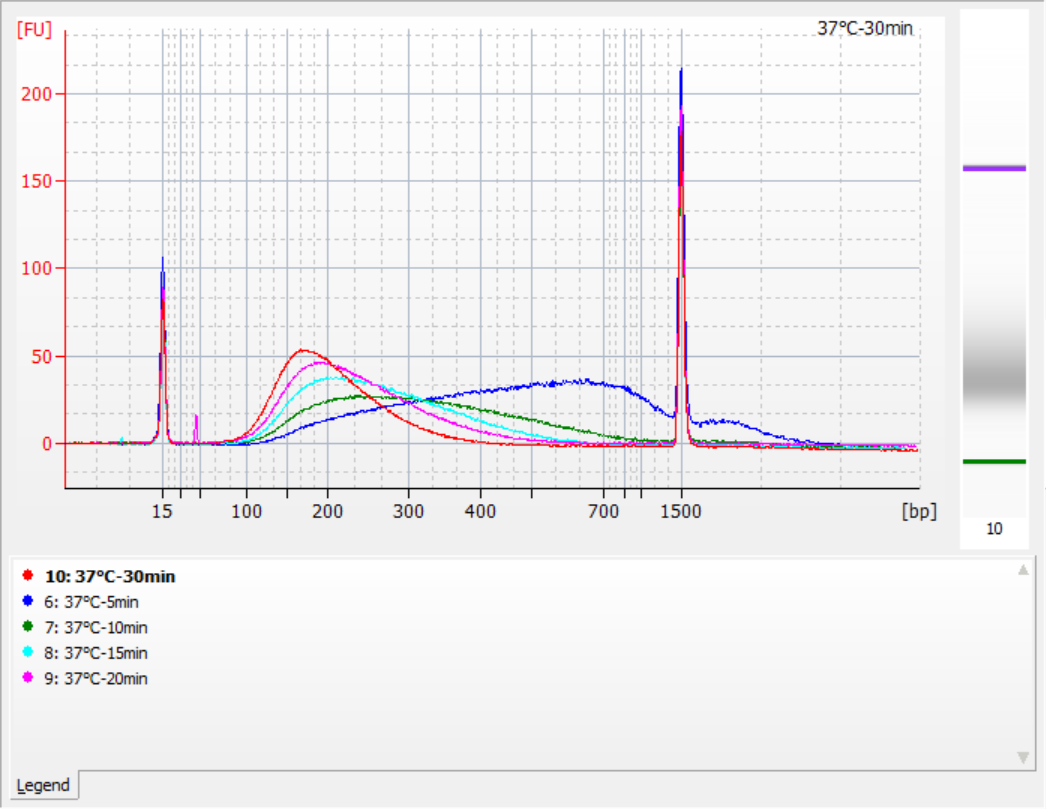
다양한 단편화 조건에서 얻은 삽입 단편의 크기
표준 gDNA 500ng을 템플릿으로 사용하여 이 키트로 라이브러리를 구성했습니다. 단편화 조건은 각각 32°C, 35°C 및 37°C에서 5, 10, 15, 20 및 30분 동안 효소 소화였습니다. 단편화된 생성물은 1.2x 자기 비드로 정제하고 ddH 2 O 21 μL로 용출했습니다. 농도는 Qubit을 사용하여 측정했으며 회수된 삽입 단편의 분포는 다음 그림에 나와 있습니다.
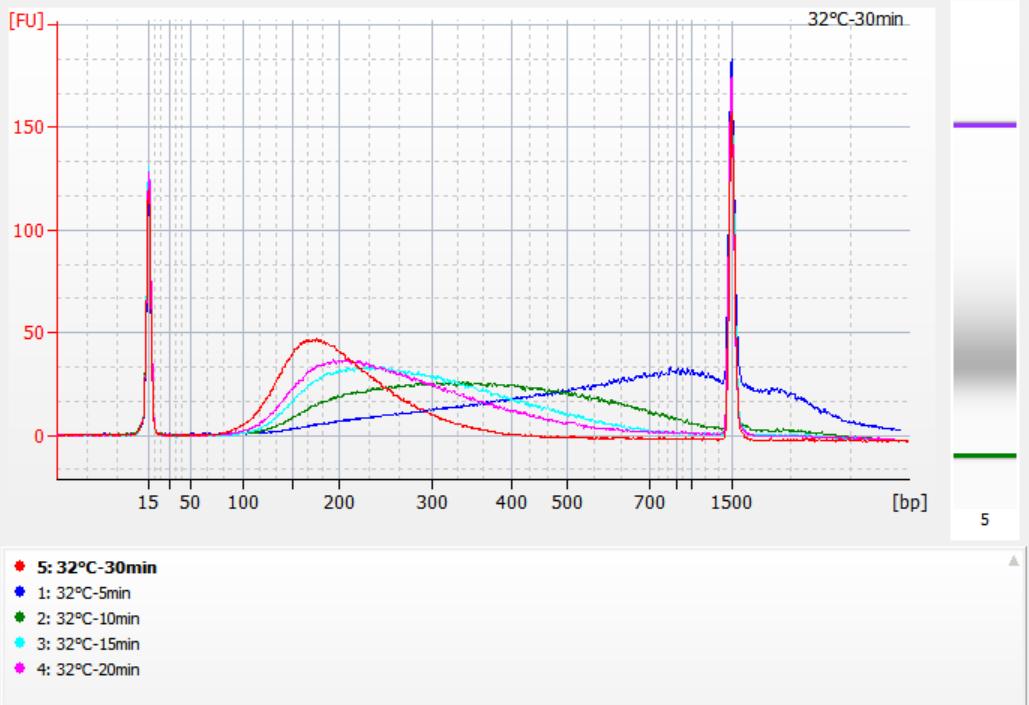 |
그림 2. 다양한 효소 소화 시간에 대한 32°C에서의 라이브러리 프로파일
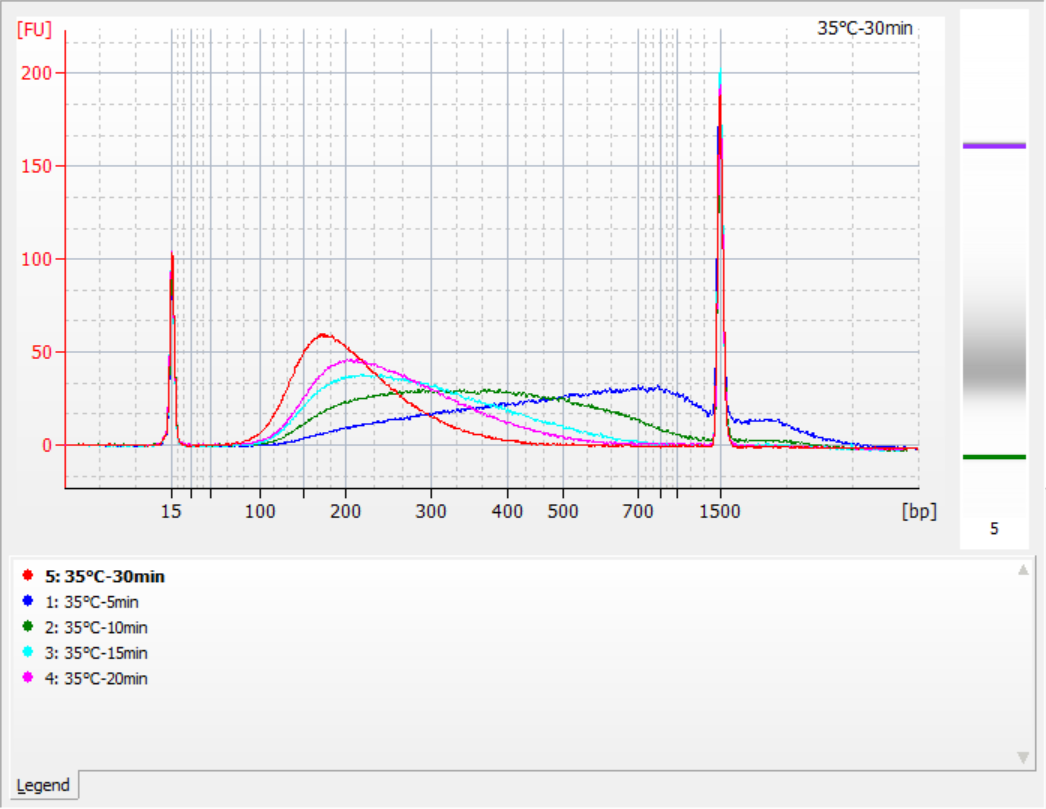 |
 |
결제 및 보안
귀하의 결제 정보는 안전하게 처리됩니다. 당사는 신용카드 정보를 저장하지 않으며 귀하의 신용카드 정보에 접근할 수 없습니다.
문의
당신은 또한 좋아할 수 있습니다
자주 묻는 질문
이 제품은 연구 목적으로만 사용되며 인간이나 동물의 치료 또는 진단용으로 의도되지 않았습니다. 제품과 콘텐츠는
특정 애플리케이션에는 추가적인 제3자 지적 재산권이 필요할 수 있습니다.
예슨은 윤리적 과학에 헌신하며, 우리의 연구가 안전과 윤리적 기준을 보장하는 동시에 중요한 문제를 해결해야 한다고 믿습니다.