설명
CHO 숙주 세포 DNA 잔류물 검출 키트는 다양한 생물학적 제품의 중간 시료, 반제품 및 완제품에 존재하는 CHO 숙주 세포 DNA 잔류물의 정량 분석에 사용됩니다.
이 키트는 Taqman 형광 프로브와 중합효소 연쇄 반응(PCR) 방법을 채택하는데, 이는 fg 수준의 최소 검출 한계를 가지고 있으며 잔류 CHO 세포 DNA를 특이적이고 빠르게 검출할 수 있습니다. 이 키트는 잔류 DNA 샘플 준비 키트(Cat# 18461ES)와 함께 사용해야 합니다.
제품 구성 요소
| 아니요. |
이름 |
41332 ES50 ( 50T ) |
41332 ES60 ( 100T ) |
|
41332-A |
CHO qPCR 믹스 |
0.75ml (100ml) |
1.5ml(1.5ml) |
|
41332-B |
CHO 프라이머&프로브 믹스 |
250 μL |
500 μL |
|
41332-C |
DNA 희석 완충액 |
2×1.8ml |
4×1.8ml |
|
41332-D |
CHO DNA 컨트롤(30ng/μL) |
25 μL |
50 μL |
배송 및 보관
1. 건조 얼음에 담아 배송하고 -20°C에서 2년간 보관
본 제품은 -25~-15℃에서 2년간 보관하시기 바랍니다.
41332-A와 41332-B는 모두 빛으로부터 보호하여 보관해야 합니다.
적용 가능한 계기 모델
다음을 포함하되 이에 국한되지 않습니다.
Bio-Rad: CFX96 광학 모듈.
Thermo Scientific: ABI 7500; ABI Quant Studio 5.
지침
- CHO DNA 표준 희석 및 표준 곡선 준비
CHO DNA Control은 키트 에 제공된 DNA 희석 완충액을 사용하여 농도구배 희석되었으며 희석액은
농도는 3 ng/μL, 300 pg/μL, 30 pg/μL, 3 pg/μL, 300 fg/μL, 30 fg/μL, 3 fg/μL입니다.
자세한 지침은 아래를 참조하세요.
- CHO DNA 대조군과 DNA 희석 완충액을 얼음 위에서 녹입니다. 완전히 녹인 후, 부드럽게 흔들어 섞고, 저속으로 10초 동안 원심분리합니다.
- Std0, Std1, Std2, Std3, Std4, Std5, Std6이라고 표시된 1.5 mL 튜브 7개를 꺼냅니다.
- 90 μL DNA 희석 완충액과 10 μL CHODNA 대조군을 Std0로 표시된 1.5 mL 마이크로퓨지 튜브에 넣고 3 ng/μL로 희석합니다. 혼합한 다음 10초 동안 원심분리합니다. 희석된 DNA 표준을 하위 포장하면 단기적으로(최대 3개월) -25~-15℃에서 보관할 수 있습니다. ** 반복적인 동결-해동은 피하십시오.
- 다른 튜브에 DNA 희석 완충액 90 μL를 첨가합니다 *** , 그다음 연속 희석을 위한 아래 절차를 따릅니다 **** .
|
튜브 |
희석 비율 |
표준 농도 |
|
1단계 |
10 μL Std0 + 90 μL DNA 희석 완충액 |
300pg/μL |
|
2단계 |
10 μL Std1 + 90 μL DNA 희석 완충액 |
30pg/μL |
|
3단계 |
10 μL Std2 + 90 μL DNA 희석 완충액 |
3pg/μL |
|
4학년 |
10 μL Std3 + 90 μL DNA 희석 완충액 |
300fg/μL |
|
표준5 |
10 μL Std4 + 90 μL DNA 희석 완충액 |
30fg/μL |
|
6학년 |
10 μL Std5 + 90 μL DNA 희석 완충액 |
3fg/μL |
표1 표준 기울기 희석
* 각 농도마다 3개의 복제 웰이 필요합니다. 검출 범위는 3 fg/μL~300pg/μL이며 이 범위는 확장될 수 있습니다.
** 반복적인 동결-해동 횟수를 줄이고 오염을 피하기 위해, DNA 대조군은 처음에는 -25~-15℃에서 일정량으로 보관하는 것이 좋습니다.
*** 해동 후, DNA 희석 완충액은 2-8°C에서 7일간 보관 가능합니다. 장기간 사용하지 않을 경우 -25~-15℃에서 보관하시기 바랍니다.
**** 템플릿이 완전히 혼합되었는지 확인하고, 각 그라데이션 희석마다 15초~1분 동안 혼합물을 가볍게 흔듭니다.
- 추출 회수 제어(ERC) 준비
필요에 따라 ERC의 CHO DNA 농도를 설정합니다(ERC 샘플은 예시로 30 pg CHO DNA로 준비되었습니다). 다음과 같습니다.
- 깨끗한 1.5mL 튜브에 검사 샘플 100 μL를 넣은 다음, 3pg/μL CHO DNA 표준(Std3) 10 μL를 넣고 잘 섞은 후 ERC로 표시합니다.
- 정제된 ERC 샘플을 준비하기 위해 시험 샘플과 함께 ERC 샘플의 DNA 추출을 수행합니다.
- 음성대조액(NCS) 제조
실험에서 음성 대조군을 설정합니다. 구체적인 작업 단계는 다음과 같습니다.
1) 깨끗한 1.5mL 튜브에 100 μL 샘플 매트릭스(또는 DNA 희석 완충액)를 넣은 다음 NCS로 표시합니다.
2) 정제된 NCS 샘플을 준비하기 위해 시험 샘플과 함께 NCS 샘플의 DNA 추출을 수행합니다.
- 템플릿 제어 없음(NTC) 준비
실험에서 템플릿 없는 제어를 설정합니다. 구체적인 작업 단계는 다음과 같습니다.
1) NTC는 샘플 전처리가 필요 없으며, 잔류 DNA의 qPCR 검출 단계에서 구성될 수 있습니다.
2) 각 튜브 또는 웰의 NTC 샘플은 20 μL 혼합물(즉, 15 μL CHO qPCR 혼합물 + 4 μL CHO 프라이머&프로브 혼합물 + 1 μL IC) + 10 μL DNA 희석 완충액입니다. 3개의 복제 웰을 구성하는 것이 좋습니다.
- PCR 반응 시스템
|
요소 |
용량(μL) |
|
CHO qPCR 믹스 * |
15 |
|
CHO 프라이머&프로브 믹스 |
5 |
|
DNA 템플릿 |
10 |
|
총 볼륨 ** |
30 |
Table2 반응계
* 반응 수로 총 PCR 반응 부피를 계산합니다: qPCR Mix = (반응 수+2) × (15+4+1) μL (두 반응 웰의 손실 포함). 실험에서는 각 샘플에 대해 3회 이상의 반복이 권장됩니다.
** 튜브를 캡핑하거나 플레이트를 밀봉한 후 반응 튜브 또는 플레이트를 저속으로 10초 동안 원심분리합니다. 5초 동안 충분히 흔들고 섞은 후 원심분리를 반복하여 뚜껑이나 벽에서 바닥까지 액체를 수집합니다. 작동 중에 거품이 생기지 않도록 합니다.
권장되는 플레이트 설정은 아래 표를 참조하세요.
|
1 |
2 |
3 |
4 |
5 |
6 |
7 |
8 |
9 |
10 |
11 |
12 |
|
|
에이 |
엔티씨씨 |
티에스1 |
티에스1 |
티에스1 |
표준 1 |
표준 1 |
표준 1 |
|||||
|
비 |
엔티씨씨 |
티에스2 |
티에스2 |
티에스2 |
2학년 |
2학년 |
2학년 |
|||||
|
기음 |
엔티씨씨 |
티에스3 |
티에스3 |
티에스3 |
3학년 |
3학년 |
3학년 |
|||||
|
디 |
4학년 |
4학년 |
4학년 |
|||||||||
|
이자형 |
NCS |
1. ERC1 |
1. ERC1 |
1. ERC1 |
표준 5 |
표준 5 |
표준 5 |
|||||
|
에프 |
NCS |
ERC2는 2019년 10월 28일에 출시될 예정입니다. |
ERC2는 2018년 10월 28일에 출시되었습니다. |
ERC2는 2019년 10월 28일에 출시될 예정입니다. |
6학년 |
6학년 |
6학년 |
|||||
|
G |
NCS |
ERC3 (에이알씨3) |
ERC3 (에이알씨3) |
ERC3 (에이알씨3) |
||||||||
|
시간 |
Table3 컴퓨터-온 레퍼런스 보드
플레이트 레이아웃에는 다음이 포함됩니다. 6 Std(6가지 표준 농도의 표준 곡선), 1 NTC(템플릿 제어 없음), 1 NCS(음성 제어 용액), 3 TS(테스트 샘플), 3 ERC(추출 회수 제어). 각 샘플에 대한 3개의 복제 웰.
- PCR 기기 설정 지침 (2단계 방법) (예: Thermo ABI 7500 qPCR 기기, 버전 2.0 소프트웨어)
다음 지침은 Thermo ABI 7500 qPCR 기기(소프트웨어 버전 2.0)에만 적용됩니다. 다른 기기를 사용하는 경우 해당 기기 가이드에서 설정 지침을 참조하세요.
1) 새로운 실험을 생성하고 절대 정량화 또는 사용자 정의 템플릿을 선택합니다.
2) "CHO-DNA"라는 이름의 검출 프로브 1개를 만들고, 리포터 형광체를 "FAM"으로, 퀀칭 형광체를 "없음"으로 선택합니다. 검출 프로브 1개를 더 만들고, "IC"라는 이름을 지정하고 리포터 형광체를 "CY5"로, 퀀칭 형광체를 "없음"으로 선택합니다. 기준 형광은 "ROX"입니다(기준 형광은 기기 모델 등에 따라 다를 수 있으며, 추가해야 하는지 여부를 선택합니다).
3) '샘플' 창에서 모든 샘플 정보를 차례로 추가합니다. 그런 다음 웰을 선택하고 대상과 샘플을 각각 선택합니다. CHO DNA 표준의 작업을 표준으로 설정하고 수량 열에 300000, 30000, 3000, 300, 30, 3(각 웰의 DNA 농도 단위는 fg/μL) 값을 할당하고 웰의 이름을 각각 Std 1, Std 2, Std 3, Std 4, Std 5, Std 6으로 지정합니다. NTC의 작업을 NTC로 설정합니다. NCS, TS, ERC를 Unknown으로 설정하고 위의 Plate 레이아웃에 따라 각각 이름을 지정합니다. 그런 다음 다음을 클릭합니다.
4) 증폭 프로그램을 설정합니다: 반응 용량을 30 μL로 설정합니다.
|
사이클 단계 |
온도(℃) |
시간 |
사이클 |
|
초기 변성 |
95℃ |
5분 |
1 |
|
변성 |
95℃ |
15초 |
40 |
|
어닐링/확장(형광수집) |
60℃ |
30초 |
표4 증폭 절차
- qPCR 결과 분석
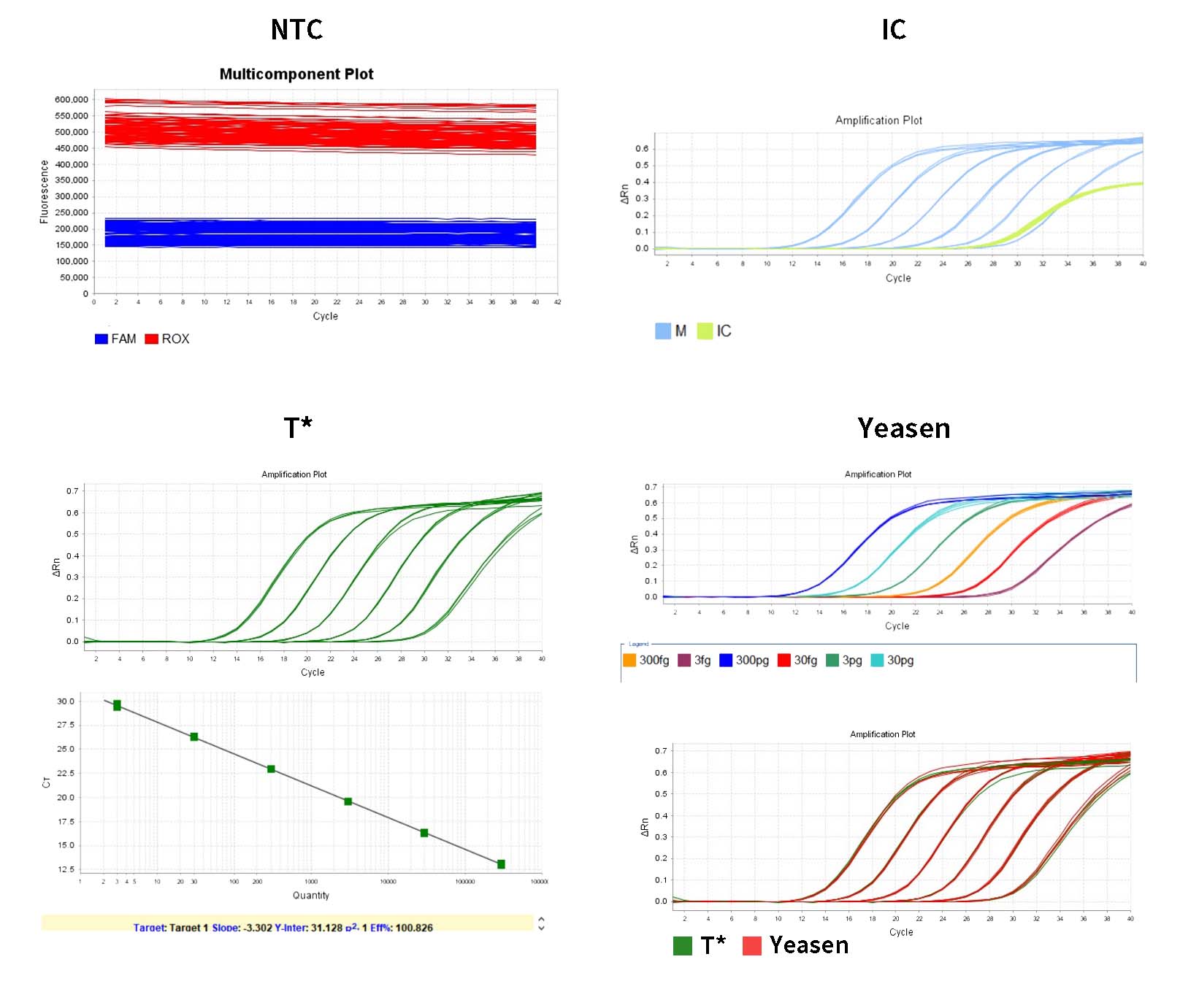
1) 시스템은 자동으로 분석의 Amplification Plot 패널에서 Threshold를 제공합니다. 시스템에서 제공하는 Threshold는 때때로 기준선에 너무 가까워서 복제 웰 간의 Ct에 큰 차이가 발생합니다. Threshold를 적절한 위치로 수동으로 조정하고 Analyze를 클릭할 수 있습니다. 그런 다음 Multicomponent Plot에서 증폭 곡선이 정상인지 처음에 확인할 수 있습니다.
2) 결과 분석 탭에서 표준 곡선 플롯을 검토합니다. R 2 , 효율성, 기울기 및 Y 절편의 값을 확인합니다. 정규 표준 곡선의 경우 R²>0.99, 90%≤Eff%≤110%, -3.6≤Slope≤-3.1입니다.
3) 분석의 '웰 테이블 보기' 창에서 각 샘플의 농도가 수량으로 표시되며, 단위는 fg/μL이고, 단위는 분석 보고서에서 변환할 수 있습니다.
4) 결과 분석의 매개변수 설정은 사용된 특정 모델과 소프트웨어 버전을 기반으로 해야 하며, 일반적으로 계측기에서 자동으로 해석할 수 있습니다.
5) 측정할 샘플 TS의 시험 결과와 샘플 스파이크 회수율 ERC를 토대로 스파이크 회수율을 계산합니다. 스파이크의 회수율은 50%~150% 사이여야 합니다. 스파이크 회수율 측정기 공식: 회수율(%) = {샘플 스파이크 검정량(예: pg/μL) - 샘플 검정량(예: pg/μL)} x 용출량(μL) / DNA 첨가량(예: pg)의 이론값 x 100%.
6) 음성 대조군 NCS의 Ct 값은 표준물질의 가장 낮은 농도 Ct의 평균보다 커야 합니다.
- 템플릿 없는 제어 NTC는 미확인이거나 Ct 값 ≥3이어야 합니다.
노트
- 이 제품은 연구 목적으로만 사용됩니다.
- 안전을 위해 실험실 가운과 일회용 장갑을 착용해 주시기 바랍니다.
3. 본 시약을 사용하기 전에 본 설명서를 주의 깊게 읽어 보시기 바랍니다. 또한, 시료 취급, 반응 시스템 준비 및 시료 추가를 포함하여 실험은 표준화되어야 합니다.
4. 사용하기 전에 각 성분이 완전히 진탕되고 저속으로 원심분리되었는지 확인하세요.
결제 및 보안
귀하의 결제 정보는 안전하게 처리됩니다. 당사는 신용카드 정보를 저장하지 않으며 귀하의 신용카드 정보에 접근할 수 없습니다.
문의
당신은 또한 좋아할 수 있습니다
자주 묻는 질문
이 제품은 연구 목적으로만 사용되며 인간이나 동물의 치료 또는 진단용으로 의도되지 않았습니다. 제품과 콘텐츠는
특정 애플리케이션에는 추가적인 제3자 지적 재산권이 필요할 수 있습니다.
예슨은 윤리적 과학에 헌신하며, 우리의 연구가 안전과 윤리적 기준을 보장하는 동시에 중요한 문제를 해결해야 한다고 믿습니다.