De afgelopen jaren is de wereldwijde incidentie van infectieziekten toegenomen, waarbij pathogenen steeds diverser en complexer worden. Een belangrijke uitdaging in dit scenario is dat ongeveer de helft van de patiënten te maken heeft met het dilemma van onbekende pathogenen, waardoor de snelle diagnose van deze pathogenen een ontmoedigende taak is. Momenteel is klinische pathogeendetectie voornamelijk afhankelijk van traditionele kweekmethoden, PCR-technieken, metagenomische sequencing (mNGS) en pathogeengerichte sequencing (tNGS).
Real-time fluorescente kwantitatieve PCR heeft aan populariteit gewonnen in de nucleïnezuurdetectie van SARS-CoV-2. Op vergelijkbare wijze heeft mNGS aandacht gekregen voor zijn bijdragen aan de strijd tegen SARS-CoV-2 en is het overgegaan van klinische settings naar dagelijks gebruik. tNGS, dat de voordelen van zowel PCR als NGS biedt, zoals snelheid, nauwkeurigheid en kosteneffectiviteit in sequentieservices, wint steeds meer aan populariteit op het gebied van klinische testen.
Tegelijkertijd is de toepassing van biologische producten uitgebreid naar biomedische wetenschap, biologische landbouw, bio-energie, biologische productie, milieubescherming en andere domeinen. Naarmate het marktaandeel van biologische producten blijft groeien, hebben overheidsinstanties en relevante autoriteiten gestandaardiseerde kwaliteitsmanagementsystemen en -normen voor deze producten vastgesteld. Een specifiek aandachtsgebied betreft de aanwezigheid van nucleïnezuurresiduen van gastheercellen in biologische producten.
Biologische producten zoals recombinante proteïnegeneesmiddelen, antilichaamgeneesmiddelen, vaccins en cel- en gentherapieën worden geproduceerd met behulp van opeenvolgende stammen of cellijnen, wat mogelijk resulteert in de retentie van gastheernucleïnezuren in de eindproducten. Residuen van gastheernucleïnezuren kunnen nadelige gevolgen hebben, zoals ongecontroleerde celproliferatie die leidt tot tumorvorming of verergering van immuunreacties door de introductie van virale genen. Daarom zijn effectieve verwijdering van nucleïnezuurresiduen en rigoureuze residudetectie cruciaal om de veiligheid van biologische producten te waarborgen en te voldoen aan onderzoeksvereisten. In de industrie worden qPCR/RT-qPCR-methoden op grote schaal gebruikt om detectiekits te ontwikkelen voor de detectie van gastheernucleïnezuurresiduen in biologische producten.
Bovendien worden commerciële moleculaire enzymen doorgaans tot expressie gebracht met behulp van recombinante engineeringstammen, zoals E. coli, wat resulteert in de aanwezigheid van gastheergenoom-DNA in deze moleculaire enzymen. Bovendien kunnen omgevings- en menselijke factoren verontreinigd DNA in moleculaire enzymproducten introduceren.
Tijdens het pathogeendetectieproces kunnen bacteriële achtergrondnucleïnezuren van contaminatie doelnucleïnezuren met een lage abundantie overschaduwen of samen met doelnucleïnezuren worden gedetecteerd. Dit kan de gevoeligheid van doeldetectie beïnvloeden of leiden tot vals-positieve resultaten, waardoor de diagnose en behandelingsbeslissingen van medische professionals worden gecompliceerd.
Bij de ontwikkeling en productie van kwaliteitscontroleproducten voor host nucleïnezuurresidutesten kan de aanwezigheid van residuele host nucleïnezuren, waaronder die van mensen, muizen, E. coli, gist en andere, in moleculaire enzymen leiden tot onnauwkeurigheden in de kwantificering van host nucleïnezuurresidukwaliteitscontroleproducten. Dit brengt potentiële veiligheidsrisico's met zich mee bij de productie van biologische producten.
Om het probleem van achtergrondbacteriën en interferentie van gastheer-nucleïnezuurresiduen op te lossen,
Tafel 1 De lijst van
| Productclassificatie | Productnaam | Catalogusnr. | Criteria van E. coli gDNA kwaliteitscontrole |
| UCF.METM ultra-low-residu enzymproducten voor qPCR/RT-qPCR | Hoog UCF.METM Hotstart-gevoelige Taq DNA-polymerase (5 (U/μL) | 14314ES | <0,005 kopieën/U |
| Hifair UCF.METM V Reverse transcriptase (200 U/μL) | 14608ES | <0,005 kopieën/U | |
| UCF.METM Muizen-RNase-remmer (40 U/μL) | 14672ES | <0,001 kopieën/U | |
| UCF.METM Hoge affiniteit RNase-remmer (40 U/μL) | 14675ES | <0,001 kopieën/U | |
| UCF.METM Uracil DNA-glycosylase (UDG/UNG), hittelabiel, 1 U/μL | 14466ES | <0,1 kopieën/U | |
| UCF.METM Uracil-DNA-glycosylase (UDG/UNG), 1 U/μL | 14454ES | <0,1 kopieën/U |
Gedeeltelijke gegevenspresentatie (UCF.ME)TM Hotstart Sensitive Taq DNA Polymerase als voorbeeld)
- Roverblijfselvan coli gDNA <0.005 exemplaren/U
- coligDNA-residuen van verschillende batches UCF.METMTaq-enzym (Cat#14314ES) werd gedetecteerd. Het resultaat toonde aan dat E. coli gDNA-residuen residuen van Taq DNA-polymerase waren ruim onder de 0,005 kopieën/E.
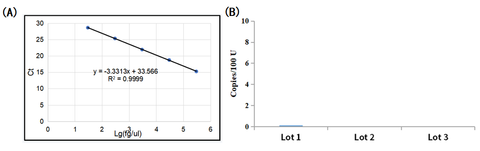
Figuur 1: Detectie van E. coli GDNA-residu van UCF.METM Taq-enzym (Cat#14314ES)
- Er werd geen resterend plasmide-DNA gedetecteerd
Plasmide-DNA-residuen van UCF.METM Taq-enzym (Cat#14314ES) en Taq DNA-polymerase van merk A werden gedetecteerd. De resultaten toonden aan dat er plasmide-DNA-residu in Taq DNA-polymerase van merk A zat. Er werd geen plasmide-DNA gedetecteerd in UCF.METM Taq-enzym (Cat#14314ES).
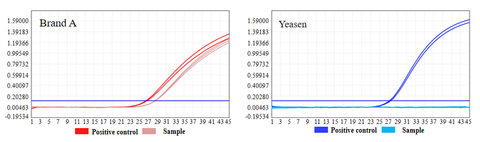
Figuur 2: Resultaten van plasmide-DNA-residudetectie
- Elf soorten veelvoorkomende achtergrondbacteriën werden niet gedetecteerd
Gebruik de UCF.METM Taq-enzym (Cat#14314ES) gekoppeld aan primers en probes van Pseudomonas aeruginosa, Acinetobacter baumannii, Serratia marcescens, Klebsiella pneumoniae, Stenotrophomonas maltophil, Streptokokken pneumoniae, Enterococcus faecium, Staphylococcus aureus, E. coli, Enterococcus faecalis om qPCR-premix te bereiden. NTC (No Template Control) werd gedetecteerd, de resultaten lieten zien dat er geen restanten van de bovenstaande 11 veelvoorkomende achtergrondbacteriën werden gedetecteerd in UCF.METM Taq-enzym (Cat#14314ES).
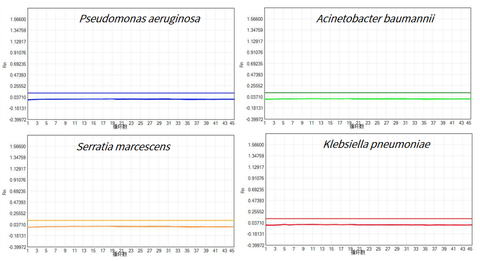
Figuur 3: Testresultaten van 11 veel voorkomende achtergrondbacteriën (beperkt door ruimte, Pseudomonas aeruginosa, Acinetobacter baumannii, Serratia marcescens En Klebsiella pneumoniae worden als voorbeelden getoond)
- Presentatie van de testcase van de klant
Het commerciële Taq-enzym en
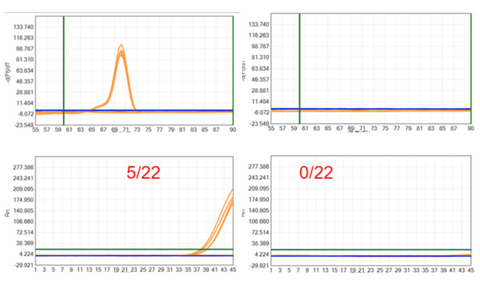
Figuur 4: Detectie resultaat van E.coli gDNA-residu (testcase van de klant)
Links: Commercieel verkrijgbaar conventioneel Taq-enzym; Rechts: UCF.METM Taq-enzym (Cat#14314ES)
Aanbeveling gerelateerd product
| Productclassificatie | Productnaam | Catalogusnr. |
| Detectiekit voor nucleïnezuurresiduen van de gastheer | 41332ES | |
| 41331ES | ||
| 41307ES | ||
| E. coli Detectiekit voor DNA-residuen van gastheercellen (2G) | 41308ES | |
| Hansenula polymorpha Host Cell DNA Residu Detectie Kit | 41317ES | |
| E. coli Host Cell RNA Residu Detectie Kit | 41318ES |
