Diepgaand leren van de belangrijkste enzymgrondstoffen die worden gebruikt in de PCR-detectie van SARS-CoV-2
Sinds maart 2022 heeft de sluwe Omicron-mutante stam opnieuw het vredige leven van mensen verstoord, de nieuwe coronavirusepidemieën braken uit in het hele land en troffen 30 provincies (autonome regio's en gemeenten). Als een effectief middel voor nauwkeurige preventie en bestrijding van de SARS-CoV-2-epidemie is nucleïnezuurdetectie een normale manier van leven geworden. "Heb je vandaag de nucleïnezuurtest gedaan?" Het is ook een dagelijkse begroeting van mensen geworden. Over dat gesproken, weet je welke kerngrondstoffen nodig zijn bij nucleïnezuurdetectie? Dit artikel introduceert de kritieke kerngrondstof in nucleïnezuurdetectie-enzym.
1. Detectieproces van nucleïnezuur voor SARS-CoV-2
2. Kernenzymen bij de extractie van nucleïnezuur
3. Kernenzymen tijdens RT-qPCR
4. Kernenzymen van SARS-CoV-2-nucleïnezuurdetectie van
1. Detectieproces van nucleïnezuur voor SARS-CoV-2
Een enzym is een uiterst belangrijke klasse biokatalysatoren met een hoge katalytische efficiëntie en reactiespecificiteit. De meeste biochemische reacties vereisen de deelname van enzymen. In het proces van nucleïnezuurdetectie van 2019-nCoV (weergegeven in Figuur 1) spelen verschillende soorten moleculaire enzymen een belangrijke rol in verschillende experimentele stadia, zoals nucleïnezuurextractie en RT-qPCR. Vervolgens zullen we, op basis van de verschillende experimentele schakels in nucleïnezuurdetectie, de belangrijkste enzymgrondstoffen sorteren die worden gebruikt in het nucleïnezuurdetectieproces.
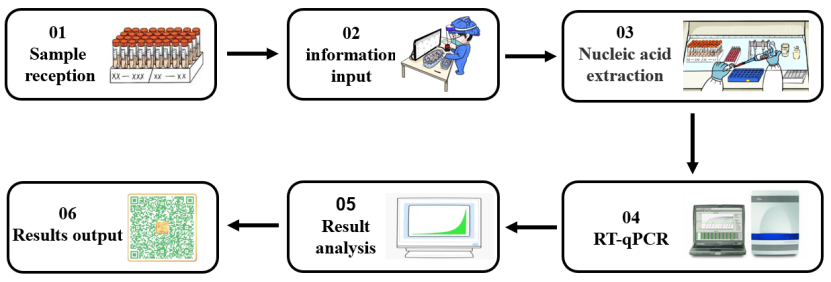
Figuur 1. Detectieproces van nucleïnezuur voor SARS-CoV-2
2. Kernenzymen bij de extractie van nucleïnezuur
Het nieuwe coronavirus nucleïnezuur extractieproces omvat hoofdzakelijk twee stappen: lysis en zuivering. Lysis is het proces van het vernietigen van de celstructuur van het monster zodat het nucleïnezuur in het monster vrij is in het lysissysteem; Zuivering is de volledige scheiding van nucleïnezuur van andere componenten in het lysissysteem, zoals eiwit, zout en andere onzuiverheden, en het reactieproces vereist de deelname van proteinase K, deoxyribonuclease I en RNase-remmers.
2.1 Protease K
Proteinase K is een serineprotease met brede splitsingsactiviteit, de splitsingsplaatsen zijn de carboxyterminale peptidebindingen van alifatische en aromatische aminozuren (Figuur 2). In het proces van nucleïnezuurextractie kan proteinase K histonen afbreken die stevig gebonden zijn aan nucleïnezuren, de scheiding van nucleïnezuren bevorderen en het extractie van nucleïnezuur in monsters gemakkelijker maken. Bovendien kan proteinase K RNA-hydrolase (RNase)-activiteit afbreken en RNase-hydrolyse van template-RNA remmen.
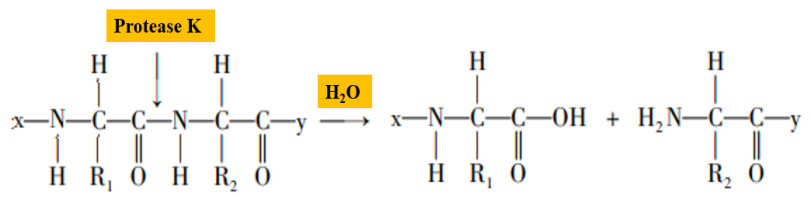
Figuur 2. Schematisch diagram van proteinase K dat peptidebindingen hydrolyseert
2.2 Desoxyribonuclease I
Deoxyribonuclease I (DNase I) kan verschillende vormen van DNA katalyseren, zich richten op het verbreken van fosfodiësterbindingen grenzend aan pyrimidines en polynucleotiden genereren met een fosfaatgroep aan het 5'-uiteinde en een hydroxylgroep aan het 3'-uiteinde. Het gemiddelde verteringsproduct is het kleinste polytetranucleotide.Bij de extractie van SARS-CoV-2-nucleïnezuur wordt DNase I voornamelijk gebruikt om genomische verontreinigingen in RNA-monsters te verwijderen, DNA-resten in RNA-templates te vermijden en de zuiverheid van templates te verbeteren.
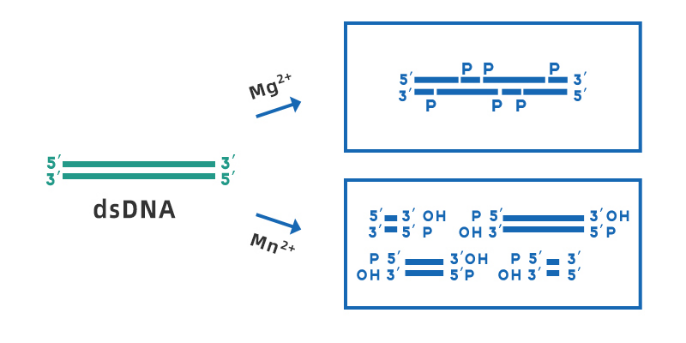
Figuur 3. Schematisch diagram van DNase I dat dsDNA splitst in aanwezigheid van Mg2+ en Mn2+
2.3 RNase-remmer
In het proces van SARS-CoV-2 nucleïnezuurdetectie kan de extractie en zuivering van monsternucleïnezuur of de bereiding van het experimentele reactiesysteem ribonuclease (RNase)-contaminatie introduceren, wat resulteert in de degradatie van de RNA-template. Om RNase-contaminatie te voorkomen, is RNase Inhibitor vereist.
RNase-remmer is een specifieke RNase-remmer in de menselijke placenta, die zich specifiek aan RNase kan binden om een complex te vormen met een niet-covalente binding en RNase te inactiveren.
3. Kernenzymen tijdens RT-qPCR
Nadat de nucleïnezuurextractie van het SARS-CoV-2-monster is voltooid, kan de nucleïnezuurdetectie worden voltooid door RT-qPCR. In het proces van deze experimenten zijn DNA-polymerase, reverse transcriptase en uracil-DNA-glycosylase allemaal essentiële kernenzymgrondstoffen.
3.1 Omgekeerde transcriptase
Na extractie en zuivering heeft het SARS-CoV-2 RNA reverse transcriptase nodig om dNTP-polymerisatie te katalyseren om een cDNA-sequentie te genereren die complementair is aan het template-RNA (Figuur 4). Voor RT-qPCR-reactie moet een hoge temperatuurbestendige reverse transcriptase worden geselecteerd. Momenteel wordt MMLV reverse transcriptase het meest gebruikt, vanwege het gebrek aan DNA-endonuclease-activiteit en lage RNase H-activiteit, heeft het meer voordelen bij de toepassing van cDNA-klonering.
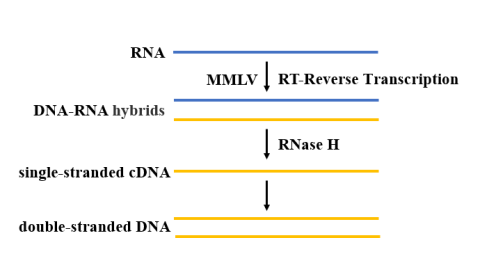
Figuur 4. Schematisch diagram van het omgekeerde transcriptieproces
3.2 DNA-polymerase
Nadat het proces van omgekeerde transcriptie van de template is voltooid om dubbelstrengs cDNA te genereren, moet de DNA-polymerase van de 'zielenspeler' in de PCR-reactie verschijnen door vrije deoxyribonucleotiden te polymeriseren om de DNA-keten te verlengen. Een grote hoeveelheid template-DNA wordt in vitro versterkt om het doel van virale nucleïnezuurdetectie te bereiken.
De DNA-polymerase die gewoonlijk wordt gebruikt in RT-qPCR-reacties is hot-start Taq DNA-polymerase. Dit type enzym is inactief bij kamertemperatuur.Het heeft alleen polymerisatieactiviteit na hot-starting, wat de generatie van achtergrondsignalen kan minimaliseren. Het lost de problemen op van niet-specifieke amplificatie veroorzaakt door primer-dimeergeneratie of mismatch in conventionele PCR-reacties. Momenteel omvatten de algemeen gebruikte DNA-polymerase hot-start modificatiemethoden voornamelijk chemische modificatie, ligandmodificatie en antilichaammodificatie. De principes van verschillende hot-start modificatiemethoden worden weergegeven in Figuur 5.
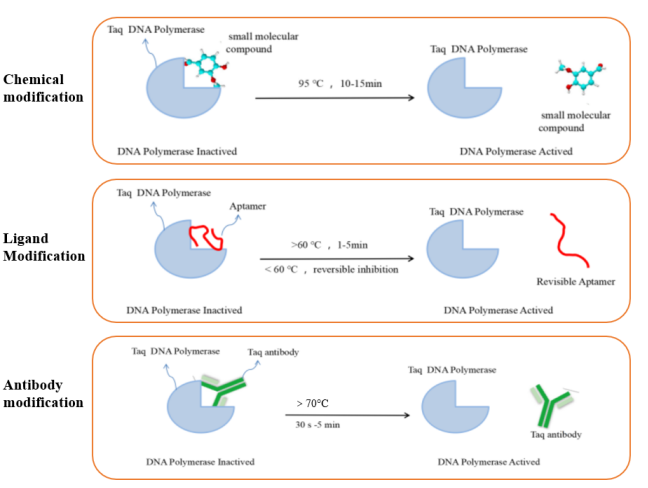
Figuur 5. Schematisch diagram van verschillende typen gemodificeerde hot-start-enzymen
3.3 Uracil-DNA-glycosylase
Bij het detecteren van nieuw coronavirus nucleïnezuur is aerosolvervuiling in de werkomgeving de meest voorkomende factor die vals-positieve PCR-resultaten veroorzaakt. Door UDG-enzym (Uracil DNA Glycosylase, uracil DNA glycosylase) toe te voegen aan het amplificatiesysteem, kunnen de resterende amplificatieverontreinigende stoffen (meestal in de vorm van aerosolen) die in het PCR-systeem zijn gemengd, effectief worden geëlimineerd om de nauwkeurigheid van de amplificatieresultaten te garanderen. Het anti-vervuilingsprincipe van het UDG-enzym wordt weergegeven in Figuur 6.
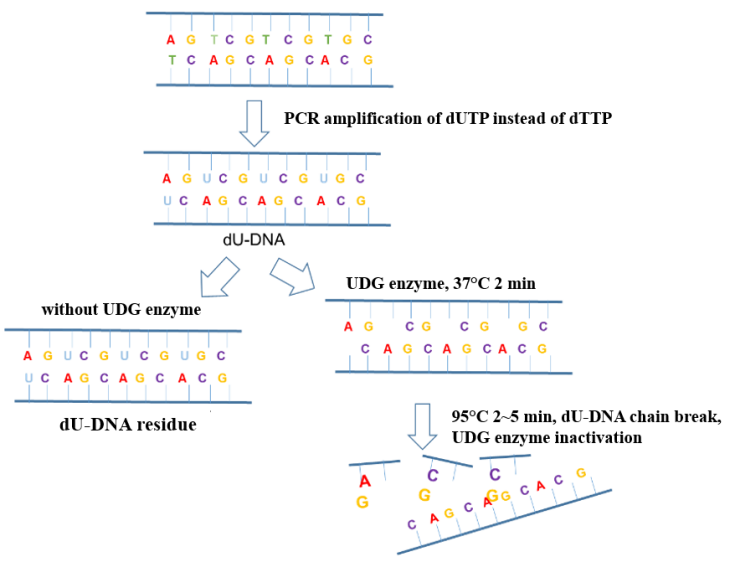
Figuur 6. Schematisch diagram van het anti-vervuilingsprincipe van het UDG-enzym
4.Kernenzymen van SARS-CoV-2-nucleïnezuurdetectie van Yeasen
| Proces | Beschrijving | Productnaam | SKU |
| Monsterverwerking | Eiwitvertering | 10401ES | |
| RNA-extractie | Recombinant DNase I (RNase-vrij) (Vraag) | 10325ES | |
| RNase-remming | Muizen RNase-remmer (40 eenheden/μL) | 10603ES | |
| Omgekeerde transcriptie | Geschikt voor RT-qPCR | Hifair™ V Omgekeerde transcriptase (200 eenheden/μL) | 11300ES |
| HifairTM V Reverse Transcriptase (600U/μL) GLyceroL-vrij (Vraag) | 11301ES | ||
| PCR-amplificatie | DNA-polymerase met snelle start | 10726ES | |
| Thermische UDG | Uracil DNA-glycosylase (UDG/UNG), hittelabiel, 1 U/μL | 10303ES |
Over het lezen:
Selectie van reverse transcriptase
Muizen-RNase-remmers - Elimineer RNase-verontreiniging succesvol en behoud RNA
