Endonucleases de restrição da série Yeasen FuniCut™, digestão de 5 minutos com tampão universal
A clonagem molecular é usada em quase todos os laboratórios. Endonucleases de restrição são um componente crucial em experimentos de clonagem molecular, mas as pessoas também são frequentemente incomodadas por vários problemas com experimentos de endonucleases de restrição. Problemas comuns são os seguintes:
- Existem tantos tipos de endonucleases de restrição que é difícil escolher.
- Clivagem incorreta, clivagem aleatória ou clivagem enzimática incompleta.
- A taxa de digestão é lenta; pode levar uma hora ou até mesmo a noite toda.
- Além disso, experimentos de digestão enzimática múltipla também precisam escolher uma variedade de enzimas de digestão bTo
Para provar com ide a você uma compreensão mais completa das endonucleases de restrição, o seguinte explicará brevemente o que são endonucleases de restrição e como várias endonucleases são classificadas em resposta à primeira pergunta. A série
1. O que é endonuclease de restrição?
2. Restrições nomenclatura endonuclease
3. Restrições na classificação de endonucleases
4. Endonucleases de restrição rápida
1. O que é endonuclease de restrição?
Endonucleases de restrição são uma classe de enzimas que podem reconhecer sequências de nucleotídeos específicas em moléculas de DNA fita dupla e cortar ligações fosfodiéster em cadeias de DNA em locais específicos.
Diferentes endonucleases de restrição reconhecerão diferentes sequências de DNA. Elas podem cortar o DNA dentro da sequência de reconhecimento ou em um local não muito distante da sequência de reconhecimento, resultando em vários produtos, conforme ilustrado na Figura 1. BamHI forma extremidades pegajosas, KpnI forma extremidades 3'pegajosas e EcoRV forma extremidades rombas.
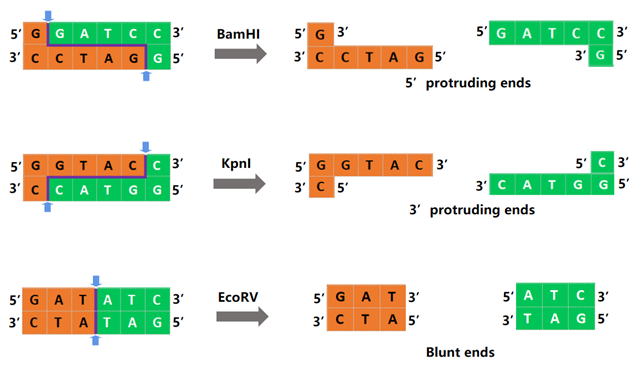
Figura 1. Diagrama esquemático da digestão de BamHI, KpnI, EcoRV
2. Restrições nomenclatura endonuclease
O nome do gênero, o nome da espécie, a cepa bacteriana ou sorotipo e a ordem de descoberta são os principais fatores usados para nomear endonucleases de restrição. As diretrizes de nome são exibidas na Tabela 1 abaixo usando BstEII como exemplo:
Tabela 1. Nomenclatura de restrições de endonuclease
| Abreviação | Nome completo | Implicação |
| B | Bacilo | Primeira letra do nome do gênero |
| st | estearotérmofilo | As duas primeiras letras do nome da espécie |
| E | E | A primeira letra do nome da cepa |
| II | Segunda descoberta | A ordem encontrada em tais bactérias |
3.Restrições na classificação de endonucleases
De acordo com a complexidade da estrutura, o modo de ação e a diferença entre os cofatores, as enzimas de restrição podem ser divididas em quatro categorias. A tabela 2 a seguir resume as características e enzimas típicas de cada categoria.
Tabela 2. As variações entre várias endonucleases de restrição
| Classe de endonuclease | Características | Espécies típicas de enzimas |
| Tipo I | 1. Clivagem de reconhecimento e modificação. 2. Ele pode reconhecer sequências específicas de DNA, e o local de digestão fica indefinidamente distante do local de reconhecimento, até milhares de bases. 3. A ação necessita de ATP. | EcoB, EcoK, etc. |
| Tipo II | 1. Tem apenas a função de identificar o corte. 2. A sequência de reconhecimento é frequentemente uma sequência palíndrômica curta (cerca de 4–8 pb), e o sítio de digestão específico da endonuclease de restrição é normalmente a sequência reconhecida. 3. A ação necessita de Mg2+. 4. O tipo de enzimas de restrição mais frequentemente utilizadas na clonagem molecular. | HindIII, NotI, etc. |
| Tipo III | 1. Reconhecimento e modificação da digestão. 2. Sobreseparatesp separe o local de reconhecimento do local de digestão. 3. A ação necessita de ATP. | HinfIII et al. |
| Tipo IV | 1. Corta apenas sequências de DNA metiladas. 2. Cerca de 30 pb separam o local de reconhecimento do local de digestão. | McrA, McrBC, etc. |
Conforme indicado na Tabela 3 abaixo, ele também pode ser dividido em três grupos com base nas semelhanças e diferenças entre os locais de reconhecimento e digestão.
Tabela 3.Análise comparativa de Isosquizômero, Neoesquizômero e Isocaudarner
|
| Isosquizômero | Neoesquizômero | Isocaudanos |
| sequência de reconhecimento | mesmo | mesmo | diferente |
| locais de digestão | mesmo | diferente | mesmo |
| representante típico | AgeI e BshTI: ambos reconhecem e clivam 5′-A↓CCGGT-3′ | SmaI (5′-CCC↓GGG-3 ′) e XmaI (5′-C↓CCGGG-3 ′) | BamHI (5'-G↓GATCC-3') e BglII (5'-A↓GATCT-3') |
Tendo em vista o fato de que algumas endonucleases de restrição no mercado ainda requerem digestão durante a noite, e os produtos são propensos à digestão incorreta, a
4. Yeasen FuniCut™ Endonucleases de restrição rápida
Figura 2. Endonucleases de restrição rápida
4.1 Características
- Digestão rápida: a digestão pode ser concluída em 5 a 15 minutos, o que pode ser reduzido em mais de 1 a 2 horas em comparação com experimentos de digestão convencionais.
- Tampão universal: A reação de clivagem multienzimática é simplificada pelo fato de que qualquer combinação de endonucleases pode usar o mesmo tampão.
- Efeito da digestão enzimática: comparável ao N* e apropriado para seu tampão.
- A eletroforese direta simplifica o processo ao fornecer um tampão de cor vermelha e permitir que os produtos da digestão sejam entregues diretamente para eletroforese.
- Digestão precisa: Mesmo com a digestão noturna, há muito pouca atividade estelar.
4.2 Exposição de desempenho
4.2.1 No FuniCut™ Buffer, a digestão simples, dupla e tripla ocorre em 5 minutos.

Figura 3. Digestão simples, dupla e tripla usando o FuniCut™ O tampão normalmente pode ser concluído em 5 minutos, por exemplo, a digestão tripla usando EcoRI+KpnI+SmaI.
4.2.2 O impacto da digestão enzimática é comparável ao de N* e T*, e funciona bem com seus tampões.

Figura 4. Comparado a produtos N*, T* similares, o efeito de digestão enzimática do
4.2.3 Digestão noturna com atividade estelar muito baixa
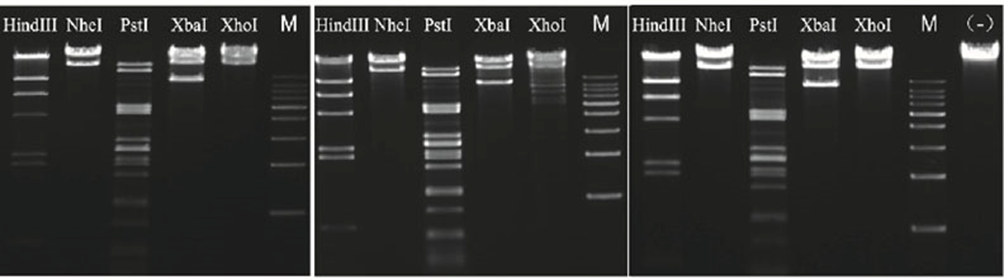
Figura 5. Utilize as endonucleases rápidas HindIII, NheI, PstI, XbaI e XhoI de
4.2.4 Altamente redundante e capaz de lidar com algum substrato excedente.
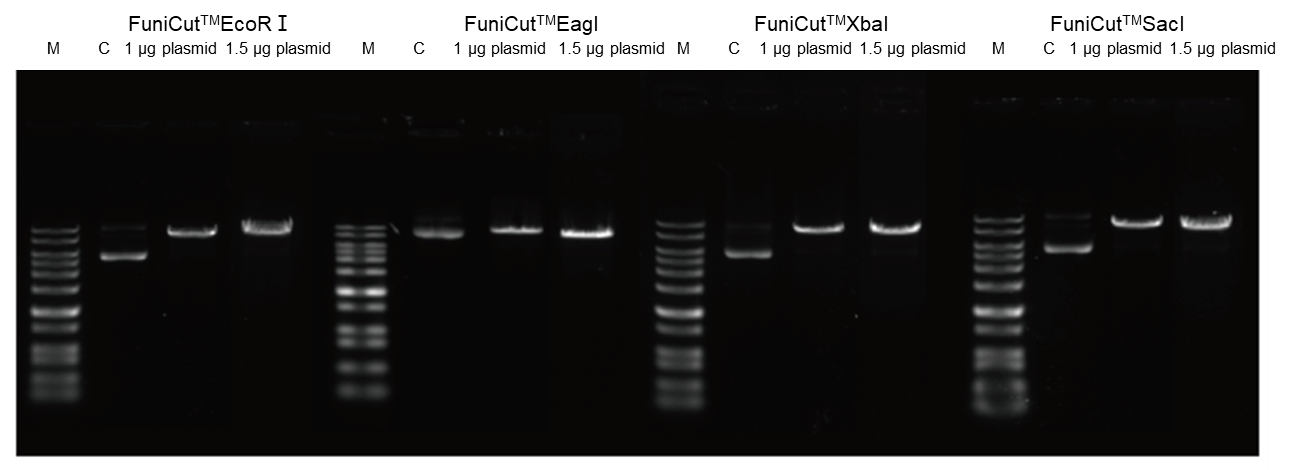
Figura 6. Usando as endonucleases de restrição rápida FuniCut™ EcoR I, Eag I, XbaI e Sac I, 1 μg e 1,5 μg de plasmídeo foram usados como substratos, respectivamente, e digeridos por 5 min, e os produtos digeridos foram submetidos à eletroforese em gel de agarose.
Tabela 4.Compatibilidade de produtos e tampão de reação
| Nome do produto | Gato# | Sequência de reconhecimento | Temperatura | Temperatura de desativação térmica |
| Tampão T* | Tampão N* | Tampão Ta* | Base protegida (bp) | Respostas |
| FuniCut™ AscI (indagar) | 15001ES50 | GG/CGCGCC | 37 | 80 | 100 | 100 | 100 | 100 | 1 | 50T |
| FuniCut™ AvrII (indagar) | 15002ES25 | C/CTAGG | 37 | 80 | 100 | 100 | 100 | 100 | 3 | 25T |
| FuniCut™ BamHI (indagar) | 15003ES76 | G/GATCC | 37 | 80 | 100 | 100 | 100 | 100 | 3 | 500T |
| FuniCut™ BclI (indagar) | 15004ES62 | T/GATCA | 37 | 80 | 100 | 100 | 100 | 100 | 3 | 125T |
| FuniCut™ BsaI (indagar) | 15005ES50 | GGTCTC(1/5) | 37 | 80 | 100 | 100 | 100 | 100 | Não | 50T |
| FuniCut™ BstEII (indagar) | 15006ES60 | G/GTNACC | 37 | 80 | 100 | 75 | 100 | 75 | Não | 100T |
| FuniCut™ ClaI (indagar) | 15007ES50 | AT/CGAT | 37 | 80 | 100 | 100 | 100 | 100 | 4 | 50T |
| FuniCut™ DpnII (indagar) | 15008ES50 | /GATC | 37 | 80 | 100 | 75 | 100 | 75 | Não | 50T |
| FuniCut™ EagI (indagar) | 15009ES25 | C/GGCCG | 37 | 80 | 100 | 100 | 100 | 100 | 2 | 25T |
| FuniCut™ EcoRI (indagar) | 15010ES78 | G/AATTC | 37 | 80 | 100 | 100 | 100 | 100 | 5 | 600T |
| FuniCut™ EcoRV (indagar) | 15011ES70 | GAT/ATC | 37 | 80 | 100 | 100 | 100 | 100 | 5 | 200T |
| FuniCut™ HindIII (indagar) | 15012ES76 | Um/AGCTT | 37 | 80 | 100 | 75 | 100 | 100 | 3 | 500T |
| FuniCut™ HpaI (indagar) | 15013ES50 | GTT/AAC | 37 | 80 | 100 | 100 | 100 | 50 | 1 | 50T |
| FuniCut™ KpnI (indagar) | 15015ES70 | GGTAC/C | 37 | 80 | 100 | 100 | 100 | 100 | 2 | 200T |
| FuniCut™ MluI (indagar) | 15016ES60 | Um/CGCGT | 37 | 80 | 100 | 75 | 100 | 100 | 3 | 100T |
| FuniCut™ NcoI (indagar) | 15018ES30 | C/CATGG | 37 | 80 | 100 | 100 | 100 | 100 | 3 | 30T |
| FuniCut™ NdeI (indagar) | 15019ES70 | CA/TATG | 37 | 80 | 100 | 100 | 100 | 100 | 3 | 200T |
| FuniCut™ NheI (indagar) | 15020ES30 | G/CTAGC | 37 | 80 | 100 | 100 | 100 | 100 | 3 | 30T |
| FuniCut™ NotI (indagar) | 15021ES50 | GC/GGCCGC | 37 | 80 | 100 | 100 | 100 | 100 | 2 | 50T |
| FuniCut™ PstI (indagar) | 15022ES76 | CTGCA/G | 37 | Não | 100 | 100 | 100 | 100 | 2 | 500T |
| FuniCut™ SacI (indagar) | 15023ES60 | GAGCT/C | 37 | 80 | 100 | 100 | 100 | 100 | 3 | 100T |
| FuniCut™ SalI (indagar) | 15024ES70 | G/TCGAC | 37 | 80 | 100 | 100 | 100 | 100 | 3 | 200T |
| FuniCut™ SbfI (indagar) | 15025ES25 | CCTGCA/GG | 37 | 80 | 100 | 100 | 100 | 100 | 2 | 25T |
| FuniCut™ Pequeno (indagar) | 15027ES60 | CCC/GGG | 37 | 80 | 100 | 100 | 100 | 100 | 1 | 100T |
| Jogo FuniCut™ (indagar) | 15028ES50 | Um/CTAGT | 37 | 80 | 100 | 100 | 100 | 100 | 2 | 50T |
| FuniCut™ SpH I (indagar) | 15029ES50 | GCATG/C | 37 | 80 | 100 | 100 | 100 | 100 | 1 | 50T |
| FuniCut™ SspI (indagar) | 15030ES56 | AAT/ATT | 37 | 80 | 100 | 50 | 100 | 100 | 2 | 60T |
| FuniCut™ StuI (indagar) | 15031ES60 | AGG/CCT | 37 | 80 | 100 | 100 | 100 | 100 | 1 | 100T |
| FuniCut™ TaqI (indagar) | 15032ES70 | T/CGA | 65 | Não | 100 | 100 | 100 | 100 | 4 | 200T |
| FuniCut™ XbaI (indagar) | 15033ES76 | T/CTAGA | 37 | 80 | 100 | 50 | 100 | 100 | 2 | 500T |
| FuniCut™ XhoI (indagar) | 15034ES76 | C/TCGAG | 37 | 80 | 100 | 100 | 100 | 100 | 4 | 500T |
| FuniCut™ FspI (indagar) | 15036ES50 | TGC/GCA | 37 | 80 | 100 | 100 | 100 | 100 | 3 | 50T |
| FuniCut™ HinfI (indagar) | 15038ES76 | G/ANTC | 37 | 80 | 100 | 100 | 100 | 50 | 2 | 500T |
| FuniCut™ ApaLI (indagar) | 15039ES70 | G/TGCAC | 37 | 80 | 100 | 75 | 100 | 100 | 4 | 200T |
| FuniCut™ NruI (indagar) | 15040ES50 | TCG/CGA | 37 | Não | 100 | 100 | 100 | 100 | 5 | 50T |
| FuniCut™ PacI (indagar) | 15041ES25 | TTAAT/TAA | 37 | 80 | 100 | 100 | 100 | 100 | 2 | 25T |
| FuniCut™ PvuII (indagar) | 15042ES70 | CAG/CTG | 37 | Não | 100 | 100 | 100 | 100 | 4 | 200T |
| FuniCut™ SacII (indagar) | 15043ES50 | CCGC/GG | 37 | 80 | 100 | 100 | 100 | 100 | 4 | 50T |
| FuniCut™ NsiI (indagar) | 15044ES25 | ATGCA/T | 37 | 80 | 100 | 100 | 100 | 100 | 3 | 25T |
| FuniCut™ Esp3I (BsmBI) (indagar) | 15048ES30 | CGTCTC(1/5) | 37 | 80 | 100 | 100 | 100 | 100 | 5 | 30T |
| FuniCut™ BglII (indagar) | 15049ES60 | Um/GATCT | 37 | 80 | 100 | 100 | 100 | 50 | 2 | 100T |
| FuniCut™ BstBI (indagar) | 15050ES60 | TT/CGAA | 37 | 80 | 100 | 100 | 100 | 100 | Não | 100T |
| FuniCut™ MnlI (indagar) | 15051ES50 | CCTC(7/6) | 37 | 80 | 100 | 100 | 100 | 100 | 2 | 50T |
CpG: Afetado pela metilação de CpG.
EK: Afetado pela metilação de EcoKI.
EB: Afetado pela metilação do EcoBI.
Dcm: Afetado pela metilação de Dcm.
Barragem: Afetada pela metilação da barragem.

