O que fazer quando os resultados do qPCR são estranhos?
Os experimentos de qPCR podem parecer fáceis e, às vezes, difíceis. Após a conclusão do design do primer, as amostras de qPCR são adicionadas passo a passo como PCR, e o experimento pode ser concluído na máquina. Existem vários problemas comuns em experimentos de qPCR pelo método de corante e, dadas as possíveis causas e soluções, esperamos que você possa usá-los para resolver os problemas em experimentos de qPCR.
1. O que é qPCR?
2. Curva de amplificação qPCR anormal
3. Curva de fusão qPCR anormal
4. Informações sobre pedidos
5. Produtos relacionados
6. Sobre a leitura
1. O que é qPCR?
Desde que a técnica de reação em cadeia da polimerase (PCR) foi inventada, a PCR é provavelmente a técnica mais amplamente usada em biologia molecular devido à sua simplicidade, baixo custo, confiabilidade, rapidez e alta sensibilidade. qPCR é uma tecnologia desenvolvida pela tecnologia PCR. Durante o processo de amplificação de DNA, o método de detecção da quantidade total de produtos após cada ciclo de PCR com corantes fluorescentes não só tem a rapidez e sensibilidade da PCR, mas também tem mais especificidade e monitoramento alto em tempo real, quantificação repetível e precisa e outras vantagens. qPCR é uma tecnologia que usa um dispositivo de amplificação de PCR quantitativo em tempo real, ou seja, um instrumento qPCR, para monitorar produtos de amplificação de ácido nucleico em tempo real durante o processo de PCR. qPCR realiza a análise quantitativa do modelo inicial por meio da análise do valor Ct e da curva padrão. Em 1992, o japonês Higuchi propôs pela primeira vez a "tecnologia de PCR quantitativa fluorescente em tempo real". Em 1996, a American Biological Company lançou o primeiro instrumento de PCR quantitativo fluorescente do mundo, que é composto por um sistema de ciclo térmico de amplificação de PCR, sistema óptico de detecção de fluorescência e software de computador e aplicativo. Ele pode monitorar produtos de amplificação de ácido nucleico em tempo real por meio de corantes fluorescentes ou sondas fluorescentes. , por meio da relação de função matemática, combinada com o software para análise de resultados, o cálculo da quantidade inicial do modelo da amostra a ser testada é realizado. Portanto, a tecnologia de PCR quantitativa fluorescente em tempo real tem sido amplamente utilizada. Os métodos de marcação fluorescente para qPCR são divididos em método de mosaico de corante fluorescente com base no método de corante SYBR Green I, método de sonda fluorescente com base no método de sonda Taqman (sonda de ciclagem, Bracon molecular, etc.), método de primer de corante extintor.
Os experimentos de qPCR podem parecer fáceis e, às vezes, difíceis. Após a conclusão do design do primer, as amostras de qPCR são adicionadas passo a passo como PCR, e o experimento pode ser concluído na máquina. No entanto, muitos pequenos detalhes precisam de atenção, como nenhum líquido pendurado na parede do tubo, nenhuma bolha no tubo e a necessidade de minimizar o contato direto entre a parede do tubo, a tampa do tubo, etc., para reduzir erros experimentais ou resultados experimentais estranhos. Xiaoyi compilou vários problemas comuns em experimentos de qPCR pelo método de corante e forneceu as possíveis causas e soluções, que esperamos que você possa usar para resolver os problemas em experimentos de qPCR.
2. Curva de amplificação qPCR anormal
2.1 Anormalidades na fase basal do gráfico logarítmico da curva de amplificação
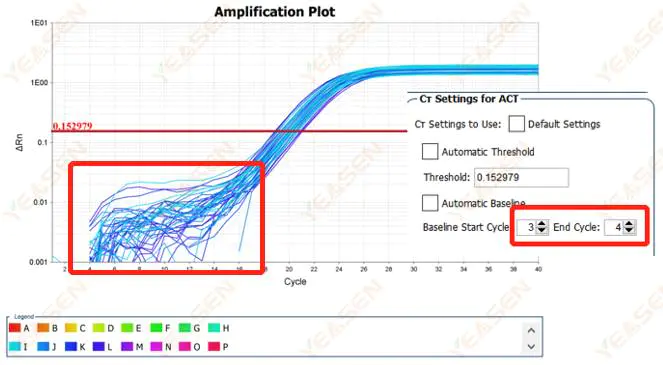
Figura 1. Gráfico de amplificação
Possíveis causas:
Configuração de linha de base inadequada.
Soluções:
Recomenda-se aumentar o valor final da linha de base.O diagrama de exemplo de ajuste é o seguinte.

Figura 2. Gráfico de amplificação
2.2 Segmentação da curva de amplificação do gráfico de logaritmo
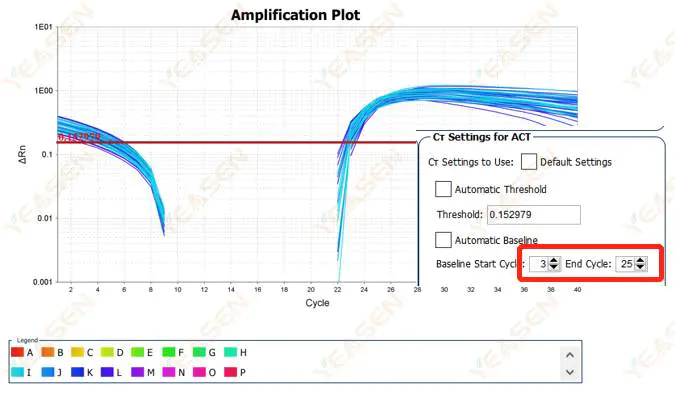
Figura 3. Gráfico de amplificação
Possíveis causas:
A linha de base está definida muito alta.
Soluções:
É recomendado reduzir o valor do ponto final da linha de base. O diagrama de exemplo de ajuste é o seguinte.
2.3 Gráfico de linearidade da curva de amplificação não suave
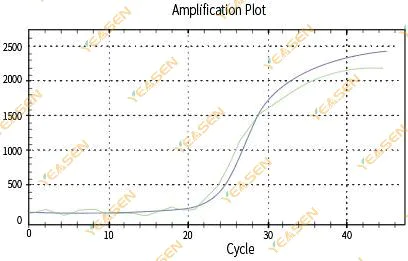
Figura 4. Gráfico de amplificação
Possíveis causas:
1) O tubo de reação de PCR não foi bem tampado e a solução de reação vazou.
2)Solução de reação de PCR com parede suspensa.
3) O instrumento não está calibrado (incluindo calibração automática ou calibração ROX).
4) O sistema possui muitos inibidores, o que leva à fluorescência instável.
5) O uso excessivo do instrumento resulta em coleta de fluorescência instável.
Soluções:
1)Pressione firmemente a tampa do tubo.
2) Misture bem o reagente e coloque-o cuidadosamente no instrumento quantitativo após a centrifugação completa.
3)Calibre o instrumento.
4) Melhorar a pureza do RNA e selecionar reagentes de transcrição reversa apropriados.
2.4 Gráfico linear de curva amplificada em ziguezague em um platô
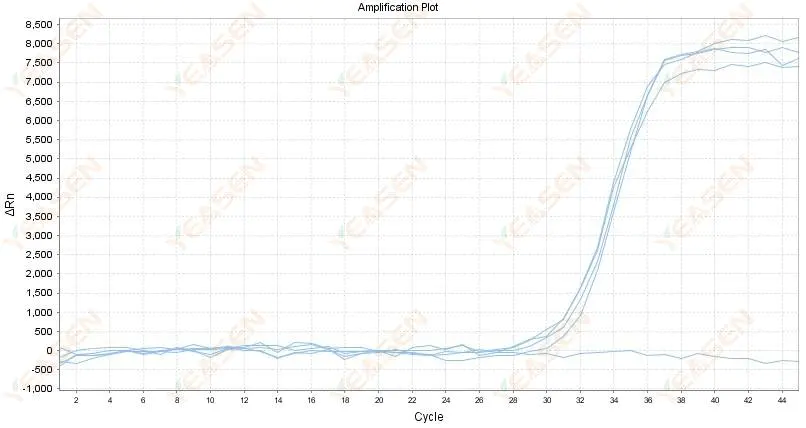
Figura 5. Gráfico de amplificação
Possíveis causas:
1) A pureza do RNA é baixa e há muitas impurezas.
2) O instrumento é usado por muito tempo.
Soluções:
1) Extraia novamente RNA de alta qualidade.
2) Dilua o molde de RNA para reduzir a concentração de impurezas.
3) Calibre o instrumento.
2.5 O gráfico linear da curva de amplificação não consegue atingir um platô
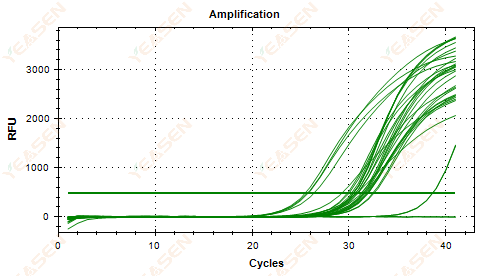
Figura 6. Gráfico de amplificação
Possíveis causas:
1) Baixa concentração de modelo (valor Ct em torno de 35).
2) Poucos ciclos de amplificação.
3) Baixa eficiência de amplificação do reagente (Ct pequeno, mas também incapaz de atingir o platô).
Soluções:
1) Aumente a concentração do modelo.
2) Aumente o número de ciclos.
3) Aumentando Mg2+ concentração.
2.6 Curva de amplificação, gráfico linear, flacidez de platô
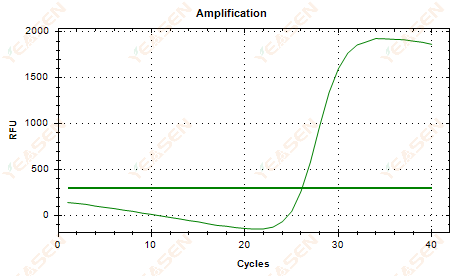
Figura 7. Gráfico de amplificação
Possíveis causas:
1) Presença de degradação (degradação do produto de amplificação, degradação SYBR).
2) A tampa do tubo não está devidamente coberta e o reagente é volatilizado.
3) A concentração de cDNA está muito alta (se o valor de Ct for muito pequeno, o limite de fluorescência é elevado e a flacidez é mais grave).
4) Bolhas de ar no tubo e depois desaparecem.
Soluções:
1) Melhore a pureza do sistema.
2) Reduza a quantidade de cDNA (dilua o modelo).
3) Diminua o valor final da linha de base.
2.7 Grande valor de Ct na curva de amplificação
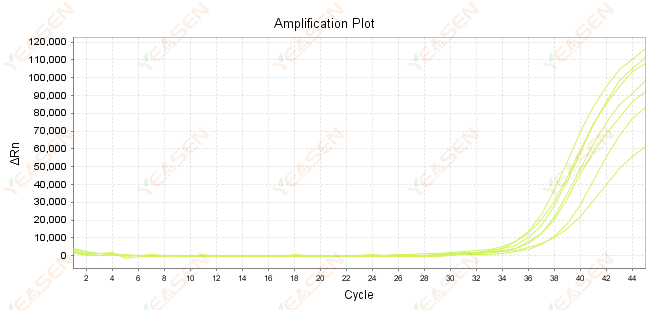
Figura 8. Gráfico de amplificação
Possíveis causas:
1) Pouca quantidade de modelos.
2) Baixa eficiência de amplificação.
3) O fragmento de PCR é muito longo.
4) Presença de inibidores no sistema de reação.
Soluções:
1) Reduza os tempos de diluição ou aumente a quantidade de modelo para que o valor de Ct fique entre 15 e 30 o máximo possível.
2) Otimize as condições de reação, tente um procedimento de amplificação em três etapas ou redesenhe os primers.
3) O comprimento do produto de PCR foi projetado para ficar entre 100 e 150 pb, não sendo recomendado exceder 300 pb.
4) Reprodução de modelos de maior pureza.
2.8 Baixa repetibilidade das curvas de amplificação
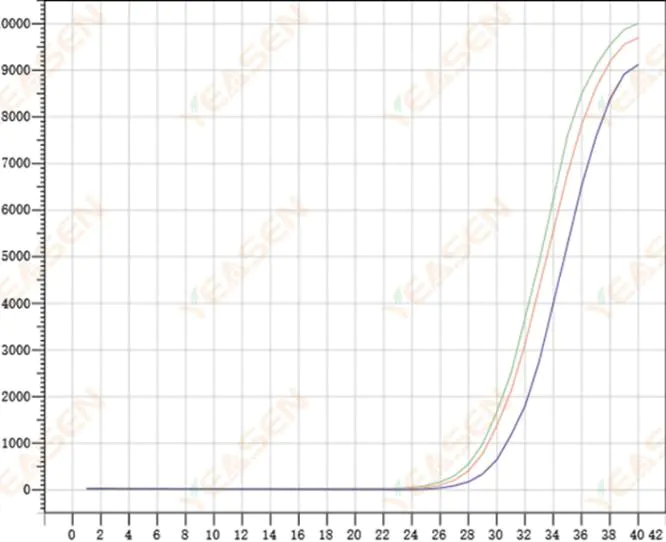
Figura 9. Gráfico de amplificação
Possíveis causas:
1) Grandes erros de pico.
2) Os reagentes e sistemas não estão bem misturados.
3) Baixo número de cópias de cDNA.
4) Nenhuma calibração Rox foi usada.
Soluções:
1) Calibrando pipetas.
2) Mistura completa do sistema de reação.
3) Baixa concentração de modelo, baixa repetibilidade, 4-6 furos compostos e 1-2 desvios de valores maiores podem ser descartados adequadamente.
4) É melhor usar a calibração Rox. Se o reagente usado não contiver Rox, o corante de referência precisa ser selecionado como Nenhum.
2.9 Curvas de amplificação aleatórias
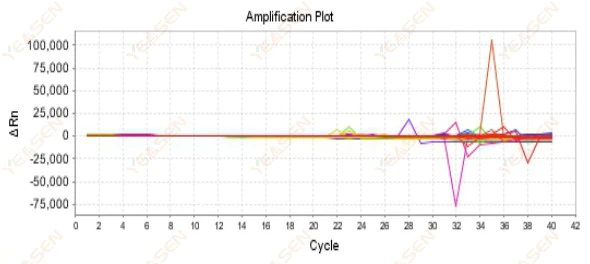
Figura 10. Gráfico de amplificação
Possíveis causas:
A incompatibilidade entre a concentração de Rox e o modelo.
Soluções:
Altere a configuração do corante de referência no instrumento de ROX para NENHUM e veja se a curva de amplificação retorna ao normal.
2.10 Pico NTC
2.10.1 Ct>35, curva de fusão valor Tm <80℃
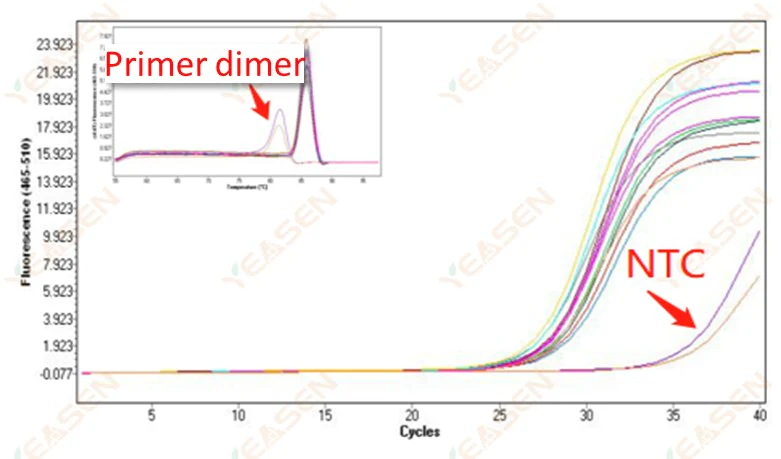
Figura 11. Gráfico de amplificação
Possíveis causas:
Resultados da dimerização do primer.
Soluções:
Primers otimizados.
2.10.2 Valor Ct <35, curva de fusão NTC e formato de pico da curva de fusão de genes se sobrepõem
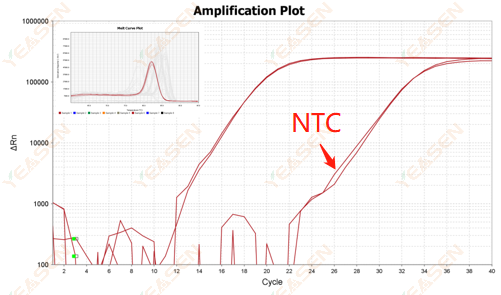
Figura 12. Gráfico de amplificação
Possíveis causas:
Contaminação do sistema de reação.
Soluções:
Verifique as fontes de poluição uma por uma.
3. Curva de fusão qPCR anormal
3.1 Curva de fusão de pico único, mas não acentuada
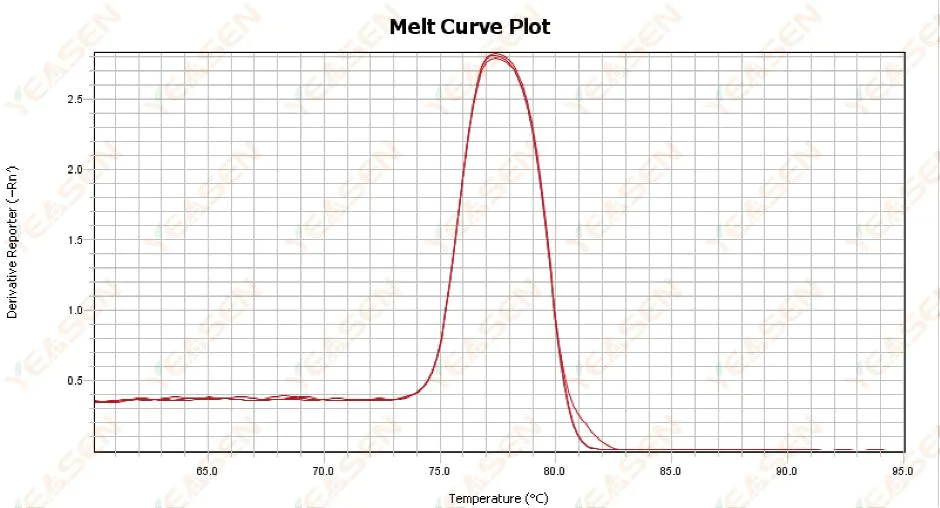
Figura 13. Gráfico da curva de fusão
Possíveis causas:
1) Está relacionado à composição do reagente e ao modelo do instrumento.
2) Presença de amplificação não específica de fragmentos de tamanho semelhante.
Soluções:
1) O intervalo de temperatura do pico inicial ao pico final não é maior que 7°C. Os resultados são considerados utilizáveis, ou seja, pico único.
2) Eletroforese em agarose de alta concentração (por exemplo, 3% de agarose) foi realizada para auxiliar na determinação.
3.2 A curva de fusão é de pico duplo e o pico inferior Tm é anterior a 80°C
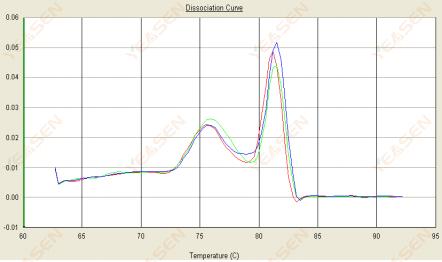
Figura 14. Curva de dissociação
Possíveis causas:
Presença de dímeros de primer.
Soluções:
Aumente a temperatura de recozimento, diminua a concentração do primer ou redesenhe os primers.
3.3 A curva de fusão é de pico duplo e o pico inferior Tm é após 80°C
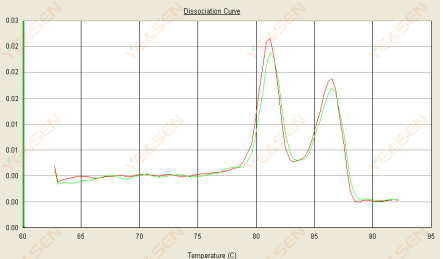
Figura 15. Curva de dissociação
Possíveis causas:
1) A baixa especificidade do primer leva à amplificação de produtos não específicos.
2) Contaminação por gDNA.
Soluções:
1) Especificidade do primer de verificação de explosão, redesenho se for ruim.
2) Confirmação pelo controle negativo do NRC, se houver, precisa preparar novamente o modelo.
3.4 Picos de curva de fusão desordenados
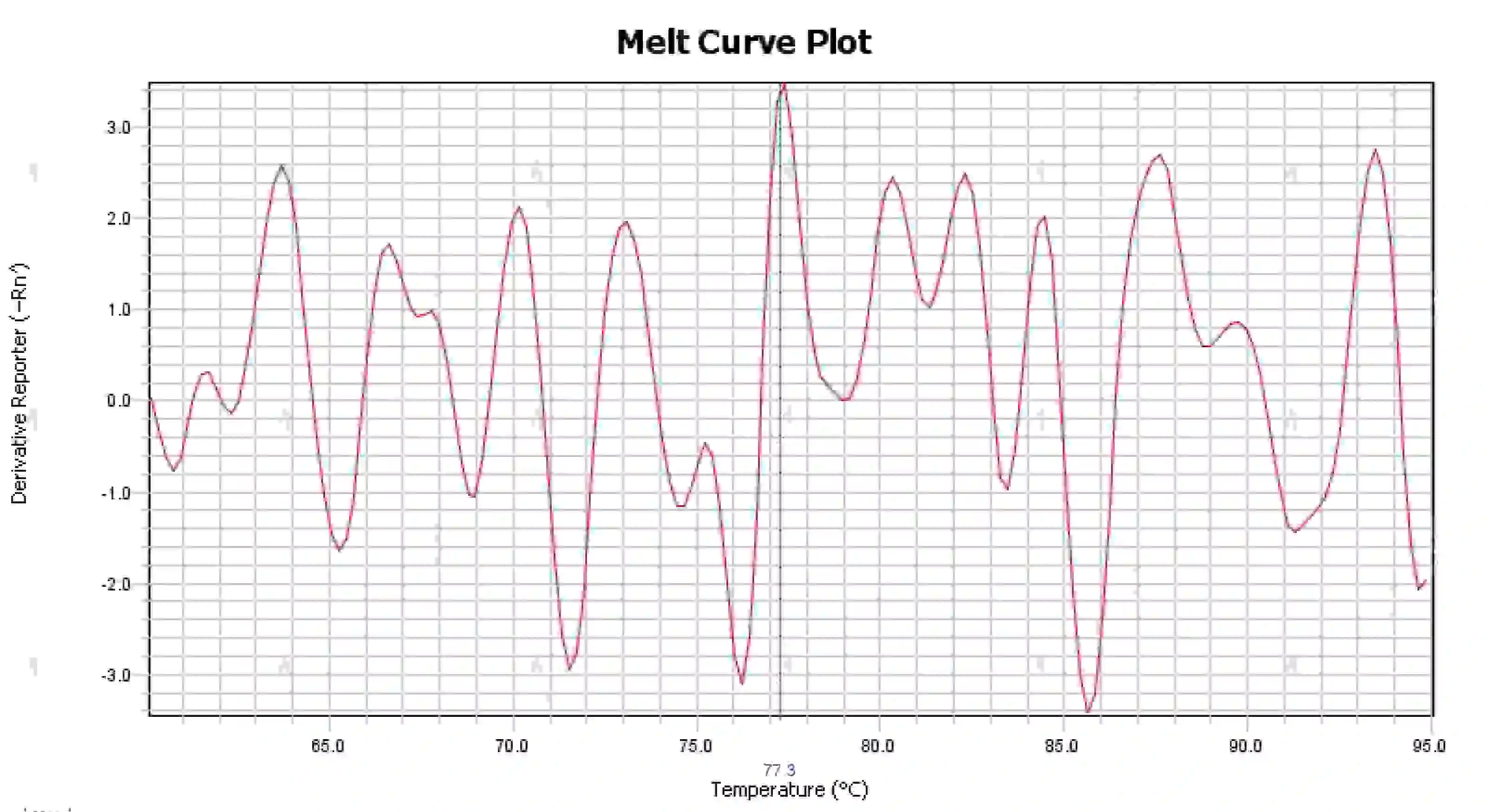
Figura 16. Gráfico da curva de fusão
Possíveis causas:
1) Contaminação do sistema de reação.
2) A exposição dos reagentes à luz forte ou a altas temperaturas leva à falha do reagente.
3) O instrumento não foi calibrado há muito tempo.
4) Incompatibilidade entre consumíveis e instrumentos.
Soluções:
1) Combinando os resultados de NTC e NRC para confirmar a contaminação, é recomendado excluir a contaminação da água, primers, enzimas e do ambiente, um por um.
2) Recomenda-se fazer experimentos de comparação com novos reagentes.
3) Recomenda-se a manutenção regular da calibração do instrumento.
4) Confirme os requisitos do instrumento correspondente para consumíveis.
3.5 Picos espúrios na extremidade frontal da curva de fusão
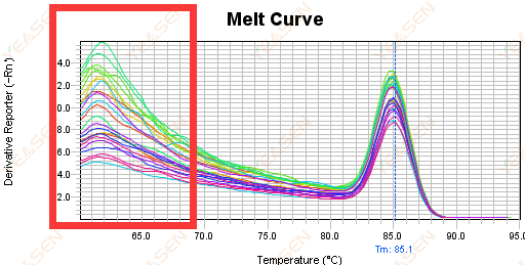
Figura 17. Gráfico da curva de fusão
Possíveis causas:
A concentração de Rox não corresponde ao modelo.
Soluções:
É recomendável cancelar a correção de Rox para verificar se a curva de fusão está normal.
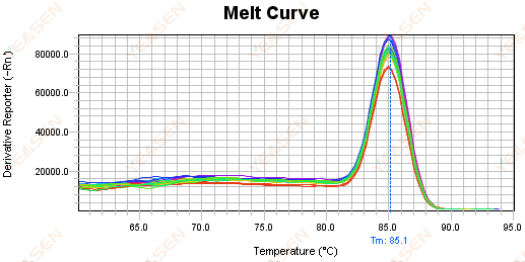
Figura 18. Gráfico da curva de fusão
4. Informações sobre pedidos
Tabela 1. Informações para pedidos
| Nome do produto | Código do produto | Especificação |
| Hieff Unicon™ Universal Blue qPCR Master Mix (baseado em corante) | 11184ES03 | 1 mL |
| 11184ES08 | 5×1 mL | |
| 11184ES50 | 50×1 mL | |
| 11184ES60 | 100×1 mL |
5. Produtos relacionados
Os produtos fornecidos pela
Tabela 2. Produtos relacionados
| Posicionamento do produto | Nome do produto | Código do produto |
| Remoção de gDNA em uma etapa e transcrição reversa | Hifair™V digestão RT-gDNA em uma etapa SuperMix para qPCR (Investigação) | 11142ES |
| Master Mix de alta sensibilidade (com etapa de remoção de gDNA) | Hifair™ III 1ª fita cDNA Synthesis SuperMix para qPCR (digestor de gDNA plus) | 11141ES |
| Kit de alta sensibilidade (com etapa de remoção de gDNA) | Kit de síntese de cDNA Hifair™ III 1ª fita (digestor de gDNA plus) (Investigação) | 11139ES |
| Master mix quantitativo comum (método de sondagem) | Mistura principal da sonda Hieff™ qPCR TaqMan (Investigação) | 11205ES |
6. Sobre a leitura
DNase I e suas aplicações em biomedicina
A Diretriz de Seleção da Transcriptase Reversa
