DNase I e suas aplicações em biomedicina
A desoxirribonuclease I (DNase I) é uma endonuclease, sua aplicação não é apenas para manter a integridade do RNA, mas também para análise de pegada de DNA, geração de bibliotecas de DNA aleatórias, redução de viscosidade em lisados celulares ou extratos de proteína, etc. Em uma palavra, a DNase I pode ser usada em quase qualquer aplicação que exija clivagens enzimáticas de DNA. A seguir, uma introdução detalhada à DNase I e sua aplicação específica.
1. O que é DNase I?
2. DNase I para preparação de extração de RNA livre de DNA
3. DNase I para transcrição in vitro para remover DNA molde
4. DNase I para remoção de rRNA
5. DNase I para marcação de DNA
6. Outras aplicações
7. Guia de seleção de produtos DNase I
1. O que é DNase I?
Desoxirribonuclease I (DNase I) é uma endonuclease não específica que pode digerir DNA fita simples ou dupla, que está presente em diferentes tecidos e fluidos corporais. Pode hidrolisar ligações fosfodiéster para produzir mono e oligodesoxinucleotídeos contendo um grupo 5'-fosfato e um grupo 3'-OH. A faixa de pH de trabalho ideal da DNase I é 7-8, sua atividade depende de Ca2+ e pode ser ativada por íons metálicos divalentes, como Mn2+, Mg2+, Zn2+, etc. Na presença de Mg2+, a DNase I corta aleatoriamente qualquer sítio de DNA fita dupla; na presença de Mn2+, a DNase I pode cortar DNA fita dupla no mesmo sítio para formar uma extremidade romba ou uma extremidade pegajosa de 1-2 nucleotídeos.
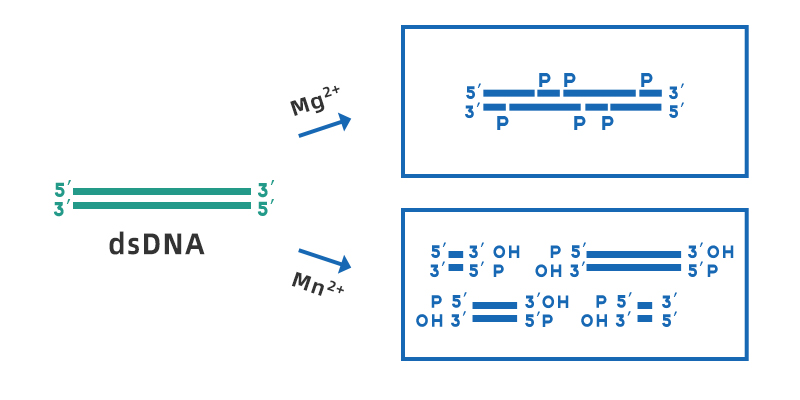
Figura 1. Diagrama esquemático da clivagem do dsDNA pela DNase I na presença de Mg2+ e Mn2+.
Embora as clivagens da DNase I sejam geralmente consideradas clivagens não específicas, a DNase I tem mais probabilidade de atuar em certos fragmentos de sequência, como a região do sulco menor, e é mais propensa a clivagens de sequências de purina-pirimidina. No entanto, quando a DNase I atua em dsDNA heterogêneo, todas as quatro bases serão clivadas, e o efeito em uma base específica não será mais do que 3 vezes maior do que o de outras bases.
2. DNase I para preparação de extração de RNA livre de DNA
Em experimentos biológicos, o primeiro passo é preparar o ácido nucleico para estudar várias funções do RNA. No entanto, como o DNA e o RNA são frequentemente liberados juntos durante o processo de lise celular, a interferência nos dois não pode ser evitada, não importa qual solução de extração seja usada, então enzimas específicas precisam ser usadas para remover a interferência. Para extração de RNA de alta qualidade, a DNase I é usada para remover o DNA residual da amostra.
A DNase I pode degradar DNA fita dupla e fita simples em oligonucleotídeos e nucleotídeos simples, e o DNA no produto de preparação de RNA pode ser efetivamente degradado. A DNase I é então inativada por aquecimento com um tampão de parada. Durante o processo de aquecimento, a estrutura em grampo da molécula de RNA pode ser aberta, o que facilita a entrada direta do RNA no processo de transcrição reversa.
A qualidade do RNA afetará diretamente os dados experimentais em grande extensão. Em geral, resíduos de gDNA não podem ser completamente evitados durante a extração de RNA, portanto, é geralmente recomendado tratar amostras de RNA com DNase I para digerir o gDNA residual antes de desafiar aplicações downstream (por exemplo, análise de expressão de mRNA, análise de transcriptoma, etc.). A etapa de digestão do gDNA pode ser realizada durante a extração de RNA, após a extração de RNA ou antes da transcrição reversa do RNA.De acordo com o posicionamento do produto, os produtos fornecidos pela
Tabela 1: Lista de produtos relacionados à remoção de DNA da extração de RNA ou antes da transcrição reversa
| Posicionamento do produto | Nome do produto | Gato # |
| Extração de RNA | Reagente de extração de RNA total TRIeasy™ [indagar] | 10606ES |
| 19221ES | ||
| Kit MolPure™ Plant Plus RNA [indagar] | 19292ES | |
| Kit de DNA/RNA viral MolPure™ [indagar] | 19321ES | |
| remoção de gDNA | 10325ES | |
| Transcrição reversa | Hifair™Ⅲ1st Strand cDNA Synthesis SuperMix para qPCR (digestor de gDNA plus) | 11141ES |
| qPCR | 11184ES |
3. DNase I para transcrição in vitro para remover DNA molde
A transcrição in vitro (IVT) usa principalmente DNA como molde, mais substratos e tampões correspondentes para obter RNA por meio da transcrição in vitro. Em experimentos de transcrição in vitro, RNA polimerases como T7, T3 e SP6 são comumente usadas para síntese de RNA. O RNA sintetizado pode ter resíduos de DNA. Eliminar os resíduos de DNA é benéfico para o desenvolvimento de experimentos posteriores. Por exemplo, no estágio de desenvolvimento da vacina de mRNA, a remoção de resíduos é uma etapa crítica, que pode reduzir a dificuldade de purificação posterior e aumentar a pureza do produto. O molde de DNA é tipicamente removido usando DNase I recombinante (sem RNase).De acordo com o processo de síntese de mRNA, os produtos fornecidos pela
| Processo de síntese de mRNA | Nome do produto | Gato# |
| Preparação do modelo | Hieff Canace™ Plus DNA polimerase de alta fidelidade [indagar] | 10148ES |
| 10922ES | ||
| 10125ES | ||
| FuniCut™BsaI [indagar] | 15005ES | |
| FuniCut™ XbaI [indagar] | 15033ES | |
| BspQI[indagar] [indagar] | 16215ES | |
| Transcrição in vitro | 10623ES | |
| 10624ES | ||
| T7 RNA polimerase (50 U/μL)[indagar] | 10618ES | |
| 10133ES | ||
| 10620ES | ||
| 10621ES | ||
| Remover DNA modelo | 10611ES | |
| Modificação de mRNA | 10614ES | |
| 10612ES | ||
| 10132ES | ||
| 10619ES | ||
| Purificação de mRNA | 12602ES |
4. DNase I para remoção de rRNA
In vivo, o rRNA é altamente abundante e muito conservador, o que tem pouca importância na obtenção de informações biológicas, por isso o rRNA é frequentemente removido primeiro na construção e sequenciamento da biblioteca de RNA.Atualmente, o método de remoção de rRNA é principalmente a digestão de RNase H. As principais etapas da depleção de rRNA baseada em enzimas são mostradas na figura 2: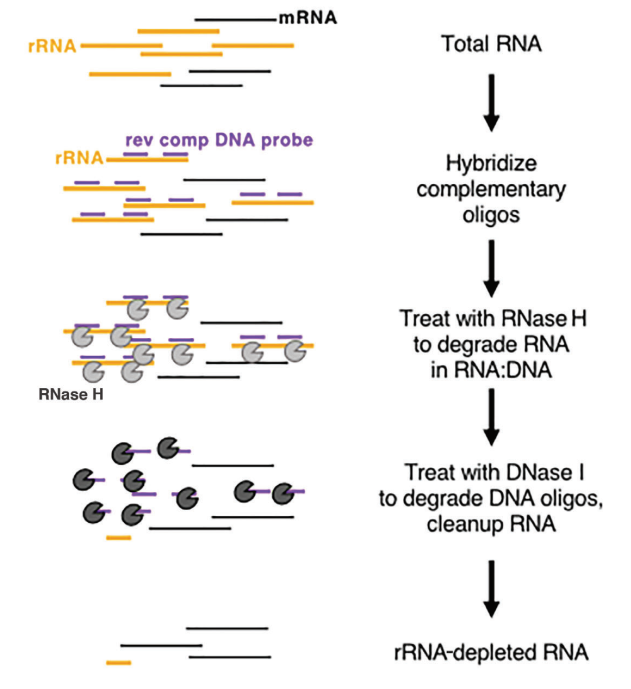
Figura 2: Diagrama esquemático do princípio de depleção de rRNA baseada em enzimas (Baldwin, A. et al. 2021, Current Protocols)
Primeiro, extraia o RNA total, então hibridize a sonda de DNA fita simples com rRNA, projete e sintetize a sonda de DNA fita simples específica para rRNA, então use a RNase H para degradar o rRNA hibridizado, e use a DNase I para degradar a sonda de DNA. Por fim, deixando o molde de RNA não rRNA. Os produtos relacionados à remoção de rRNA fornecidos por
| Processo de síntese de mRNA | Nome do produto | Gato# |
| Humano/Rato/Rato Depleção de rRNA | Hieff NGS™ Kit de depleção de rRNA MaxUp (humano/camundongo/rato) MaxUp [indagar] | 12253ES |
| Depleção de rRNA vegetal | 12254ES | |
| Remoção de RNA ribossômico e regiões 45S ITS/ETS do RNA total humano | Hieff NGS™ Kit de depleção de rRNA humano MaxUp (rRNA e ITS/ETS) | 12257ES |
| Degradação do rRNA | 12906ES | |
| Degradação da sonda de DNA | 10325ES |
5.DNase I para marcação de DNA
A tradução nick é um dos métodos de marcação de sonda de ácido desoxirribonucleico mais comumente usados pelo laboratório. Este método utiliza várias atividades enzimáticas da DNA polimerase I para incorporar desoxirribonucleosídeos trifosfatos marcados em cadeias de DNA recém-sintetizadas. Assim, sondas de DNA uniformemente marcadas para alta atividade específica são sintetizadas. As características da tradução nick são rápidas, simples, deliberadas, de alta especificidade e sondas uniformemente marcadas, que são adequadas para DNA de fita dupla mais longo.O método é realizado pela ação combinada da DNase I e da DNA Polimerase I de E. coli. As principais etapas da marcação de DNA por tradução de nick são mostradas na figura 3:
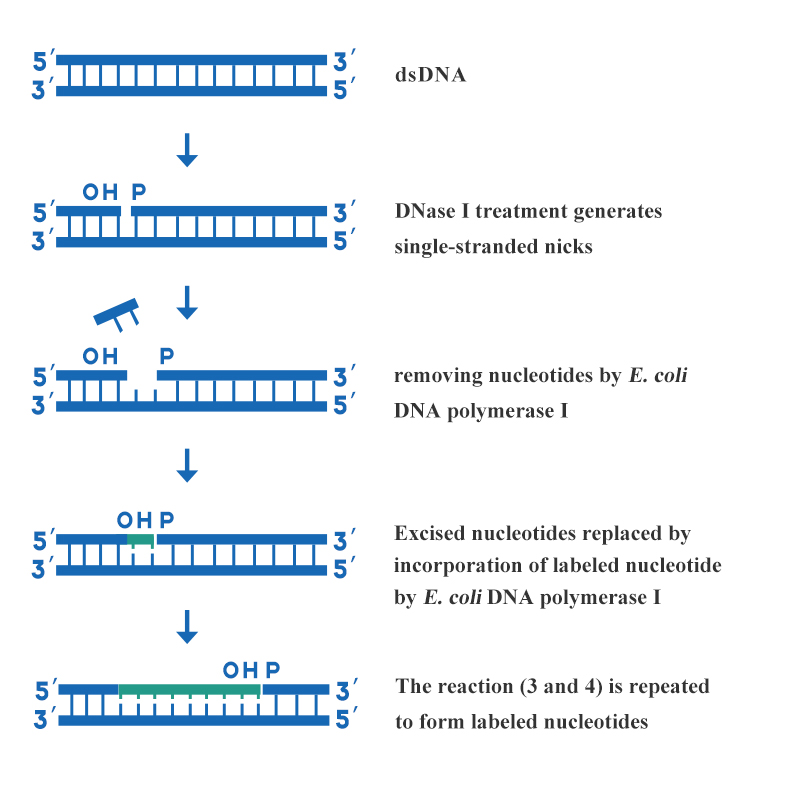
Figura 3: Diagrama esquemático da marcação de DNA por tradução de nick
Uma concentração adequada de DNase I é usada para criar várias lacunas de fita simples em cada fita do DNA fita dupla a ser marcado, e o terminal hidroxila 3' é formado na lacuna. Use a atividade de exonuclease 5'→3' da E. coli DNA Polymerase I para cortar um nucleotídeo da extremidade 5' do nick, e ao mesmo tempo a atividade de polimerase 5'→3' da E. coli DNA Polymerase I introduz um nucleotídeo marcado com a extremidade 3' da lacuna para reparar a lacuna. À medida que a lacuna se move ao longo da fita de DNA, os nucleotídeos marcados são incorporados à fita recém-sintetizada.Os produtos relacionados à rotulagem de DNA fornecidos pela
| Posicionamento do produto | Nome do produto | Gato# |
| Ordinário | Desoxirribonuclease I (DNase I) do pâncreas bovino [indagar] | 10607ES/10608ES |
| RNase livre | 10325ES | |
| E. coli fonte | 12903ES |
6. Outras aplicações
Acima estão várias aplicações comumente usadas. Mais aplicações da DNase I incluem as seguintes, como ensaio de footprinting da DNase I e sítios hipersensíveis à DNase I. O ensaio de footprinting da DNase I é um método de detecção que pode identificar com precisão os sítios de ligação de proteínas de ligação ao DNA no DNA. Quando uma proteína se liga a um fragmento de DNA, ela pode proteger o sítio de ligação de ser danificado pela DNase I e os fragmentos de DNA serão deixados para trás após a digestão enzimática ("footprint"), e sua sequência pode ser determinada. Na imagem do gel, não há bandas onde o DNA se liga à proteína. Para ler mais, clique no link.Sítios hipersensíveis à DNase I referem-se a clivagens em um pequeno número de sítios específicos quando a cromatina é tratada com baixa DNase I e esses sítios específicos são chamados sítios hipersensíveis à DNase I. O princípio é que quando um gene está em um estado transcricionalmente ativo, a cromatina que contém o gene é significativamente mais sensível à degradação da DNase do que a região inativa. Para ler mais, clique no link.
7. Guia de seleção de produtos DNase I
| Nome do produto (Cat#) | Posicionamento do produto | Aplicações recomendadas |
| DNase I do pâncreas bovino (CAT#10607,10608)[indagar] | RNase removida, não detectada | Usado principalmente em pesquisa de proteínas: remoção de DNA de preparações de proteínas. |
| DNase I recombinante (sem RNase)(CAT#10325) | RNase-Free, para pesquisa | Ideal para uma variedade de aplicações: remoção de DNA de preparações de RNA e proteínas, como bibliotecas de cDNA sensíveis à RNase ou preparação de amostras para experimentos de RT-PCR. |
| Sem RNase, grau farmacêutico GMP. | Ideal para uma variedade de aplicações: remoção de DNA de preparações de RNA e proteínas, como bibliotecas de cDNA sensíveis à RNase ou preparação de amostras para experimentos de RT-PCR. |
Sobre a leitura:
Reagentes de grau GMP para síntese in vitro de mRNA
Os princípios da pegada da DNase I e suas aplicações biomédicas
Referências
1. Baldwin A, Morris AR, Mukherjee N. Um método fácil, econômico e escalável para esgotar o RNA ribossômico humano para RNA-seq[J]. Protocolos atuais, 2021.
2. Song C, Zhang S, Huang H. Escolhendo um método adequado para a identificação de origens de replicação em genomas microbianos[J]. Frontiers in Microbiology, 2015, 6:1049.
