Trabalhe em ambas as linhas, a análise da expressão de miRNA ficou mais fácil
O comprimento do miRNA maduro é de apenas cerca de 20nt, geralmente, o primer forward cobrirá seu comprimento total ou até mesmo terá uma parte excedente, e o primer reverse não terá onde ser colocado. A solução atual do mercado é aumentar o comprimento da transcrição reversa durante a transcrição reversa. Os métodos comuns incluem o método tailing e o método stem-loop.
1. Introdução
2. Produtos Recomendados
3. Lista de produtos relacionados
1. Introdução
MicroRNAs (miRNAs) são pequenos RNAs de fita simples com um tamanho de cerca de 21-23 bases, que são gerados a partir de precursores de RNA de fita simples com uma estrutura de grampo de cabelo de cerca de 70-90 bases após o processamento da enzima Dicer. O miRNA se liga principalmente à região não codificadora da extremidade 3' do mRNA do gene alvo e regula o processo de tradução do mRNA do gene alvo pós-transcricional, bloqueando assim a tradução do mRNA e promovendo a degradação do mRNA, resultando na diminuição da expressão da proteína. O papel dos miRNAs está envolvido na ocorrência e no desenvolvimento de várias doenças cardiovasculares, como ontogenia, diferenciação tecidual, proliferação celular e apoptose.
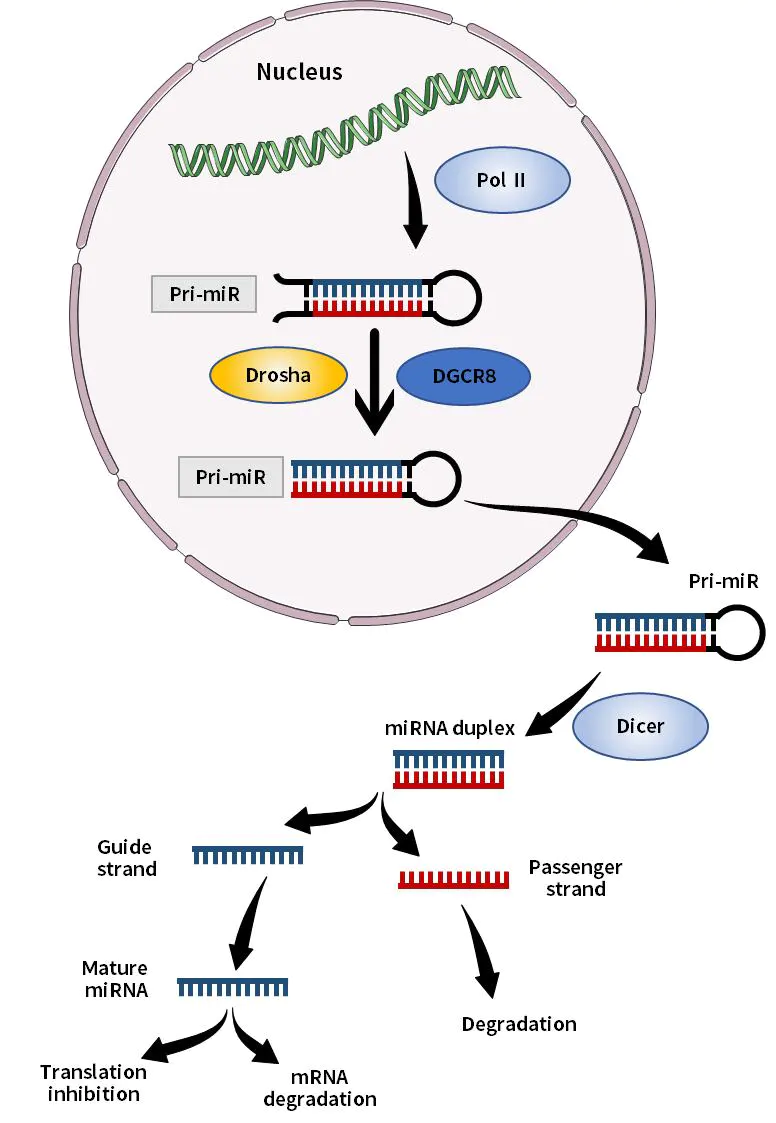
Figura 1. Diagrama esquemático da síntese de miRNA in vivo
O comprimento do miRNA maduro é de apenas cerca de 20nt, geralmente, o primer forward cobrirá seu comprimento total ou até mesmo terá uma parte excedente, e o primer reverse não terá onde ser colocado. A solução atual do mercado é aumentar o comprimento da transcrição reversa durante a transcrição reversa. Os métodos comuns incluem o método tailing e o método stem-loop.
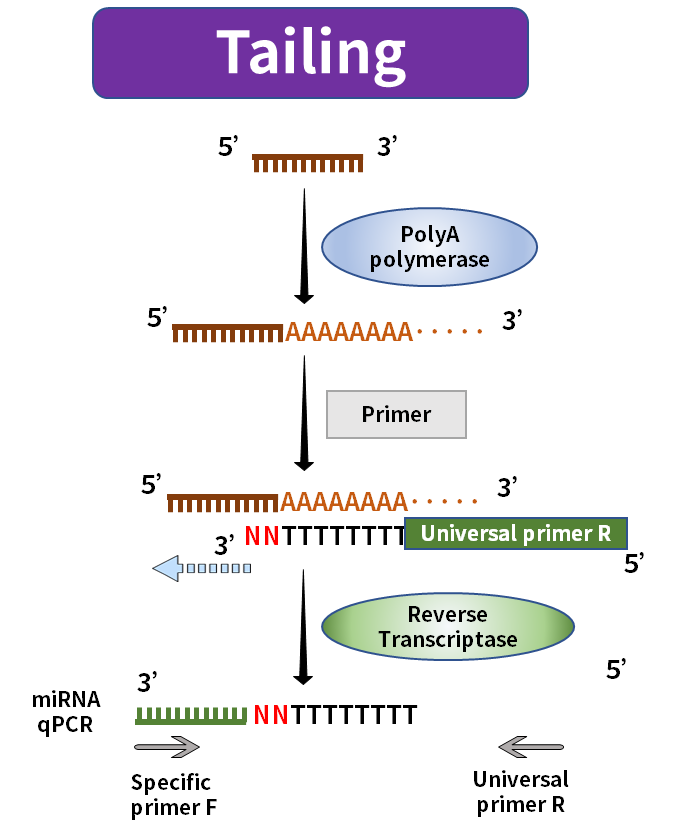
Figura 2. Síntese de miRNA pelo método de cauda
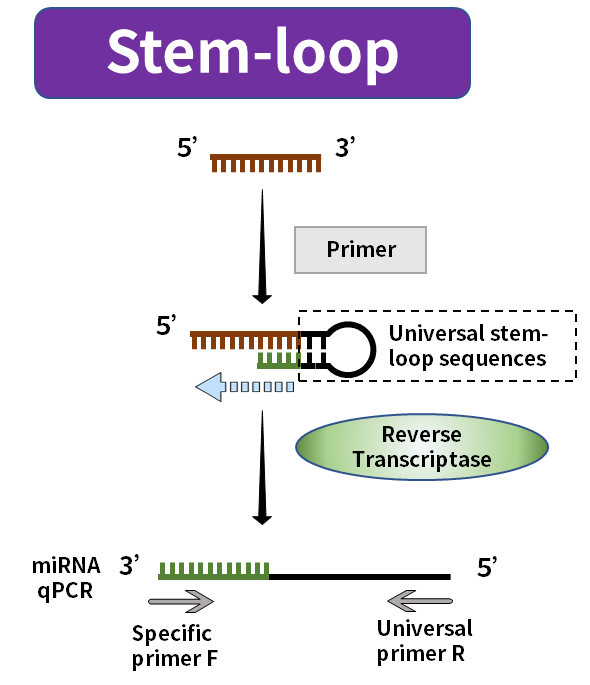
Figura 3. Síntese de miRNA pelo método stem-loop
O método de cauda usa a polimerase PolyA para adicionar a cauda Poly(A) ao miRNA maduro e usa Oligo-dT com a sequência universal como um primer de transcrição reversa para sintetizar o miRNA alongado correspondente à primeira fita do cDNA. O método stem-loop usa a sequência universal dobrada na estrutura stem-loop como um primer para sintetizar a primeira fita do cDNA correspondente ao miRNA alongado. A estrutura stem-loop será aberta a uma temperatura adequada durante o processo de amplificação subsequente e combinados com os primers de amplificação relevantes.
Tabela 1.Comparação de Tailing e Stem-loop
| Categoria | Rejeito | Haste-laço |
| Aplicabilidade | Adequado para estudar vários miRNAs diferentes simultaneamente | Adequado para detecção quantitativa precisa de alguns miRNAs em uma amostra |
| Vantagem | Uma única transcrição reversa pode detectar vários miRNAs com alto rendimento | Apenas um miRNA pode ser detectado por transcrição reversa por vez, com forte especificidade |
| Sistema | Juntamente com o miRNA envolvido, a transcrição reversa no mesmo sistema de reação com alta precisão | Os miRNAs envolvidos internamente são separados e transcritos reversamente nos dois sistemas, o que é propenso a erros |
| Especificidade | ★★★★☆ | ★★★★★ |
| Sensibilidade | ★★★★★ | ★★★★☆ |
| Tempo | ★★★★☆ | ★★★★★ |
2. Produtos Recomendados
2.1 Combinação quantitativa de inversão de cauda de miRNA de Yeasen (11148ES e 11171ES)
Figura 4. Kit de síntese de cDNA de 1ª fita de miRNA 11148ES-Hifair™
O kit adota o método de cauda para transcrição reversa do cDNA da primeira fita do miRNA. O tampão 2×Hifair™ miRNA RT no produto contém todas as matérias-primas e primers para reação de cauda de miRNA e reação de transcrição reversa, e foi cuidadosamente otimizado. Ele pode garantir que os processos de modificação de poli(A) e transcrição reversa da extremidade 3' do miRNA sejam realizados de forma eficiente simultaneamente.
Figura 5. 11171ES -Hieff™ miRNA Universal qPCR SYBR Master Mix
Uma nova geração de reagentes de detecção de PCR quantitativa de fluorescência pré-misturados especialmente desenvolvidos para detecção quantitativa de miRNA, contendo ROX Passive Reference Dye especial, adequado para todos os instrumentos qPCR, sem necessidade de ajustar a concentração de ROX em diferentes instrumentos, apenas na reação de preparação A amplificação pode ser realizada adicionando primers e modelos ao sistema. A DNA polimerase adota polimerase de partida a quente quimicamente modificada e coopera com um sistema Buffer especial, o que torna a especificidade e a sensibilidade da reação mais altas e pode quantificar com precisão em uma faixa mais ampla.
2.2 Desempenho do produto
2.2.1 Compatível com instrumentos qPCR de todas as plataformas

Figura 6. Resultados compatíveis com diferentes instrumentos de qPCR
Usando o RNA total de células 293T como modelo, a transcrição reversa foi realizada com o kit de síntese de cDNA de primeira fita de miRNA Hifair™ (Cat# 11148ES).O cDNA obtido foi diluído 10 vezes e então usado Hieff™ miRNA Universal qPCR SYBR Master Mix (Cat# 11171ES) para detectar os genes de referência internos de hsa-miR-let-7b-5p, hsa-miR-let-7c-5p e U6, respectivamente.
2.2.2 Alta taxa de detecção, até 10 pg
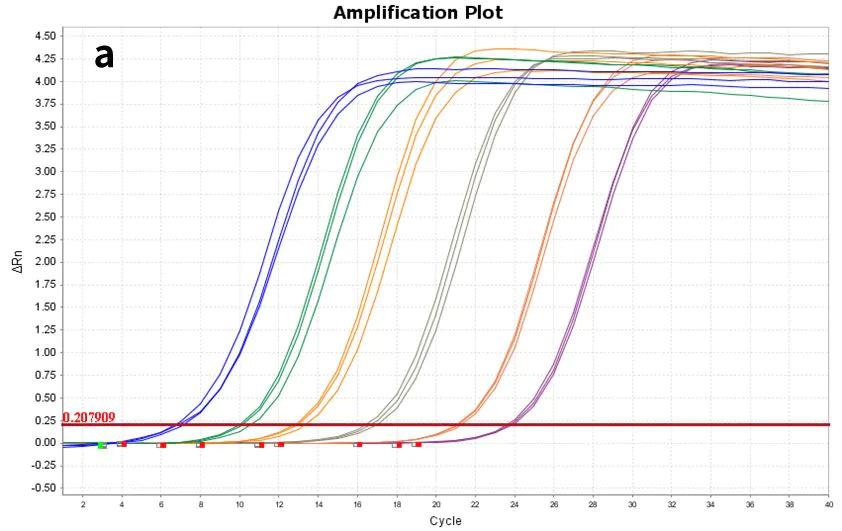
Figura 7. Gráfico de amplificação
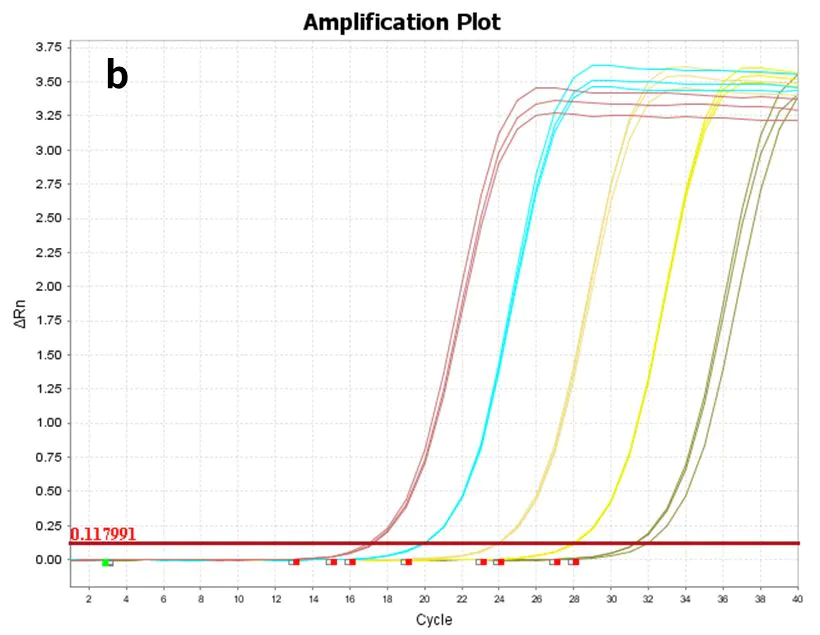
Figura 8. Gráfico de amplificação
Usando hsa-miR-let-7e-5p(a) sintético e RNA total de células 293T(b) como modelos, diluídos nos seguintes gradientes, respectivamente: 60 cópias para 606 cópias (6 gradientes) e 10 pg-100 ng (5 gradientes), transcritos reversamente com o kit de síntese de cDNA de primeira fita de miRNA Hifair™ (Cat# 11148ES), e o cDNA resultante foi detectado com o Hieff™ miRNA Universal qPCR SYBR Master Mix (Cat# 11171ES) para expressão do gene hsa-miR-let-7e -5p.
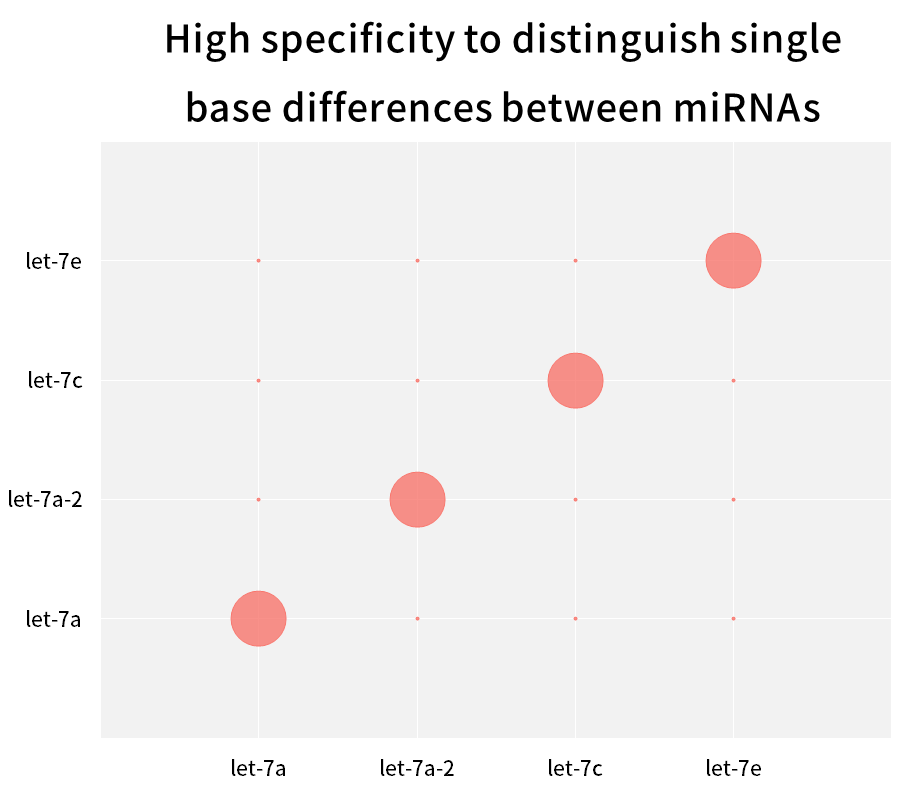
2.2.3 Alta especificidade, capaz de distinguir diferenças de base única entre miRNAs da mesma família
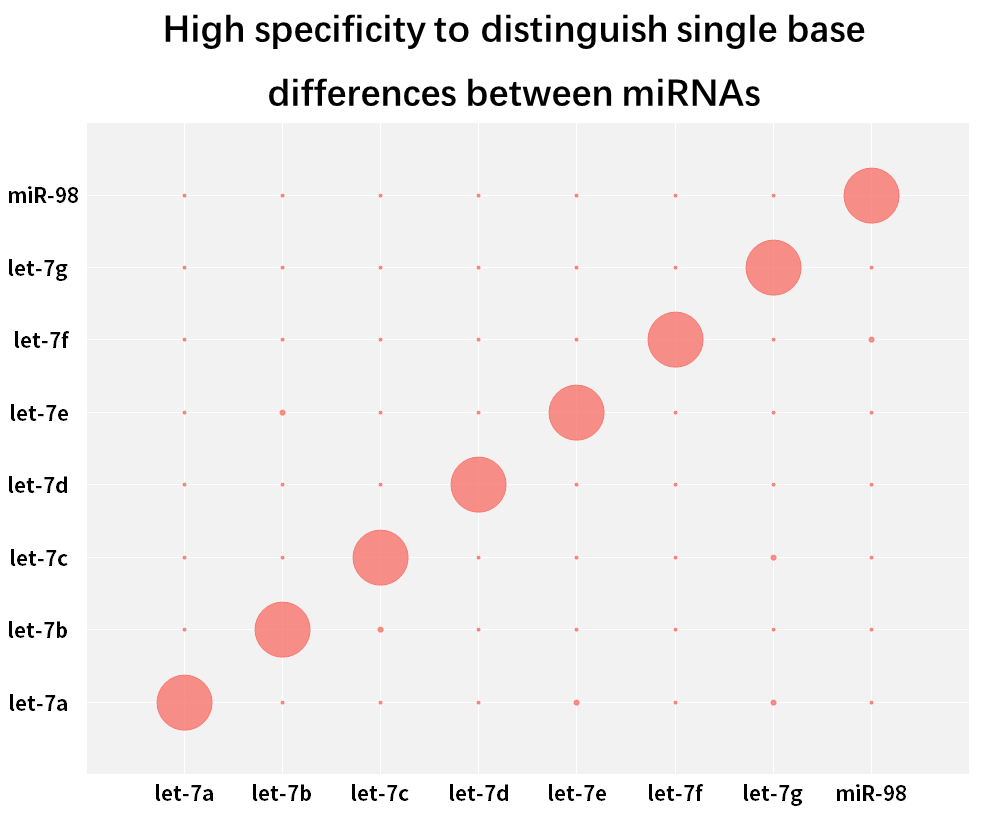
Figura 9. Distinguir diferenças de base única entre miRNAs
A família humana hsa-let-7 tem vários miRNAs, com poucas bases que diferem entre si, ou mesmo apenas uma única diferença de base. Os miRNAs sintéticos para cada isoforma Let-7 (hsa-miR-let-7a~Let-7i, has-miR-98) foram transcritos reversamente em cDNA usando o Hifair™ miRNA 1st Strand cDNA Synthesis Kit (Cat# 11148ES), usando Hieff™ miRNA Universal qPCR SYBR Master Mix (Cat# 11171ES) para amplificar esses miRNAs separadamente com diferentes primers, e calcular a taxa de correspondência usando a Equação 2-ΔCT x 100%. Os resultados mostram que os membros da família de subtipos intimamente relacionados podem ser efetivamente distinguidos.
2.2.4 Excelente eficiência de amplificação
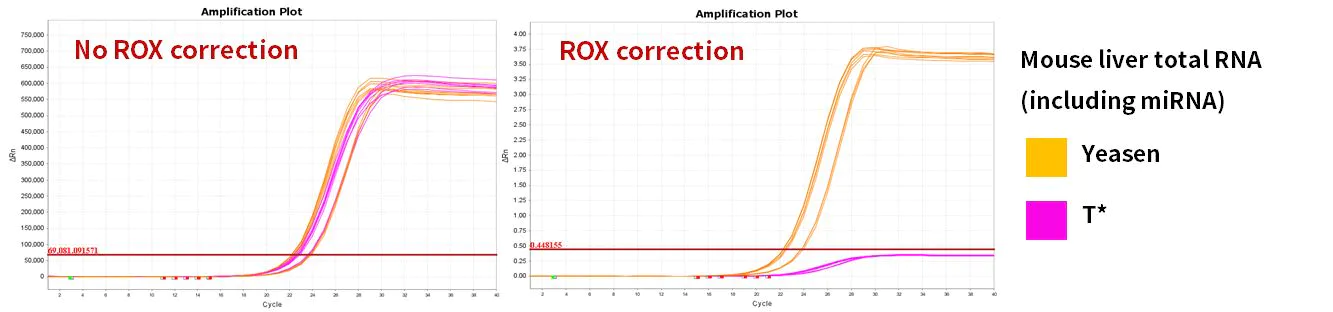
Figura 10. Eficiência de amplificação
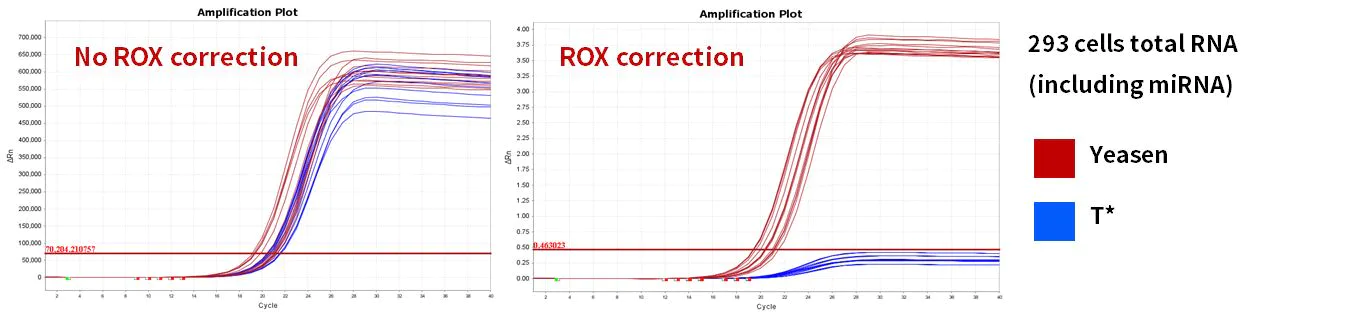
Figura 11. Eficiência de amplificação
Os genes miR-let-7b-5p, miR-let-7c-5p, miR-let-7e-5p, miR-let-7f-5p foram amplificados usando células 293T humanas e RNA total de fígado de camundongo como modelos. Os resultados mostraram que, em comparação com produtos semelhantes, o Hifair™ miRNA 1st Strand cDNA Synthesis Kit (Cat# 11148ES) correspondeu ao sistema de reação do Hieff™ miRNA Universal qPCR SYBR Master Mix (Cat# 11171ES) com excelente desempenho.
2.2.5 Excelente estabilidade

Figura 12. Análise de estabilidade do produto a 37℃ por 7 dias

Figura 13. Análise dos resultados após 50 ciclos de congelamento e descongelamento
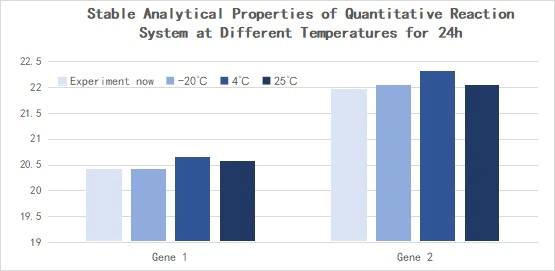
Figura 14. Propriedades analíticas estáveis do sistema de reação quantitativa em diferentes temperaturas por 24h
A transcrição reversa foi realizada com o Hifair™ miRNA 1st Strand cDNA Synthesis Kit (Cat# 11148ES) usando 293T cell Total RNA como molde, e o cDNA obtido foi diluído 10 vezes e detectado com Hieff™ miRNA Universal qPCR SYBR Master Mix (Cat# 11171ES). Expressão de hsa-miR-let-7b-5p, hsa-miR-let-7c-5p, hsa-miR-let-7e-5p. △Ct<0,5.
2.3 Combinação quantitativa de inversão de haste-laço de miRNA de Yeasen (11139ES e 11170ES)
Figura 15. Kit de síntese de cDNA 11139ES-Hifair™ III 1ª fita (digestor de gDNA mais)
O kit foi desenvolvido com base na Transcriptase Reversa Hifair™ III.A velocidade de síntese de cDNA da enzima é rápida, e a estabilidade térmica é muito melhorada. Ao mesmo tempo, a enzima aumenta a afinidade com o molde e é adequada para a transcrição reversa de uma pequena quantidade de moldes e genes de baixa cópia. A capacidade da Hifair™ III Reverse Transcriptase de sintetizar cDNA de comprimento total também foi melhorada, permitindo a síntese de cDNA de até 19,8 kb.

Figura 16. 11170ES-Hieff™ miRNA Universal qPCR SYBR Master Mix
Este produto é uma mistura mestre especial para quantificação de miRNA usando o método de fluorescência quimérica SYBR Green I. Como as sequências de miRNA são curtas e as sequências de miRNA na mesma família são frequentemente muito semelhantes, a especificidade é extremamente necessária durante a quantificação. Este produto é baseado na DNA polimerase de partida a quente, com Buffer otimizado, que pode reduzir muito a amplificação não específica; ao mesmo tempo, o ROX Reference Dye especial torna a mistura mestre adequada para todos os instrumentos qPCR, e não há necessidade de ajustar o ROX em diferentes instrumentos. A amplificação pode ser realizada adicionando primers e modelos ao preparar o sistema de reação.
2.3.1 Compatível com instrumentos qPCR de todas as plataformas
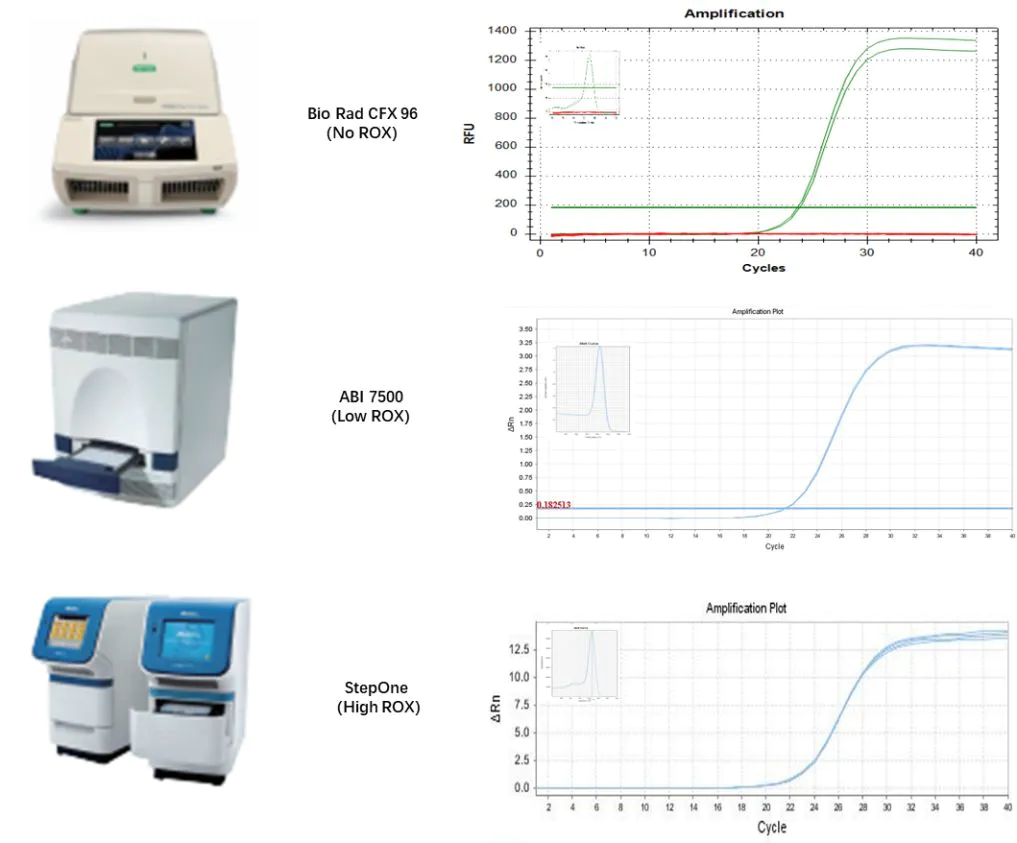
Figura 17. Resultados compatíveis com diferentes instrumentos de qPCR
Utilizando o RNA total de células do fígado de camundongo como modelo, a transcrição reversa foi realizada com Hifair™ III 1st Strand
Kit de síntese de cDNA (gDNA digester plus) (Cat# 11139ES), e o cDNA obtido foi diluído 40 vezes e usado Hieff™ miRNA Universal qPCR SYBR Master Mix (Cat# 11170ES) para amplificar o gene humano mmu-miR-125a, respectivamente.
2.3.2 Alta sensibilidade, até 10 pg

Figura 18. Detecção de sensibilidade
Utilizando o RNA total de células do fígado de camundongo como modelo, a transcrição reversa foi realizada com o kit de síntese de cDNA Hifair™ III 1st Strand (gDNA digester plus) (Cat# 11139ES), e o cDNA obtido foi diluído em série 10 vezes (intervalo 10pg/μL). -0,1 μg/μL), amplificar os genes mmu-miR-125a, mmu-miR-132 e mmu-miR-351 de origem murina.
2.3.3 Alta especificidade, capaz de distinguir diferenças de base única entre miRNAs da mesma família
Figura 19. Distinguir diferenças de base única entre miRNAs
A família humana hsa-let-7 tem vários miRNAs, com poucas bases que diferem entre si, ou mesmo apenas uma única diferença de base. Esses miRNAs foram amplificados separadamente com diferentes primers usando Hieff™ miRNA Universal qPCR SYBR Master Mix (Cat# 11170ES), e a taxa de correspondência foi calculada usando a fórmula 2^-Δct x 100%.
2.3.4 Boa repetibilidade

Figura 20. Detecção de repetibilidade
Usando o RNA total de células 293T humanas como um modelo para transcrição reversa, a transcrição reversa foi realizada com o kit de síntese de cDNA Hifair™ III 1st Strand (gDNA digester plus) (Cat# 11139ES), e o cDNA obtido foi diluído 50 vezes e usado. O Hieff™ miRNA Universal qPCR SYBR Master Mix (Cat# 11170ES) foi usado para amplificar o gene humano hsa-let-7a em um experimento de replicação de 90 poços.
2.3.5 Boa estabilidade

Figura 21.Análise de estabilidade do 11170ES em diferentes temperaturas por 14 dias
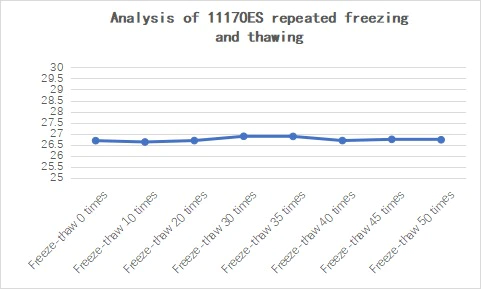
Figura 22. Análise de congelamento e descongelamento repetidos de 11170ES

Figura 23. Propriedades de análise estáveis do sistema de reação quantitativa em diferentes temperaturas por 24h
O RNA total de células de fígado de camundongo foi usado como um molde para transcrição reversa com o Hifair™ III 1st Strand cDNA Synthesis Kit (gDNA digester plus) (Cat# 11139ES). O cDNA obtido foi diluído 100 vezes para amplificar o gene murino mmu-miR-132.
3. Lista de produtos relacionados
Tabela 2. Lista de produtos relacionados
| Tipo de método | Procedimento Experimental | Nome do produto | Código do produto |
| Rejeito | Transcrição reversa | 11148ES | |
| qPCR | 11171ES | ||
| Haste-laço | Transcrição reversa | Hifair™ III Kit de síntese de cDNA de 1ª fita (digestor de gDNA plus) (Investigação) | 11139ES |
| qPCR | Hieff™ miRNA Universal qPCR SYBR Master Mix (Investigação) | 11170ES |