Under de senaste åren har den globala förekomsten av infektionssjukdomar ökat, med patogener som blivit allt mer mångfaldiga och komplexa. En betydande utmaning i detta scenario är att ungefär hälften av patienterna står inför svårigheten att ha okända patogener, vilket gör den snabba diagnosen av dessa patogener till en svår uppgift. För närvarande bygger klinisk patogendetektion huvudsakligen på traditionella odlingsmetoder, PCR-tekniker, metagenomisk sekvensering (mNGS) och patogenriktad sekvensering (tNGS).
Realtidsfluorescerande kvantitativ PCR har blivit framträdande i nukleinsyradetekteringen av SARS-CoV-2. På samma sätt har mNGS fått uppmärksamhet för sina bidrag till kampen mot SARS-CoV-2 och har övergått från kliniska miljöer till daglig användning. tNGS, som erbjuder fördelarna med både PCR och NGS, såsom hastighet, noggrannhet och kostnadseffektivitet i sekvenseringstjänster, vinner allt större dragkraft inom klinisk testning.
Samtidigt har tillämpningen av biologiska produkter expanderat till biomedicin, biologiskt jordbruk, bioenergi, biologisk tillverkning, miljöskydd och andra områden. Eftersom marknadsandelen för biologiska produkter fortsätter att växa har statliga organ och relevanta myndigheter etablerat standardiserade kvalitetsledningssystem och standarder för dessa produkter. Ett särskilt fokusområde hänför sig till närvaron av nukleinsyrarester från värdceller i biologiska produkter.
Biologiska produkter som rekombinanta proteinläkemedel, antikroppsläkemedel, vacciner och cell- och genterapier produceras med användning av på varandra följande stammar eller cellinjer, vilket potentiellt kan resultera i kvarhållande av värdnukleinsyror i slutprodukterna. Kvarvarande värdnukleinsyror kan ha negativa konsekvenser, såsom okontrollerad cellproliferation som leder till tumörbildning eller förvärrar immunsvar genom att introducera virala gener. Därför är effektivt avlägsnande av nukleinsyrarester och rigorös restdetektion avgörande för att säkerställa biologisk produktsäkerhet och uppfylla forskningskrav. Inom industrin används qPCR/RT-qPCR-metoder i stor utsträckning för att utveckla detektionskit för värdnukleinsyraresterdetektering i biologiska produkter.
Dessutom uttrycks kommersiella molekylära enzymer typiskt med användning av rekombinanta tekniska stammar, såsom E. coli, vilket resulterar i närvaron av värdgenomiskt DNA i dessa molekylära enzymer. Dessutom kan miljö- och mänskliga faktorer introducera kontaminerat DNA i molekylära enzymprodukter.
Under patogendetekteringsprocessen kan bakgrundsbakteriella nukleinsyror från kontaminering överskugga målnukleinsyror med låg förekomst eller detekteras tillsammans med målnukleinsyror. Detta kan påverka måldetektionens känslighet eller leda till falskt positiva resultat, vilket komplicerar diagnosen och behandlingsbeslut som tas av medicinsk personal.
Vid utveckling och produktion av kvalitetskontrollprodukter för testning av värdnukleinsyrarester, kan förekomsten av kvarvarande värdnukleinsyror, inklusive sådana från människor, möss, E. coli, jäst och andra, i molekylära enzymer leda till felaktigheter i kvantifieringen av värdnukleinsyraresternas kvalitetskontrollprodukter. Detta innebär potentiella säkerhetsrisker vid tillverkning av biologiska produkter.
YEASEN UCF.METM molekylär enzymlösning med ultralåg resthalt
För att lösa problemet med bakgrundsbakterier och värdnukleinsyrarestinterferens, YEASEN har etablerat en komplett FoU- och produktionsplattform för UCF.METM molekylära enzymer med ultralåg resthalt. Och har uppnått storskalig produktion av en mängd olika UCF.MIGTM molekylära enzymer med ultralåg resthalt genom materialval, miljökontroll, processoptimering och kvalitetssäkring.
YEASEN UCF.METM molekylära enzymprodukter med ultralåg resthalt
YEASEN reformerade en komplett uppsättning molekylära enzymer såsom qPCR/RT-qPCR, NGS-biblioteksbyggnad. Samtidigt, UCF.ME™ process med ultralåg resthalt används för att bearbeta molekylära enzymprodukter, vi kan tillhandahålla en komplett uppsättning högpresterande och ultralåg värdrester molekylära enzymråmaterial för qPCR/RT-qPCR och NGS till förbättra detekteringsnoggrannheten.
Tabell 1 Listan över YEASEN UCF.METM molekylära enzymprodukter med ultralåg resthalt
| Produktklassificering | Produktnamn | Katalog nr. | Kriterier för E. coli gDNA kvalitetskontroll |
| UCF.METM enzymprodukter med ultralåg resthalt för qPCR/RT-qPCR | Hieff UCF.METM Hotstart Sensitive Taq DNA Polymerase (5 U/μL) | 14314ES | <0,005 exemplar/U |
| Hifair UCF.METM V omvänt transkriptas (200 U/μL) | 14608ES | <0,005 exemplar/U | |
| UCF.METM Murin RNas-hämmare (40 U/μL) | 14672ES | <0.001 exemplar/U | |
| UCF.METM RNas-hämmare med hög affinitet (40 U/μL) | 14675ES | <0.001 exemplar/U | |
| UCF.METM Uracil DNA Glycosylas(UDG/UNG), värmelabilt, 1 U/μL | 14466ES | <0,1 exemplar/U | |
| UCF.METM Uracil DNA Glycosylas (UDG/UNG), 1 U/μL | 14454ES | <0,1 exemplar/U |
Partiell datapresentation (UCF.METM Hotstart Sensitive Taq DNA Polymerase som ett exempel)
- Råterstodav coli gDNA <0.005 exemplar/U
- coligDNA-rester från olika satser av UCF.METMTaq-enzym (Cat#14314ES) detekterades. Resultatet visade det E. coli gDNA-rester rester av Taq DNA-polymeras låg långt under 0,005 exemplar/U.
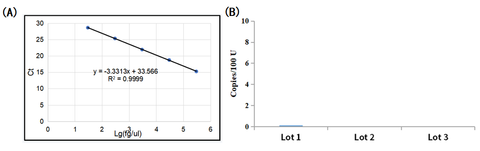
Figur 1: Detektering av E.coli gDNA-rest av UCF.METM Taq-enzym (Katt#14314ES)
- Inget kvarvarande plasmid-DNA detekterades
Plasmid-DNA-rester av UCF.METM Taq-enzym (Cat#14314ES) och Taq DNA-polymeras av märke A detekterades. Resultaten visade att det fanns en plasmid-DNA-rest i Taq DNA-polymeras av märke A. Inget plasmid-DNA detekterades i UCF.METM Taq-enzym (Katt#14314ES).
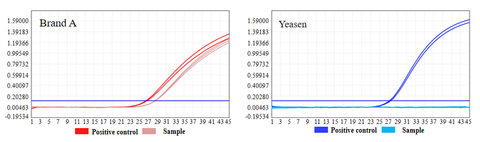
Figur 2: Resultat av detektering av plasmid-DNA-rester
- Elva typer av vanliga bakgrundsbakterier upptäcktes inte
Använd UCF.METM Taq-enzym (Cat#14314ES) kopplat med primers och prober av Pseudomonas aeruginosa, Acinetobacter baumannii, Serratia marcescens, Klebsiella pneumoniae, Stenotrophomonas maltofil, Streptococcus pneumoniae, Enterococcus faecium, Staphylococcus aureus, E. coli, Enterococcus faecalis för att förbereda qPCR-premix. NTC (No Template Control) upptäcktes, resultaten visade att inga rester av ovanstående 11 vanliga bakgrundsbakterier upptäcktes i UCF.METM Taq-enzym (Katt#14314ES).
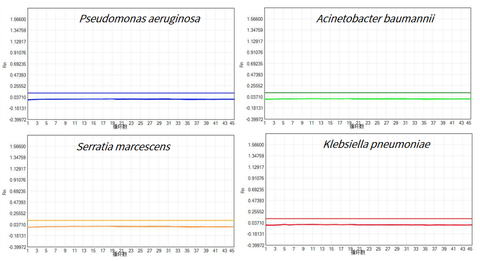
Figur 3: Testresultat av 11 vanliga bakgrundsbakterier (begränsat av utrymme, Pseudomonas aeruginosa, Acinetobacter baumannii, Serratia marcescens och Klebsiella pneumoniae visas som exempel)
- Kundens testfallspresentation
Det kommersiella Taq-enzymet och YEASEN UCF.METM Taq-enzym (Cat#14314ES) användes för att detektera NTC tillsammans med kundens E. coli specifik primer, och resultaten visade att det kommersiella Taq-enzymet toppade 5 gånger i 22 upprepade experiment. UCF.METM Taq-enzym (Cat#14314ES) toppade inte i 22 replikat, vilket indikerar att UCF.METM Taq-enzym (Cat#14314ES) detekterade inte ut E. coli gDNA.
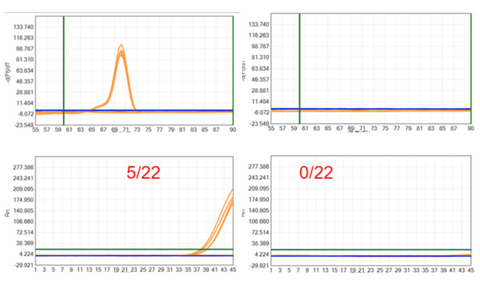
Figur 4: Detektion resultat av E.coli gDNA-rest (kundens testfall)
Vänster: Kommersiellt tillgängligt konventionellt Taq-enzym; Höger: UCF.METM Taq-enzym (Katt#14314ES)
Relaterad produktrekommendation
| Produktklassificering | Produktnamn | Katalog nr. |
| Värdnukleinsyrarester detektionskit | 41332ES | |
| 41331ES | ||
| 41307ES | ||
| 41308ES | ||
| Hansenula polymorpha värdcells-DNA-restdetektionskit | 41317ES | |
| E.coli Värdcell RNA-restdetektionskit | 41318ES |
