I de senere år har den globale forekomst af infektionssygdomme været stigende, hvor patogener er blevet stadig mere forskelligartede og komplekse. En væsentlig udfordring i dette scenarie er, at omkring halvdelen af patienterne står over for den vanskelige situation at have ukendte patogener, hvilket gør den hurtige diagnose af disse patogener til en skræmmende opgave. I øjeblikket bygger klinisk patogendetektion hovedsageligt på traditionelle dyrkningsmetoder, PCR-teknikker, metagenomisk sekventering (mNGS) og patogenmålrettet sekventering (tNGS).
Real-time fluorescerende kvantitativ PCR har vundet fremtræden i nukleinsyredetektionen af SARS-CoV-2. På samme måde har mNGS fået opmærksomhed for sine bidrag til kampen mod SARS-CoV-2 og er gået fra kliniske omgivelser til daglig brug. tNGS, der tilbyder fordelene ved både PCR og NGS, såsom hastighed, nøjagtighed og omkostningseffektivitet i sekventeringstjenester, vinder stadig mere indpas inden for klinisk test.
Samtidig er anvendelsen af biologiske produkter udvidet til biomedicin, biologisk landbrug, bioenergi, biologisk fremstilling, miljøbeskyttelse og andre domæner. I takt med at markedsandelen for biologiske produkter fortsætter med at vokse, har statslige organer og relevante myndigheder etableret standardiserede kvalitetsstyringssystemer og standarder for disse produkter. Et særligt fokusområde vedrører tilstedeværelsen af nukleinsyrerester fra værtsceller i biologiske produkter.
Biologiske produkter som rekombinante proteinlægemidler, antistoflægemidler, vacciner og celle- og genterapier produceres ved hjælp af på hinanden følgende stammer eller cellelinjer, hvilket potentielt resulterer i tilbageholdelse af værtsnukleinsyrer i slutprodukterne. Resterende værtsnukleinsyrer kan have negative konsekvenser, såsom ukontrolleret celleproliferation, der fører til tumordannelse eller forværring af immunresponser ved at introducere virale gener. Effektiv fjernelse af nukleinsyrerester og streng restpåvisning er derfor afgørende for at sikre biologisk produktsikkerhed og opfylde forskningskrav. I industrien anvendes qPCR/RT-qPCR-metoder i vid udstrækning til at udvikle detektionskit til påvisning af værtsnukleinsyrerester i biologiske produkter.
Endvidere udtrykkes kommercielle molekylære enzymer typisk under anvendelse af rekombinante ingeniørstammer, såsom E. coli, hvilket resulterer i tilstedeværelsen af værtsgenomisk DNA i disse molekylære enzymer. Derudover kan miljømæssige og menneskelige faktorer introducere forurenet DNA i molekylære enzymprodukter.
Under patogendetektionsprocessen kan bakterielle nukleinsyrer i baggrunden fra kontaminering overskygge målnukleinsyrer med lav mængde eller blive påvist sammen med målnukleinsyrer. Dette kan påvirke følsomheden af måldetektion eller føre til falske positive resultater, hvilket komplicerer diagnose og behandlingsbeslutninger truffet af læger.
Ved udvikling og produktion af kvalitetskontrolprodukter til test af værtsnukleinsyrerester kan tilstedeværelsen af resterende værtsnukleinsyrer, herunder dem fra mennesker, mus, E. coli, gær og andre, i molekylære enzymer føre til unøjagtigheder i kvantificeringen af kvalitetskontrolprodukter for værtsnukleinsyrerester. Dette udgør potentielle sikkerhedsrisici ved fremstilling af biologiske produkter.
YEASEN UCF.METM ultra-lav restmolekylær enzymopløsning
For at løse problemet med baggrundsbakterier og værtsnukleinsyrerestinterferens, YEASEN har etableret en komplet R&D og produktionsplatform for UCF.METM molekylære enzymer med ultralav restkoncentration. Og har opnået storstilet produktion af en række UCF.MIGTM molekylære enzymer med ultralav restkoncentration gennem materialevalg, miljøkontrol, procesoptimering og kvalitetssikring.
YEASEN UCF.METM molekylære enzymprodukter med ultralav restkoncentration
YEASEN reformerede et komplet sæt af molekylære enzymer såsom qPCR/RT-qPCR, NGS biblioteksbygning. Samtidig er UCF.ME™ proces med ultra-lav restværdi bruges til at behandle molekylære enzymprodukter, vi kan levere et komplet sæt af højtydende og ultra-lave værtsrester molekylære enzymråmaterialer til qPCR/RT-qPCR og NGS til forbedre detektionsnøjagtigheden.
Tabel 1 Listen over YEASEN UCF.METM molekylære enzymprodukter med ultralav restkoncentration
| Produktklassificering | Produktnavn | Katalog nr. | Kriterier vedr E. coli gDNA kvalitetskontrol |
| UCF.METM enzymprodukter med ultralav restkoncentration til qPCR/RT-qPCR | Hieff UCF.METM Hotstart Sensitive Taq DNA Polymerase (5 U/μL) | 14314ES | <0.005 eksemplarer/U |
| Hifair UCF.METM V omvendt transkriptase (200 U/μL) | 14608ES | <0,005 kopier/U | |
| UCF.METM Murin RNase-hæmmer (40 U/μL) | 14672ES | <0.001 eksemplarer/U | |
| UCF.METM Højaffinitet RNase-hæmmer (40 U/μL) | 14675ES | <0.001 eksemplarer/U | |
| UCF.METM Uracil DNA Glycosylase(UDG/UNG), varmelabil, 1 U/μL | 14466ES | <0,1 eksemplarer/U | |
| UCF.METM Uracil DNA Glycosylase (UDG/UNG), 1 U/μL | 14454ES | <0,1 eksemplarer/U |
Delvis datapræsentation (UCF.METM Hotstart Sensitive Taq DNA Polymerase som et eksempel)
- Rrestaf coli gDNA <0.005 eksemplarer/U
- coligDNA-rester af forskellige batches af UCF.METMTaq-enzym (kat#14314ES) blev påvist. Det viste resultatet E. coli gDNA-rester rester af Taq DNA-polymerase var et godt stykke under 0,005 kopier/U.
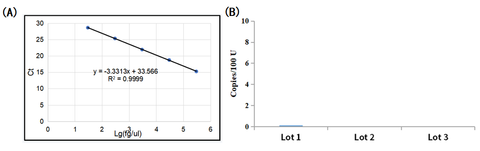
Figur 1: Påvisning af E. coli gDNA-rest af UCF.METM Taq-enzym (kat#14314ES)
- Intet resterende plasmid-DNA blev påvist
Plasmid-DNA-rester af UCF.METM Taq-enzym (Cat#14314ES) og Taq DNA-polymerase af mærke A blev påvist. Resultaterne viste, at der var plasmid-DNA-rest i Taq DNA-polymerase af mærke A. Intet plasmid-DNA blev påvist i UCF.METM Taq-enzym (kat#14314ES).
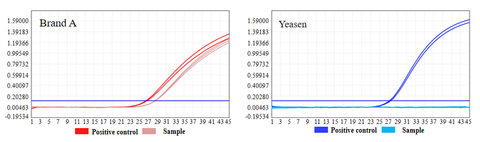
Figur 2: Resultater af plasmid-DNA-restpåvisning
- Elleve slags almindelige baggrundsbakterier blev ikke påvist
Brug UCF.METM Taq-enzym (Cat#14314ES) koblet med primere og prober af Pseudomonas aeruginosa, Acinetobacter baumannii, Serratia marcescens, Klebsiella pneumoniae, Stenotrophomonas maltophil, Streptococcus pneumoniae, Enterococcus faecium, Staphylococcus aureus, E. coli, Enterococcus faecalis at forberede qPCR-premix. NTC (No Template Control) blev detekteret, resultaterne viste, at ingen rest af de ovennævnte 11 almindelige baggrundsbakterier blev påvist i UCF.METM Taq-enzym (kat#14314ES).
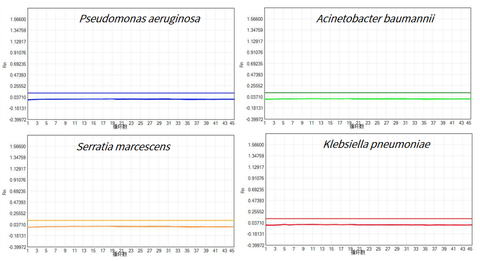
Figur 3: Testresultater af 11 almindelige baggrundsbakterier (begrænset af plads, Pseudomonas aeruginosa, Acinetobacter baumannii, Serratia marcescens og Klebsiella pneumoniae er vist som eksempler)
- Kundens testcasepræsentation
Det kommercielle Taq-enzym og YEASEN UCF.METM Taq-enzym (Cat#14314ES) blev brugt til at detektere NTC sammen med kundens E. coli specifik primer, og resultaterne viste, at det kommercielle Taq-enzym toppede 5 gange i 22 gentagne forsøg. UCF.METM Taq-enzym (Cat#14314ES) toppede ikke i 22 replikater, hvilket indikerer, at UCF.METM Taq-enzym (kat#14314ES) opdagede ikke ud E. coli gDNA.
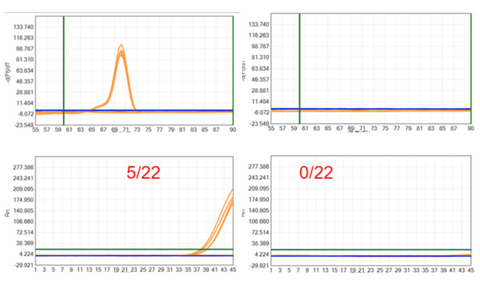
Figur 4: Detektion resultat af E.coli gDNA-rest (kundens testcase)
Til venstre: Kommercielt tilgængeligt konventionelt Taq-enzym; Til højre: UCF.METM Taq-enzym (kat#14314ES)
Relateret produktanbefaling
| Produktklassificering | Produktnavn | Katalog nr. |
| Værtsnukleinsyrerester detektionskit | 41332ES | |
| 41331ES | ||
| 41307ES | ||
| 41308ES | ||
| Hansenula polymorpha Værtscelle DNA-restdetektionskit | 41317ES | |
| E. coli Værtscelle-RNA-restdetektionskit | 41318ES |
