PCR-Methodik für den genauen Nachweis von Punktmutationen – ARMS
Mit dem Fortschreiten der medizinischen Forschung ist die zielgerichtete Therapie ausgereifter geworden. Die Prämisse der zielgerichteten Behandlung besteht darin, genetische Tests an den molekularen Zielen von Medikamenten durchzuführen, um Genmutationen zu identifizieren, die Erbkrankheiten oder bösartige Erkrankungen verursachen. ARMS-PCR ist eine neuartige Methode auf PCR-Basis, mit der zahlreiche DNA-Punktmutationen nachgewiesen werden können. Derzeit ist sie eine der wichtigsten und am weitesten verbreiteten Methoden zur maßgeschneiderten genetischen Identifizierung von Krebserkrankungen. Die Vorteile ihres therapeutischen Einsatzes werden von Fachexperten anerkannt.
1. Wie funktioniert die allelspezifische PCR?
2. Wie kann die ARMS-Spezifität verbessert werden?
3. Welche Merkmale haben die von
4. Was sind Produktleistungsdaten?
5. Wie bestellen Leute Produkte?
1. Wie funktioniert die allelspezifische PCR?
ARMS-PCR ist eine Amplification Refractory Mutation System PCR (ARMS-PCR), auch bekannt als Allel-Specific PCR (AS-PCR). Die allelspezifische Verlängerung wird reguliert, um das Wildtyp-Allel und das mutierte Wildtyp-Gen zu identifizieren, und der Fluoreszenzsignalwert wird mithilfe der Taqman-Sondentechnik gemessen.
ARMS-PCR-Primer sind mit unterschiedlichen Nukleotiden am 3'-Ende der beiden Upstream-Primer des Allels ausgestattet, und die beiden Primer sind jeweils spezifisch für den Wildtyp und den Mutantentyp. Während der Amplifikation können Upstream-Primer, die nicht vollständig mit der Vorlage übereinstimmen, keine komplementären Basenpaare bilden, was zu Fehlpaarungen, behinderter Verlängerung und der Unfähigkeit zur Erzeugung von PCR-Produkten führt, wohingegen Primersysteme, die mit der Vorlage übereinstimmen, die entsprechenden PCR-Produkte amplifizieren können. Der Fluorophor auf der Taqman-Sonde kann ein Fluoreszenzsignal aussenden, das vom Gerät erkannt werden kann, und der Genotyp kann durch Auswertung der Fluoreszenzdaten validiert werden.
Die ARMS-Technologie ist hochempfindlich, die Nachweisgrenze kann 100 Kopien/ml erreichen und die Nachweisgrenze für Tumorgewebe kann eine Mutationsrate von 0,5 % erreichen. Darüber hinaus ist ARMS zeitaufwändig und kostengünstig und die Ergebnisse sind intuitiv und leicht zu beurteilen. Dies liegt daran, dass ARMS in Kombination mit qPCR oder Elektrophorese-Technologie nur eine PCR-Reaktion erfordert, was weniger Zeit in Anspruch nimmt. Nur Upstream-Primer müssen für die ARMS-Technologie optimiert werden, die im Vergleich zur Echtzeit-PCR-Technologie mit MGB-Sonden kostengünstiger ist und intuitive Ergebnisse liefert. Da der Großteil der aus in Paraffin eingebetteten Gewebeproben extrahierten DNA fragmentiert ist, können keine genauen Nachweisergebnisse erzielt werden. Die ARMS-Methode ist darauf ausgelegt, die Länge des Zielprodukts zu minimieren und so dieses Problem bei in Paraffin eingebettetem Gewebe zu lösen. Gleichzeitig realisiert das mit der PCR-Plattform kombinierte ARMS einen geschlossenen Röhrchenbetrieb, der einfach zu handhaben ist und keine Nachbearbeitung des Produkts erfordert, wodurch die Kontamination von Amplifikationsprodukten weitgehend vermieden wird.
Theoretisch muss die Taq-DNA-Polymerase vollständig komplementär zur verbleibenden Vorlage am 3'-Ende des Primers sein, um eine effiziente Polymerisation durchzuführen. Die Stringenz der Taq-DNA wird jedoch von mehreren Faktoren beeinflusst. In einigen Fällen kann eine Verlängerung erfolgen, auch wenn die 3'-terminale Base des Primers nicht komplementär zur Vorlage ist. Wenn am 3'-Ende nur eine Basenfehlpaarung vorliegt, können die Primer fehlgepaart und verlängert werden, aber die Verlängerungseffizienz ist gering. Verschiedene 3'-Endverlagerungen haben unterschiedliche Verlängerungseffizienzen. Wenn am 3'-Ende andere fehlgepaarte Basen eingeführt werden, ist die Anzahl der Fehlpaarungen zu groß und das 3'-Ende kann über ein bestimmtes Niveau hinaus nicht mehr verlängert werden.Daher reicht eine einzige Basenfehlpaarung am 3'-Ende des Primers nicht aus, um die beiden Allele ausreichend zu unterscheiden, was zu falsch positiven Ergebnissen führt.
2. Wie kann die ARMS-Spezifität verbessert werden?
Der Schwerpunkt der Verbesserung der ARMS-Spezifität liegt auf der Verbesserung der Primer-Extension-Spezifität. Eine nicht übereinstimmende Base kann an der 2. oder 3. Base vom 3'-Ende aus eingeführt werden. Die nicht übereinstimmenden Basen wirken mit den nicht übereinstimmenden Basen am 3'-Ende zusammen, und in der Vorlage, die nicht komplementär zum 3'-Ende ist, wird die Primer-Amplifikationsproduktrate reduziert. Während Primer in Vorlagen, die komplementär zu ihren 3'-Enden sind, normal amplifizieren, hängt die Art der nicht übereinstimmenden Basen der Primer von der Art der Basenfehlpaarung am 3'-Ende ab.
Nachdem die spezifischen Primer für ARMS entwickelt wurden, wird ein Paar TaqMan-Sonden für allelspezifischen Fluoreszenzresonanzenergietransfer benötigt. TaqMan-Sonden haben zwei Fluoreszenzfarbstoffe, das 5'-Ende ist ein Fluoreszenzgen und das 3'-Ende hat ein allgemeines Fluoreszenzlöschungsgen. In einer vollständigen Sonde liegen das Löschergen und das fluorogene Gen räumlich sehr nahe beieinander, was zu einer Fluoreszenzlöschung führt. Unter normalen Umständen kann die vom Fluorophor am 5'-Ende emittierte Fluoreszenz nicht erkannt werden, und nur die Hintergrundfluoreszenz des Löschers am 3'-Ende kann erkannt werden. Im Prozess der Zielgenamplifikation sind sowohl PCR-Primer als auch fluoreszenzmarkierte Sonden während der De-OR komplementär zur Zielsequenz. Wenn das Taq-Enzym beim Verlängern des Matrizenstrangs auf eine Sonde trifft, die stabil an die Matrizen gebunden ist, wird die 5'-3'-Exonuklease des Taq-Enzyms die an die Matrizen gebundene spezifische Sonde abbauen. Der Fluorophor auf der Sonde wird durch physischen Raum getrennt, der Löscheffekt verschwindet und Fluoreszenz wird emittiert. Fehlpaarungen zwischen schwach fluoreszierenden Sonden und Zielsequenzen verringern die Menge der freigesetzten Fluoreszenz.
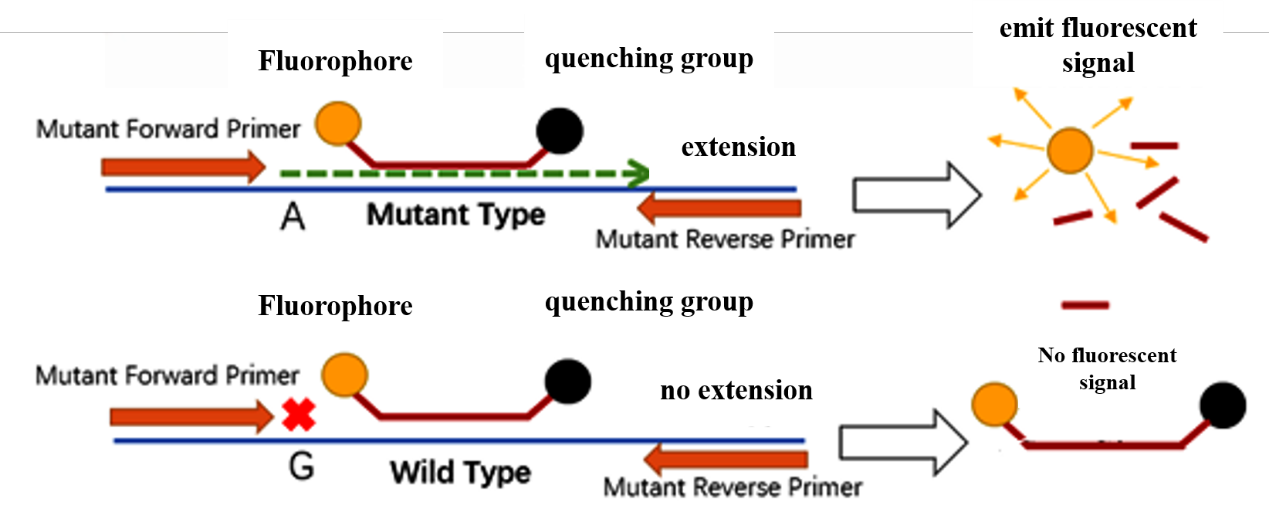
Abbildung 1 Grundlegendes Konzept der ARMS-PCR
3. Welche Merkmale haben die von Yeasen angebotenen Produkte?
YEASEN Hieff Unicon™ Multiplex ARMS qPCR Mix ist ein Kit basierend auf dem Amplifikationsblockkonzept, das eine effiziente ARMS-PCR-vermittelte Genotypisierung ermöglicht.
👍Hohe Selektivität: kann mehrere Genmutationen identifizieren;
👍Hohe Empfindlichkeit: Mutantenprimer können Mutationen bei Konzentrationen von nur 0,5 % in 10 ng/l DNA erkennen;
👍Einfach zu lesen: Die Ergebnisse sind offensichtlich und objektiv beurteilt, und es ist unkompliziert und unkompliziert;
👍Einfach zu bedienen: Es ist mit einer Reihe von PCR-Geräten kompatibel, die Funktionsweise ist standardisiert und die On-Board-Erkennung kann in 90 Minuten durchgeführt werden.
4. Was sind Produktleistungsdaten?
4.1 Überlegene Produktverstopfungswirkung
Experiment: 5L Template-Input, 10ng/L Wildtyp-Template und äquivalente Mengen mutierten Plasmids wurden mit mutierten Primern amplifiziert.
Standort: KRASG13D (38G>A)
Rot: 13755; Blau: Wettbewerber
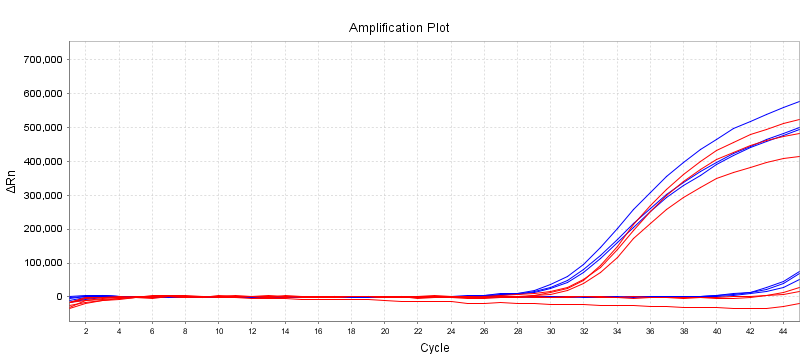
Abbildung 2 zeigt, dass Wildtypprimer sowohl Wildtyp- als auch Mutantenvorlagen amplifizieren. Laut Amplifikationskurve weist die Wildtypvorlage keinen Peak auf oder der Unterschied im Ct-Wert zwischen Wildtyp und Mutante beträgt 6-8, wodurch Wildtyp- und Mutantengene effektiv unterschieden werden können.
4.2 Die Produkterkennungsrate kann 0,05 % erreichen
Experiment 1: Selbstmischende Standardvorbereitungstechnik (50 %): 50 μl 10 ng/μl Wildtyp-DNA und 50 μl 3 x 103 Kopien des mutierten Plasmids.
Standort: KRASG13D (38G>A)
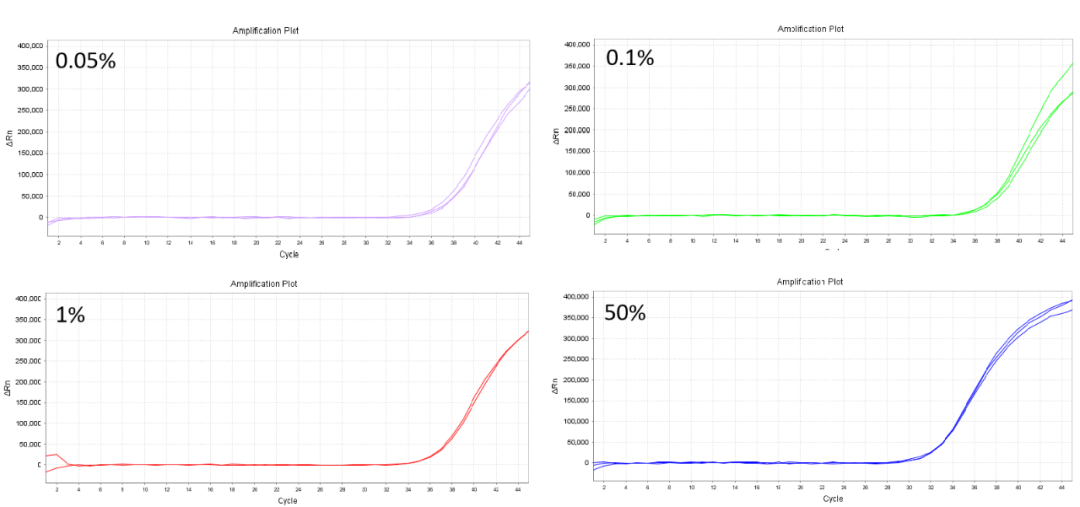
Abbildung 3.Laut der Verstärkungskurve ist das Produkt bei einer Konzentration von 0,05 % weiterhin wirksam
Experiment 2: Der gekaufte 5 %-Standard (50 ng/µl) wurde mit Bibliotheksverdünner auf 10 ng/µl verdünnt, dann weiter mit 10 ng/µl 293 g DNA auf 0,1 % und 0,05 % verdünnt.
Standort: L858R
Rot: 13755; Blau: Wettbewerber
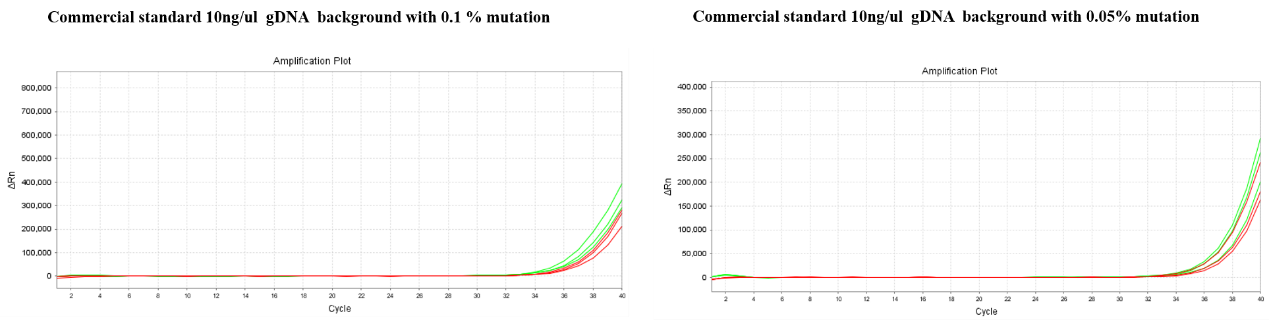
Abbildung 4: Laut Amplifikationskurve kann die Nachweisrate von Loci vor einem Wildtyp-gDNA-Hintergrund von 10 ng/μL bis zu 0,05 % betragen, und die anschließende Validierung nach kommerziellen Standards zeigt, dass unsere Produkte eine ausgezeichnete Nachweisrate aufweisen.
4.3 Ausgezeichnete Produktstabilität
Die verstärkende und blockierende Wirkung des Produkts wird durch 10 Einfrier- und Auftauzyklen nicht beeinträchtigt.
Experiment: Rot: -20 °C; Grün: 5-mal echtes Gefrieren und Auftauen; Blau: 8-mal echtes Gefrieren und Auftauen; Lila: 10-mal echtes Gefrieren und Auftauen.
Standort: L858R
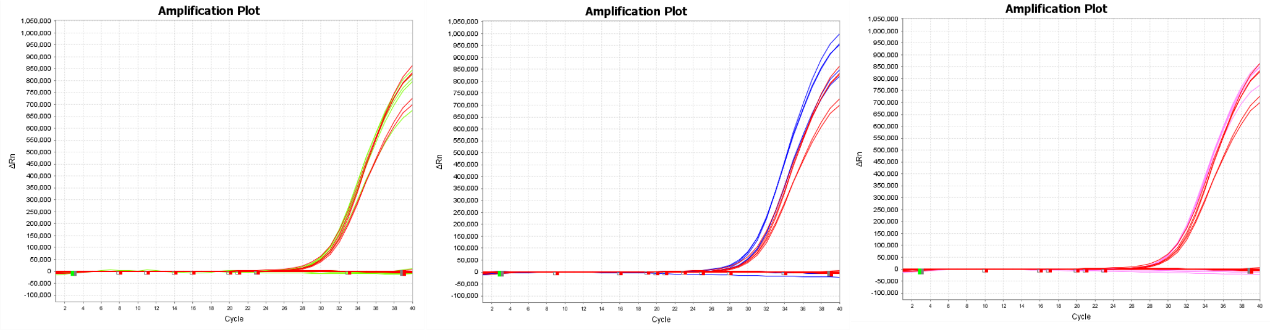
Abbildung 5 zeigt, dass zehnmaliges Einfrieren und Auftauen des Reagenz 13755 keinen Einfluss auf seine Amplifikations- und Blockierungseigenschaften hat.
Seine verstärkende und blockierende Wirkung wird durch 37 °C für 7 Tage nicht beeinträchtigt.
Experiment: Rot: -20 °C; Grün und Lila: 37 °C für 7 Tage.
Standort: L858R
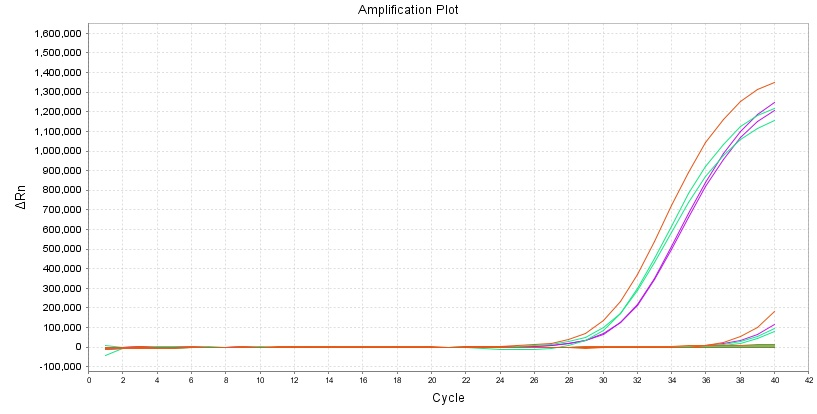
Abbildung 6 zeigt, dass sieben Tage bei 37 °C keinen Einfluss auf die Amplifikations- und Blockierungswirkung des 13755-Reagenz haben.
5. Wie bestellen Leute Produkte?
Tabelle 1: Von
| Produktname | Katze# | Größe |
| 2×Hieff UniconTM Multiplex WAFFEN qPCR Mischen (Erkundigen) | 13755ES60 | 100T |
| 13755ES80 | 1000T | |
| 13755ES92 | 10000T |
