In den letzten Jahren ist die Zahl der Infektionskrankheiten weltweit gestiegen, und die Krankheitserreger werden immer vielfältiger und komplexer. Eine große Herausforderung dabei ist, dass etwa die Hälfte der Patienten mit unbekannten Krankheitserregern infiziert ist, was die schnelle Diagnose dieser Erreger zu einer gewaltigen Aufgabe macht. Derzeit beruht die klinische Krankheitserregererkennung hauptsächlich auf traditionellen Kulturmethoden, PCR-Techniken, metagenomischer Sequenzierung (mNGS) und pathogenspezifischer Sequenzierung (tNGS).
Die quantitative Echtzeit-Fluoreszenz-PCR hat bei der Nukleinsäureerkennung von SARS-CoV-2 an Bedeutung gewonnen. Ebenso hat mNGS aufgrund seines Beitrags zur Bekämpfung von SARS-CoV-2 Aufmerksamkeit erregt und ist aus dem klinischen Umfeld in den Alltagsgebrauch übergegangen. tNGS, das die Vorteile sowohl der PCR als auch der NGS bietet, wie etwa Geschwindigkeit, Genauigkeit und Kosteneffizienz bei Sequenzierungsdiensten, gewinnt im Bereich klinischer Tests zunehmend an Bedeutung.
Gleichzeitig hat sich die Anwendung biologischer Produkte auf Biomedizin, biologische Landwirtschaft, Bioenergie, biologische Produktion, Umweltschutz und andere Bereiche ausgeweitet. Da der Marktanteil biologischer Produkte weiter wächst, haben Regierungsstellen und zuständige Behörden standardisierte Qualitätsmanagementsysteme und -standards für diese Produkte eingeführt. Ein besonderer Schwerpunkt liegt auf dem Vorhandensein von Nukleinsäurerückständen aus Wirtszellen in biologischen Produkten.
Biologische Produkte wie rekombinante Proteinmedikamente, Antikörpermedikamente, Impfstoffe sowie Zell- und Gentherapien werden unter Verwendung aufeinanderfolgender Stämme oder Zelllinien hergestellt, was möglicherweise dazu führt, dass Wirtsnukleinsäuren in den Endprodukten zurückbleiben. Restliche Wirtsnukleinsäuren können nachteilige Folgen haben, wie etwa unkontrollierte Zellvermehrung, die zur Tumorbildung führt, oder Verschlimmerung von Immunreaktionen durch Einschleusung viraler Gene. Daher sind eine effektive Entfernung von Nukleinsäurerückständen und eine strenge Rückstandserkennung von entscheidender Bedeutung, um die Sicherheit biologischer Produkte zu gewährleisten und die Forschungsanforderungen zu erfüllen. In der Industrie werden qPCR/RT-qPCR-Methoden häufig eingesetzt, um Nachweiskits für den Nachweis von Wirtsnukleinsäurerückständen in biologischen Produkten zu entwickeln.
Darüber hinaus werden kommerzielle molekulare Enzyme typischerweise unter Verwendung rekombinanter technischer Stämme wie E. coli exprimiert, was zur Anwesenheit genomischer Wirts-DNA in diesen molekularen Enzymen führt. Darüber hinaus können Umwelt- und menschliche Faktoren kontaminierte DNA in molekulare Enzymprodukte einbringen.
Während des Pathogennachweisprozesses können bakterielle Hintergrundnukleinsäuren aus Verunreinigungen Zielnukleinsäuren mit geringer Häufigkeit überdecken oder zusammen mit Zielnukleinsäuren nachgewiesen werden. Dies kann die Empfindlichkeit der Zielerkennung beeinträchtigen oder zu falsch positiven Ergebnissen führen, was die Diagnose und Behandlungsentscheidungen des medizinischen Fachpersonals erschwert.
Bei der Entwicklung und Herstellung von Qualitätskontrollprodukten für Tests auf Wirtsnukleinsäurerückstände kann das Vorhandensein von Resten von Wirtsnukleinsäuren, einschließlich solcher von Menschen, Mäusen, E. coli, Hefe und anderen, in molekularen Enzymen zu Ungenauigkeiten bei der Quantifizierung von Qualitätskontrollprodukten für Wirtsnukleinsäurerückstände führen. Dies birgt potenzielle Sicherheitsrisiken bei der Herstellung biologischer Produkte.
YEASEN UCF.METM Molekulare Enzymlösung mit extrem geringen Rückständen
Um das Problem der Interferenz von Hintergrundbakterien und Nukleinsäurerückständen des Wirtes zu lösen, YEASEN hat eine komplette F&E- und Produktionsplattform für UCF.ME aufgebautTM Molekulare Enzyme mit ultraniedrigen Rückständen. Und es ist gelungen, eine Vielzahl von UCF im großen Maßstab herzustellen.MICHTM Molekulare Enzyme mit ultraniedrigen Rückständen durch Materialauswahl, Umweltkontrolle, Prozessoptimierung und Qualitätssicherung.
YEASEN UCF.METM Molekulare Enzymprodukte mit extrem geringen Rückständen
YEASEN reformierte einen kompletten Satz molekularer Enzyme wie qPCR/RT-qPCR, NGS-Bibliotheksaufbau. Gleichzeitig hat UCF.ME™ Zur Verarbeitung molekularer Enzymprodukte wird ein Verfahren mit ultraniedrigen Rückständen verwendet. Wir können einen vollständigen Satz hochleistungsfähiger und mit ultraniedrigen Wirtsrückständen versehener molekularer Enzymrohstoffe für qPCR/RT-qPCR und NGS bereitstellen. Zu Verbessern Sie die Erkennungsgenauigkeit.
Tisch 1 Die Liste von YEASEN UCF.METM Molekulare Enzymprodukte mit extrem geringen Rückständen
| Produktklassifizierung | Produktname | Katalog-Nr. | Kriterien für Escherichia coli gDNA-Qualitätskontrolle |
| UCF.METM Enzymprodukte mit extrem geringen Rückständen für qPCR/RT-qPCR | Hieff UCF.METM Hotstart Sensitive Taq DNA-Polymerase (5 (Einheiten/µl) | 14314ES | <0,005 Kopien/Einheit |
| Hifair UCF.METM V Reverse Transkriptase (200 U/μL) | 14608ES | <0,005 Kopien/Einheit | |
| UCF.METM Muriner RNase-Inhibitor (40 U/μL) | 14672ES | <0,001 Kopien/Einheit | |
| UCF.METM RNase-Inhibitor mit hoher Affinität (40 U/μL) | 14675ES | <0,001 Kopien/Einheit | |
| UCF.METM Uracil-DNA-Glykosylase (UDG/UNG), hitzelabil, 1 U/μL | 14466ES | <0,1 Kopien/Einheit | |
| UCF.METM Uracil-DNA-Glykosylase (UDG/UNG), 1 U/μL | 14454ES | <0,1 Kopien/Einheit |
Teildatenpräsentation (UCF.ME)TM Hotstart Sensitive Taq DNA-Polymerase als Beispiel)
- RRückstandvon coli gDNA <0.005 Kopien/Einheit
- coligDNA-Reste verschiedener Chargen von UCF.METMTaq-Enzym (Cat#14314ES) nachgewiesen. Das Ergebnis zeigte, dass Escherichia coli gDNA-Reste Reste der Taq-DNA-Polymerase lagen deutlich unter 0,005 Kopien/Einheit.
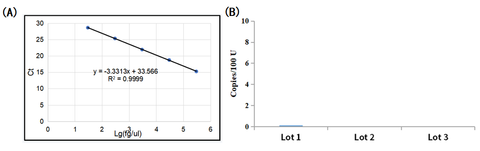
Figur 1: Erkennung von E.coli GDNA-Rest von UCF.METM Taq-Enzym (Kat.-Nr. 14314ES)
- Es wurde keine restliche Plasmid-DNA nachgewiesen
Plasmid-DNA-Reste von UCF.METM Taq-Enzym (Cat#14314ES) und Taq-DNA-Polymerase der Marke A wurden nachgewiesen. Die Ergebnisse zeigten, dass in der Taq-DNA-Polymerase der Marke A Plasmid-DNA-Rückstände vorhanden waren. In UCF.ME wurde keine Plasmid-DNA nachgewiesen.TM Taq-Enzym (Kat.-Nr. 14314ES).
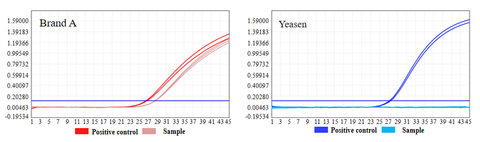
Abbildung 2: Ergebnisse der Plasmid-DNA-Rückstandserkennung
- Elf Arten häufiger Hintergrundbakterien wurden nicht nachgewiesen
Verwenden Sie UCF.METM Taq-Enzym (Cat#14314ES) gekoppelt mit Primern und Sonden von Pseudomonas aeruginosa, Acinetobacter baumannii, Serratia marcescens, Klebsiella pneumoniae, Stenotrophomonas maltophil, Streptococcus pneumoniae, Enterococcus faecium, Staphylococcus aureus, Escherichia coli, Enterococcus faecalis zur Herstellung einer qPCR-Vormischung. NTC (No Template Control) wurde nachgewiesen. Die Ergebnisse zeigten, dass in UCF.ME keine Rückstände der oben genannten 11 häufigen Hintergrundbakterien nachgewiesen wurden.TM Taq-Enzym (Kat.-Nr. 14314ES).
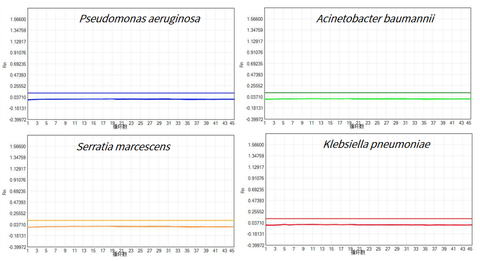
Abbildung 3: Testergebnisse von 11 häufigen Hintergrundbakterien (begrenzt durch Platz, Pseudomonas aeruginosa, Acinetobacter baumannii, Serratia marcescens Und Klebsiella pneumoniae sind als Beispiele aufgeführt)
- Testfallpräsentation des Kunden
Das kommerzielle Taq-Enzym und YEASEN UCF.METM Zur Erkennung von NTC wurden das Taq-Enzym (Cat#14314ES) und die Kunden- Escherichia coli spezifischer Primer, und die Ergebnisse zeigten, dass das kommerzielle Taq-Enzym in 22 wiederholten Experimenten 5-mal einen Peak erreichte. Die UCF.METM Das Taq-Enzym (Cat#14314ES) erreichte in 22 Replikaten keinen Peak, was darauf hinweist, dass das UCF.METM Taq-Enzym (Cat#14314ES) konnte nicht nachgewiesen werden Escherichia coli Die gDNA-Sequenz ist eine Sequenz, die von einem DNA-Strang abgeleitet ist.
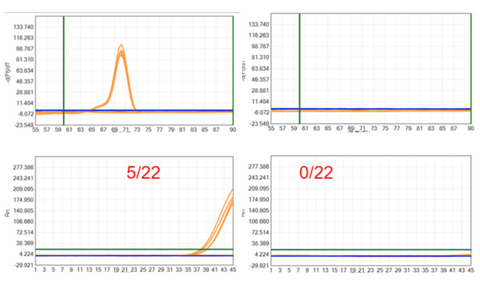
Figur 4: DErkennung Ergebnis von E.coli gDNA-Rückstände (Testfall des Kunden)
Links: Kommerziell erhältliches konventionelles Taq-Enzym; Rechts: UCF.METM Taq-Enzym (Kat.-Nr. 14314ES)
Verwandte Produktempfehlung
| Produktklassifizierung | Produktname | Katalog-Nr. |
| Kit zur Erkennung von Nukleinsäurerückständen des Wirts | 41332ES | |
| HEK293-Kit zur Erkennung von DNA-Rückständen in Wirtszellen (3G) | 41331ES | |
| 41307ES | ||
| E.coli Kit zur Erkennung von DNA-Rückständen in Wirtszellen (2G) | 41308ES | |
| Kit zur Erkennung von DNA-Rückständen in Wirtszellen von Hansenula polymorpha | 41317ES | |
| E.coli Kit zur Erkennung von RNA-Rückständen in Wirtszellen | 41318ES |
