Beschreibung
CHO Das Host Cell DNA Residue Detection Kit dient zur quantitativen Analyse von CHO Wirtszell-DNA-Rückstände in Zwischenproben, Halbfertig- und Fertigprodukten verschiedener biologischer Produkte.
Dieses Kit verwendet die Taqman-Fluoreszenzsonde und die Polymerase-Kettenreaktion (PCR), die eine Mindestnachweisgrenze auf fg-Niveau aufweist und spezifisch und schnell den Rest-CHO erkennen kann. Zell-DNA. Das Kit muss zusammen mit dem Residual DNA Sample Preparation Kit (Kat.-Nr. 18461ES) verwendet werden.
Produkt Komponenten
| NEIN. | Name | 41332ES50 (50T) | 41332ES60 (100T) |
| 41332-A | CHO qPCR Mischen | 0,75 ml | 1.5 ml |
| 41332-B | CHO Primer&Sonde Mischen | 250 μl | 500 μl |
| 41332-C | DNA-Verdünnung Puffer | 2×1,8 ml | 4×1,8 ml |
| 41332-D | CHO DNA-Kontrolle (30 ng/µL) | 25 μl | 50 μl |
Versand und Lagerung
1. Versand auf Trockeneis und bei -20°C gelagert für 2 Jahr
Dieses Produkt sollte 2 Jahre lang bei -25–15 °C gelagert werden.
Sowohl 41332-A als auch 41332-B sollten lichtgeschützt gelagert werden.
Anwendbare Instrumentenmodelle
Einschließlich (jedoch nicht beschränkt auf):
Bio-Rad: Optisches Modul CFX96.
Thermo Scientific: ABI 7500; ABI Quant Studio 5.
Anweisungen
- CHO DNA-Standardverdünnung und Standardkurvenvorbereitung
Der CHO Die DNA-Kontrolle wurde mit dem im Kit enthaltenen DNA-Verdünnungspuffer gradientverdünnt*und die Verdünnung
Konzentration beträgt 3 ng/µl, 300 pg/µl, 30 pg/µl, 3 pg/µl, 300 fg/µl, 30 fg/µl, 3 fg/µl.
Detaillierte Anweisungen finden Sie weiter unten:
- Tauen Sie die CHO-DNA-Kontrolle und den DNA-Verdünnungspuffer auf Eis auf. Mischen Sie alles nach dem vollständigen Auftauen vorsichtig mit einem Vortexer und zentrifugieren Sie es bei niedriger Geschwindigkeit 10 Sekunden lang.
- Nehmen Sie sieben saubere 1,5-ml-Röhrchen heraus, die mit Std0, Std1, Std2, Std3, Std4, Std5, Std6 gekennzeichnet sind.
- Fügen Sie 90 μL DNA-Verdünnungspuffer und 10 μL CHODNA-Kontrolle zu dem 1,5-ml-Mikrozentrifugenröhrchen mit der Bezeichnung Std0 hinzu, nämlich auf 3 ng/μL verdünnen. Mischen und dann 10 Sekunden zentrifugieren. Den verdünnten DNA-Standard in Unterpackungen verpacken und kurzfristig (nicht länger als 3 Monate) bei -25 bis -15 °C lagern.**.Bitte vermeiden Sie wiederholtes Einfrieren und Auftauen.
- 90 μL DNA-Verdünnungspuffer in andere Röhrchen geben***, dann folgen Sie dem untenstehenden Verfahren für die seriellen Verdünnungen****.
| Rohr | Verdünnungsverhältnis | Standardkonzentration |
| Std1 | 10 μL Std0 + 90 μL DNA-Verdünnungspuffer | 300 pg/µL |
| Std2 | 10 μL Std1 + 90 μL DNA-Verdünnungspuffer | 30 pg/µL |
| Std3 | 10 μL Std2 + 90 μL DNA-Verdünnungspuffer | 3 pg/µL |
| Std4 | 10 μL Std3 + 90 μL DNA-Verdünnungspuffer | 300 fg/µL |
| Std5 | 10 μL Std4 + 90 μL DNA-Verdünnungspuffer | 30 fg/µL |
| Std6 | 10 μL Std5 + 90 μL DNA-Verdünnungspuffer | 3 fg/µL |
Tabelle 1 Standardgradientenverdünnung
*Für jede Konzentration werden drei Replikationsvertiefungen benötigt. Der Nachweisbereich liegt bei 3 fg/µl bis 300 pg/µl und kann erweitert werden.
**Um die Anzahl wiederholter Einfrier- und Auftauvorgänge zu verringern und eine Kontamination zu vermeiden, wird empfohlen, die DNA-Kontrolle beim ersten Mal in Aliquots bei -25 bis -15 °C aufzubewahren.
***Nach dem Auftauen kann der DNA-Verdünnungspuffer 7 Tage lang bei 2–8 °C aufbewahrt werden. Wenn er längere Zeit nicht verwendet wird, lagern Sie ihn bitte bei -25–-15 °C.
****Stellen Sie sicher, dass die Vorlage vollständig gemischt ist, und schütteln Sie die Mischung für jede Gradientenverdünnung 15 Sekunden bis 1 Minute lang vorsichtig.
- Vorbereitung der Extraktionswiederherstellungskontrolle (ERC)
Stellen Sie die Konzentration der CHO-DNA im ERC nach Bedarf ein (die ERC-Probe wurde beispielsweise mit 30 pg CHO-DNA hergestellt), und zwar wie folgt:
- Geben Sie 100 μl Testprobe in ein sauberes 1,5-ml-Röhrchen, fügen Sie dann 10 μl 3pg/μl CHO-DNA-Standard (Std3) hinzu und mischen Sie gut, gekennzeichnet als ERC.
- Führen Sie die DNA-Extraktion der ERC-Probe zusammen mit den Testproben durch, um die gereinigte ERC-Probe vorzubereiten.
- Vorbereitung der Negativkontrolllösung (NCS)
Stellen Sie die Negativkontrolle im Experiment ein. Die spezifischen Arbeitsschritte sind wie folgt:
1) Geben Sie 100 μL Probenmatrix (oder DNA-Verdünnungspuffer) in ein sauberes 1,5-ml-Röhrchen und markieren Sie es als NCS.
2) Führen Sie die DNA-Extraktion der NCS-Probe zusammen mit den Testproben durch, um die gereinigte NCS-Probe vorzubereiten.
- No Template Control (NTC)-Vorbereitung
Stellen Sie im Experiment die Steuerung ohne Vorlage ein. Die spezifischen Betriebsschritte sind wie folgt:
1) NTC erfordert keine Probenvorbehandlung und kann im Stadium der qPCR-Erkennung von Rest-DNA konfiguriert werden.
2) Die NTC-Probe in jedem Röhrchen oder jeder Vertiefung besteht aus 20 μl Mischung (d. h. 15 μl CHO qPCR-Mischung + 4 μl CHO Primer&probe-Mischung + 1 μl IC) + 10 μl DNA-Verdünnungspuffer. Es wird empfohlen, drei Replikationsvertiefungen zu konfigurieren.
- PCR-Reaktionssystem
| Komponente | Volumen (μl) |
| CHO qPCR-Mischung* | 15 |
| CHO Primer&Sonden-Mix | 5 |
| DNA-Vorlage | 10 |
| Gesamtvolumen** | 30 |
Tabelle 2 Reaktionssystem
*Berechnen Sie das gesamte PCR-Reaktionsvolumen anhand der Anzahl der Reaktionen: qPCR-Mix = (Anzahl der Reaktionen + 2) × (15 + 4 + 1) μL (einschließlich der Verluste von zwei Reaktionskammern). Im Experiment werden mehr als drei Replikate für jede Probe empfohlen.
**Nachdem Sie das Röhrchen verschlossen oder die Platte versiegelt haben, zentrifugieren Sie das Reaktionsröhrchen oder die Platte 10 Sekunden lang bei niedriger Geschwindigkeit. Nach ausreichendem Schütteln und Mischen für 5 Sekunden wiederholen Sie die Zentrifugation, um die Flüssigkeit vom Deckel oder der Wand auf den Boden zu sammeln. Vermeiden Sie Blasen während des Betriebs.
Die empfohlene Plattenkonfiguration finden Sie in der Tabelle unten:
|
| 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | 12 |
| A | NTC |
| TS 1 | TS 1 | TS 1 |
| Std. 1 | Std. 1 | Std. 1 |
|
|
|
| B | NTC |
| TS 2 | TS 2 | TS 2 |
| Std. 2 | Std. 2 | Std. 2 |
|
|
|
| C | NTC |
| TS 3 | TS 3 | TS 3 |
| Std. 3 | Std. 3 | Std. 3 |
|
|
|
| D |
|
|
|
|
|
| Std 4 | Std 4 | Std 4 |
|
|
|
| E | NCS |
| ERC 1 | ERC 1 | ERC 1 |
| Std. 5 | Std. 5 | Std. 5 |
|
|
|
| F | NCS |
| ERC 2 | ERC 2 | ERC 2 |
| Std. 6 | Std. 6 | Std. 6 |
|
|
|
| G | NCS |
| ERC 3 | ERC 3 | ERC 3 |
|
|
|
|
|
|
|
| H |
|
|
|
|
|
|
|
|
|
|
|
|
Tabelle 3 Computer-on-Referenzplatine
Das Plattenlayout umfasst: 6 Std (die Standardkurve von 6 Standardkonzentrationen), 1 NTC (No Template Control), 1 NCS (Negative Control Solution), 3 TS (Testproben), 3 ERC (Extraktionsrückgewinnungskontrolle).Drei Replikationsvertiefungen für jede Probe.
- Aufstellen Richtlinien für ein PCR-Instrument(2-Schritt-Methode) (zB Thermo ABI 7500 qPCR-Instrument, Softwareversion 2.0)
Die folgenden Anweisungen gelten nur für das Thermo ABI 7500 qPCR-Gerät (Softwareversion 2.0). Wenn Sie ein anderes Gerät verwenden, finden Sie die Einrichtungsrichtlinien im entsprechenden Gerätehandbuch.
1) Erstellen Sie ein neues Experiment und wählen Sie die Vorlage für die absolute Quantifizierung oder benutzerdefiniert.
2) Erstellen Sie eine Detektionssonde mit dem Namen „CHO-DNA“, wählen Sie als Reporter-Fluorophor „FAM“ und als Quench-Fluorophor „Keine“ aus. Erstellen Sie eine weitere Detektionssonde, nennen Sie sie „IC“, und wählen Sie als Reporter-Fluorophor „CY5“ und als Quenching-Fluorophor „Keine“. Die Referenzfluoreszenz ist „ROX“ (die Referenzfluoreszenz kann auf dem Instrumentenmodell usw. basieren. Wählen Sie aus, ob Sie sie hinzufügen müssen).
3) Geben Sie im Bereich „Proben“ nacheinander alle Probeninformationen ein. Wählen Sie dann die Vertiefungen aus, wählen Sie das Ziel und die Proben entsprechend aus. Stellen Sie die Aufgabe von CHO ein DNA-Standard als Standard und weisen Sie die Werte 300000, 30000, 3000, 300, 30, 3 zu (die Einheit der DNA-Konzentration in jedem Well ist fg/μL) in der Spalte „Quantity“ und benennen Sie die Wells mit Std 1, Std 2, Std 3, Std 4, Std 5, Std. 6, entsprechend. Stellen Sie die Aufgabe von NTC als NTC ein. Stellen Sie NCS, TS und ERC ein als Unbekannt und benennen Sie sie entsprechend dem obigen Plattenlayout. Klicken Sie dann auf Weiter.
4) Stellen Sie das Amplifikationsprogramm ein: Stellen Sie das Reaktionsvolumen auf 30 μl ein.
| Zyklusschritt | Temperatur (℃) | Zeit | Zyklen |
| Erste Denaturierung | 95℃ | 5 Minuten | 1 |
| Denaturierung | 95℃ | 15 Sek. | 40 |
| Annealing/Verlängerung (Fluoreszenzsammlung) | 60℃ | 30 Sek. |
Tabelle 4 Amplifikationsverfahren
- Analyse der qPCR-Ergebnisse
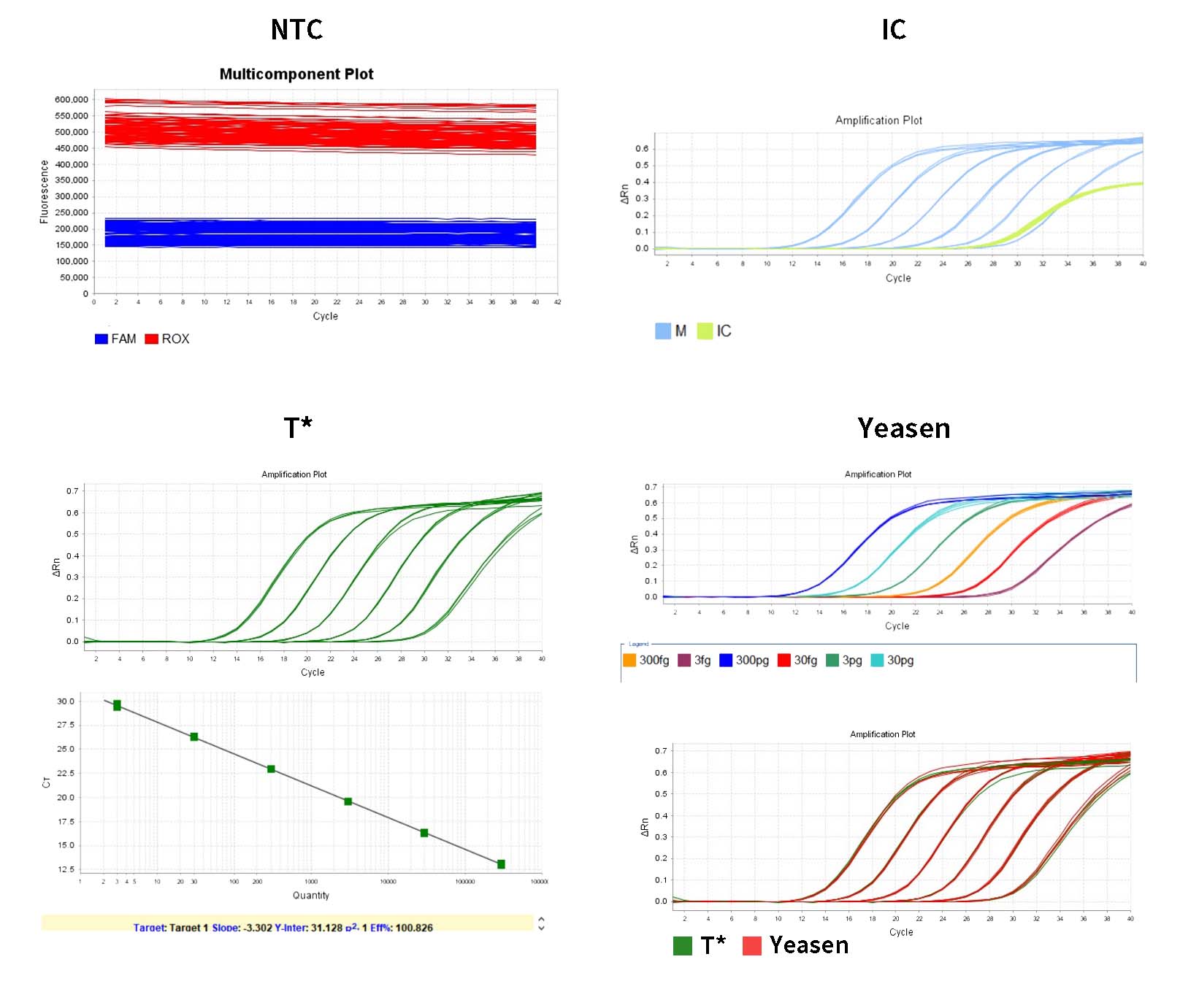
1) Das System gibt den Schwellenwert automatisch im Amplifikationsdiagramm-Panel der Analyse an. Der vom System angegebene Schwellenwert liegt manchmal zu nahe an der Basislinie, was zu einem großen Unterschied im Ct zwischen den Replikationsvertiefungen führt. Sie können den Schwellenwert manuell auf eine geeignete Position einstellen und auf „Analysieren“ klicken. Anschließend können Sie zunächst im Mehrkomponentendiagramm überprüfen, ob die Amplifikationskurve normal ist.
2) Überprüfen Sie auf der Registerkarte „Ergebnisanalyse“ das Standardkurvendiagramm. Überprüfen Sie die Werte für R2, Effizienz, Steigung und Y-Achsenabschnitt. Für eine normale Standardkurve, R²>0,99, 90 % ≤ Eff % ≤ 110 %, -3,6 ≤ Steigung ≤ -3,1.
3) Im Bereich „Vertiefungstabelle anzeigen“ in der Analyse werden die Konzentrationen der einzelnen Proben als Menge angezeigt. Die Einheit ist fg/µL. Die Einheiten können im Testbericht umgerechnet werden.
4) Die Parametereinstellungen der Ergebnisanalyse müssen auf dem jeweiligen Modell und der verwendeten Softwareversion basieren und können im Allgemeinen vom Instrument automatisch interpretiert werden.
5) Berechnen Sie die Spike-Wiederherstellungsrate basierend auf den Testergebnissen der zu messenden Probe TS und der Proben-Spike-Wiederherstellungsrate ERC. Die Wiederherstellungsrate der Spikes muss zwischen 50 % und 150 % liegen. Formel für das Messgerät für die Spike-Wiederherstellungsrate: Wiederherstellung (%) = {Sample spiked assay (eg.pg/μL) - Sample assay (eg.pg/μL)} x Elutionsvolumen (μL) / Theoretischer Wert der DNA-Zugabemenge (z. B. pg) x 100 %.
6) Der Ct-Wert des negativen Kontroll-NCS sollte größer sein als der Mittelwert der niedrigsten Ct-Konzentration des Standards.
- Der NTC der vorlagenfreien Kontrolle sollte unbestimmt sein oder der Ct-Wert sollte ≥3 sein.
Hinweise
- Dieses Produkt ist nur für Forschungszwecke bestimmt.
- Bitte tragen Sie zu Ihrer Sicherheit Laborkittel und Einweghandschuhe.
3. Bitte lesen Sie dieses Handbuch sorgfältig durch, bevor Sie dieses Reagenz verwenden. Das Experiment sollte standardisiert werden, einschließlich Probenhandhabung, Vorbereitung des Reaktionssystems und Probenzugabe.
4. Stellen Sie sicher, dass jede Komponente vor der Verwendung vollständig verwirbelt und bei niedriger Geschwindigkeit zentrifugiert wurde.
Zahlung und Sicherheit
Ihre Zahlungsinformationen werden sicher verarbeitet. Wir speichern weder Kreditkartendaten noch Zugriff auf Ihre Kreditkarteninformationen.
Anfrage
Sie können auch mögen
FAQ
Das Produkt ist nur für Forschungszwecke bestimmt und nicht für die therapeutische oder diagnostische Anwendung bei Menschen oder Tieren. Produkte und Inhalte sind durch Patente, Marken und Urheberrechte von
Für bestimmte Anwendungen sind möglicherweise zusätzliche geistige Eigentumsrechte Dritter erforderlich.