Haben Sie gelernt, wie man eine PCR-freie Bibliothek erstellt?
Bei der Hochdurchsatzsequenzierung erfordert die herkömmliche Bibliothekserstellung eine PCR-Amplifikation. Einerseits dient sie dazu, DNA-Spurenproben zu amplifizieren und die Bibliotheksausbeute zu erhöhen. Andererseits kann sie Fluoreszenzsignale amplifizieren, was es Sequenzierern erleichtert, Fluoreszenzsignale zu erfassen und zu identifizieren, um die Sequenzierungsgenauigkeit zu verbessern. PCR ist jedoch wie ein „zweischneidiges Schwert“. Während sie das Problem des geringen anfänglichen Probenvolumens löst und das Fluoreszenzsignal amplifiziert, führt sie auch Amplifikationsfehler und -verzerrungen ein und kann das „wahre Gesicht“ der Genomsequenz nicht perfekt darstellen. Darüber hinaus weist PCR-Polymerase eine gewisse Amplifikationsverzerrung auf. Einige Regionen, insbesondere Regionen mit hohem oder niedrigem GC, Sekundärstruktur und andere, weisen eine geringe Amplifikationseffizienz auf und sind schwer abzudecken. Sie führt auch eine große Anzahl falscher InDels ein und führt zu einer großen Anzahl von Duplikaten. Dies führt zu einer Verschwendung von DNA-Datenvolumen und erhöht die Kosten der Sequenzierung. Daher bietet die Einführung der PCR-freien Technologie nicht nur die Vorteile der PCR, sondern überwindet auch die Nachteile der PCR. Es kann nicht nur hochwertige Bibliotheken mit hoher Empfindlichkeit bereitstellen und die Erkennung von Spurenproben ermöglichen, sondern weist auch eine hohe Genauigkeit auf und reduziert den Validierungsaufwand für nachfolgende Variantenstudien. Was genau ist also die PCR-freie Bibliothek und was sind ihre Vorteile?
1. Was ist eine PCR-freie Bibliothek?
2. Was sind die Vorteile der PCR-freien Technologie?
3. Datenanzeige von PCR-Free
4. Häufig gestellte Fragen
5. Produktinformationen
6. Zum Thema Lesen
1. Was ist eine PCR-freie Bibliothek?
Die Sequenzierungstechnologie der nächsten Generation ist die am häufigsten verwendete Technologie in der Hochdurchsatz-Sequenzierungsforschung in der modernen Molekularbiologie. Die traditionelle Sequenzierung der ersten Generation kann die Anforderungen der Forscher nicht mehr vollständig erfüllen. Die Neusequenzierung des Genoms von Modellorganismen und die Genomsequenzierung von Nicht-Modellorganismen sind kostenintensiver. Die Sequenzierungstechnologie mit niedrigerem Durchsatz und schnellerer Geschwindigkeit führte zur Sequenzierungstechnologie der zweiten Generation. Das Kernprinzip der Sequenzierungstechnologie der zweiten Generation ist die Sequenzierung durch Synthese, d. h. die Bestimmung der DNA-Sequenz durch Erfassung der neu synthetisierten, endmarkierten Baseninformationen. Das Hauptprinzip der Sequenzierungstechnologie der zweiten Generation für die DNA-Sequenzierung besteht darin, zuerst die DNA zu fragmentieren, das Ende der fragmentierten DNA zu reparieren und dann auf beiden Seiten spezifische Adapter hinzuzufügen und dann verschiedene Methoden zu verwenden, um Millionen räumlich fixierter PCRs zu erzeugen. Bei klonalen Arrays werden Sequenzierungsdaten durch Primerhybridisierung und enzymatische Verlängerungsreaktionen gewonnen, um die durch jede Verlängerungsreaktion eingebauten Fluoreszenzmarkierungen abzubilden.
Die DNA-Sequenzierung durch Next-Generation-Sequencing umfasst im Wesentlichen zwei Prozesse: Bibliotheksvorbereitung und Sequenzierung auf der Maschine. Während der Bibliotheksvorbereitung werden zufällig unterbrochene genomische Fragmente typischerweise durch Standard-PCR amplifiziert. Bei einigen speziellen Vorlagen gibt es jedoch Faktoren wie eine komplexe Sekundärstruktur oder eine schlechte thermische Stabilität, die die Amplifikationspräferenz der Vorlagen-PCR beeinflussen, sodass nicht alle genomischen Sequenzen in der PCR-Amplifikationsbibliothek gleichermaßen wiedergegeben werden können. Insbesondere bei einigen Vorlagen mit hohem GC- oder hohem AT-Gehalt ist es manchmal schwierig, die PCR-Methode zur Amplifikation für die Bibliothekskonstruktion zu verwenden. Es gibt keinen Unterschied zwischen der PCR-freien Bibliothek und der herkömmlichen PCR-Bibliothek bei der Sequenzierung auf der Maschine, außer dass während des Bibliothekskonstruktionsprozesses keine PCR durchgeführt wird. Die PCR-freie Bibliothek kann theoretisch die Verteilung der Datenablesungen verbessern und eine gleichmäßigere Genomabdeckung erzeugen.Die PCR-freie Bibliothek ist eine Ergänzung zur Bibliothekskonstruktionsmethode. Da sie direkt in eine Onboard-Bibliothek ohne PCR-Amplifikation vorbereitet wird, kann sie die Abdeckung einiger Regionen mit hohem GC oder hohem AT verbessern und so die durch PCR, Datenverzerrung und Sequenzwiederholung eingeführten Fehlerbasen reduzieren.
Die Kernschritte beim Aufbau einer herkömmlichen NGS-Bibliothek sind Genomfragmentierung, Endreparatur, Adapteraddition, PCR und Signalverstärkung vor der Sequenzierung. Was ist also mit dem PCR-freien Bibliotheksaufbau? Wie der Name schon sagt, handelt es sich dabei um einen Bibliotheksaufbauprozess, der keine PCR erfordert. Die Kernschritte beim Bibliotheksaufbau sind Genomfragmentierung, Endmodifikation, Linkeraddition und Bibliotheksqualitätskontrolle. Tatsächlich kann dies jedoch nur im Bibliotheksaufbauprozess als PCR-frei angesehen werden. Wirklich PCR-frei sollte ein Prozess vom Bibliotheksaufbau bis zur Sequenzierung ohne Bibliotheksverstärkung (wie Einzelmolekülsequenzierungstechnologie) oder eine Sequenzierungstechnologie ohne Ansammlung von PCR-Verstärkungsfehlern (wie DNBseq von MGI) sein.

Abbildung 1. Flussdiagramm der konventionellen Bibliothekskonstruktion und der PCR-freien Bibliothekskonstruktion
2. Was sind die Vorteile der PCR-freien Technologie?
Beim routinemäßigen Bibliotheksaufbau kann das Amplifikationsenzym Fehler einbringen. Durch zyklische Amplifikation werden diese Fehler akkumuliert und verstärkt, was die Genauigkeit der DNA-Sequenzreplikation verringert. Darüber hinaus weist die DNA-Polymerase auch eine gewisse Amplifikationsneigung auf, insbesondere bei Regionen mit großen Unterschieden im GC-Gehalt oder in Sekundärstrukturen, was zu einer geringen Amplifikationseffizienz und einer schlechten Abdeckungsgleichmäßigkeit führt; während es falsche InDels einführt, führt es auch zu einer großen Anzahl von Duplikaten, was zu einer Verschwendung des DNA-Datenvolumens führt und die Sequenzierungskosten erhöht. Die PCR-freie Technologie kann die oben genannten Probleme, die durch die PCR-Amplifikation beim herkömmlichen Bibliotheksaufbau verursacht werden, gut vermeiden. Beim Prozess des Bibliotheksaufbaus und der Sequenzierung hat ein PCR-Amplifikationsprozess einen größeren Einfluss auf die Einheitlichkeit der Genomabdeckung. Bei DNA-Vorlagen haben einige komplexe Sekundärstrukturen und einige große Unterschiede in der thermischen Stabilität. Diese Faktoren wirken sich auf die Effizienz der PCR-Amplifikation aus. Daher kann beim PCR-Amplifikationsprozess nicht garantiert werden, dass alle genomischen Fragmente die gleiche Amplifikationseffizienz erreichen, sondern es gibt eine offensichtliche Amplifikationsverzerrung, wie z. B. eine geringe Abdeckung in Bereichen mit hohem GC oder niedrigem GC des Genoms. Im gesamten Prozess der PCR-freien Sequenzierung findet jedoch keine PCR statt. Im Vergleich zur PCR-Bibliothekskonstruktion wird die Abdeckung von Bereichen mit hohem GC und AT/TA-Wiederholungsbereichen des Genoms verbessert. Die spezifischen Vorteile sind wie folgt.
2.1 Optimierter Bibliotheksvorbereitungsprozess, spart Zeit
Durch die PCR-freie Bibliotheksvorbereitung entfällt die Notwendigkeit einer PCR-Amplifikation und anderer Schritte, was den Prozess der Bibliotheksvorbereitung rationalisiert und Zeit spart.
2.2 Reduzierte Kosten für die Bibliotheksvorbereitung
Im herkömmlichen Bibliotheksvorbereitungsprozess werden teure Enzyme mit hoher Genauigkeit für die PCR-Amplifikation verwendet, um die Sequenzauthentizität sicherzustellen. Gleichzeitig macht PCR-Free den PCR-Amplifikationsschritt überflüssig und reduziert so die Kosten für die Bibliotheksvorbereitung.
2.3 Reduzieren Sie den Verstärkungsbias
DNA-Amplifikationsenzyme weisen bestimmte Amplifikationsverzerrungen auf, insbesondere bei Vorlagen mit erheblichen Unterschieden im GC-Gehalt oder in der Sekundärstruktur. Daher kann PCR-Free die durch die Polymerase verursachte Amplifikationsverzerrung wirksam vermeiden.
2.4 Keine PCR-Duplikation
PCR-Free kann doppelte Lesevorgänge reduzieren und die Unique-Mapping-Rate sowie die effektive Datennutzung erhöhen.
2.5 Reduzierte Fehlerquote
Bei der PCR-Amplifikation kommt es häufiger zu Replikationsfehlern beim Einfügen und Löschen, was zu einer höheren Fehlerrate bei der Indel-Sequenzierung führt, während PCR-Free die Entstehung und Ansammlung solcher Fehler wirksam vermeiden kann.
3. Datenanzeige von PCR-Free
Hieff NGS™ Ultima Pro PCR-freies DNA-Bibliotheksvorbereitungskit V2 (Kat.-Nr. 12196ES) weist eine bessere Bibliothekskonvertierungsrate auf als Konkurrenzprodukte.
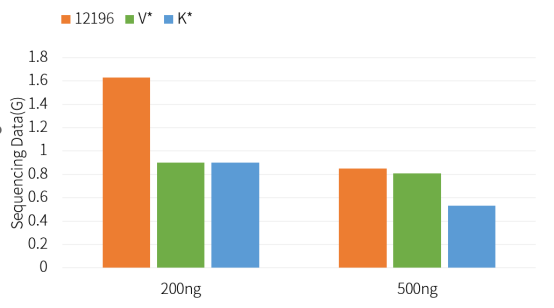
Abbildung 2. Menge der Off-Machine-Sequenzierungsdaten für den PCR-freien Bibliotheksaufbau
4. Häufig gestellte Fragen
F: Welche Arten von Proben eignen sich für PCR-freie Bibliotheken?
A: Für einige Proben mit komplexen Sekundärstrukturen, hohem GC und PCR-Präferenz kann zum Aufbau einer PCR-freien Bibliothek gewählt werden.
F: Wie groß ist die allgemeine Fragmentgröße der PCR-freien Bibliothek?
A: Die PCR-freie Bibliothek selbst hat keine besonderen Anforderungen an die Fragmentgröße. Die PCR-Produktbibliothek wird entsprechend der Größe des PCR-Produkts aufgebaut. Für die genomische Bibliothek wird eine Größe von etwa 350 bp empfohlen, damit die Qualität der Bibliotheksdaten relativ gut ist.
5. Produktinformationen
Das von
Tabelle 1. Produktinformationen
| Produkt Name | Kat.-Nr. | Größe |
| Hieff NGS™ Ultima Pro PCR-freies DNA-Bibliotheksvorbereitungskit V2 | 12196ES24/96 | 24/96T |
6. Zum Thema Lesen
Die Lösung für die NGS-Bibliotheksvorbereitung aus DNA-Proben
Wie gut wissen Sie über NGS-bezogene Technologie?
