Qubit, was für ein leistungsstarkes Tool zur Quantifizierung von NGS-Bibliotheken!
Der Kern der Quantifizierung von NGS-Bibliotheken ist die Quantifizierung von Nukleinsäuren (DNA oder RNA). Eine genaue Analyse der Quantifizierung von NGS-Bibliotheken ist entscheidend, um qualitativ hochwertige Sequenzierungsdaten zu erhalten. Wenn die quantitative Konzentration der NGS-Bibliothek hoch ist, kann die Ausbeute an Sequenzierungsdaten aufgrund unzureichender Clusterung gering sein. Eine niedrige quantitative Konzentration kann jedoch zu Signalinterferenzen zwischen Clustern führen, wodurch Daten von geringer Qualität entstehen und sogar zu Sequenzierungsfehlern führen. Darüber hinaus werden mit der Zunahme des Sequenzierungsflusses von Sequenzern häufig mehrere NGS-Bibliotheken zur Sequenzierung kombiniert. Entsprechend den Anforderungen an die Größe der Sequenzierungsdaten werden NGS-Bibliotheken mit entsprechenden Molarmengen basierend auf den quantitativen Ergebnissen gemischt, um die Ausgabe der Größe der einzelnen Probendaten zu stabilisieren. Daher ist eine genaue Quantifizierung von NGS-Bibliotheken von entscheidender Bedeutung.
Derzeit gibt es zwei Hauptmethoden zur Quantifizierung von NGS-Bibliotheken: die Ultraviolettabsorptionsmethode und die Fluoreszenzfarbstoffmethode. NanoDrop ist ein DNA-Quantifizierungsinstrument, das auf dem Prinzip der Ultraviolettabsorption basiert; Qubit ist ein DNA-Quantifizierungsinstrument, das nach dem Prinzip der Fluoreszenzfarbstoff entwickelt wurde. Die quantitative Genauigkeit und die Kosten dieser beiden Methoden sind unterschiedlich. Hier wird hauptsächlich die Qubit-Methode empfohlen. Warum empfehlen wir Ihnen also das Qubit? Welche Eigenschaften haben die von
1. Warum empfehlen wir Ihnen Qubit?
2. Welche Eigenschaften haben die von
- Wie funktioniert Qubit?
- Beliebtes Produkt von
Yeasen - Produkteigenschaften
3. Wie wählt man Qubit für verschiedene Proben aus?
1. Warum empfehlen wir Ihnen Qubit?
Es besteht kein Zweifel, dass Qubit besser ist als Nanodrop. Warum ist Qubit also besser als Nanodrop? Was ist der Unterschied zwischen Qubit und Nanodrop? Erstens unterscheiden sie sich, wie bereits erwähnt, im Prinzip der DNA-Quantifizierung. NanoDrop basiert auf Ultraviolettabsorption. Das Prinzip der Ultraviolettabsorptionsmethode besteht darin, dass es in Nukleinsäurekomponenten Purin- und Pyrimidinbasen gibt und diese Basen konjugierte Doppelbindungen aufweisen. Im UV-Bereich gibt es eine starke Lichtabsorption bei 250–280 nm und der maximale Absorptionswert liegt bei etwa 260 nm. Daher kann die UV-Absorption von Nukleinsäure zur Konzentrationsquantifizierung verwendet werden. NanoDrop ist einfach und schnell zu bedienen, kann jedoch nicht zwischen DNA, RNA, degradierten Nukleinsäuren, freien Nukleotiden und anderen Verunreinigungen unterscheiden. Es ist nicht geeignet, um genaue quantitative Bibliotheksdaten zu erhalten. Die Qubit-Quantifizierung ist jedoch die beste Wahl für eine genaue NGS-Bibliotheksquantifizierung, insbesondere für die DNA-Quantifizierung. Was sind also die Merkmale der Qubit-DNA-Quantifizierung? Bitte sehen Sie sich die folgende Tabelle an:
| Spezifität | Fluoreszierende Farbstoffe binden bevorzugt dsDNA |
| Die Länge des DNA-Fragments | Keine besondere Einschränkung |
| Genauigkeit | Hoch |
| Anforderungen an die Stichprobengröße | Verdünnte Proben von 1–20 μl |
| Der Bereich der Nachweiskonzentration | 0.2-100ng |
| Die Anzahl der Normen | Normalerweise zwei |
| Experimentieren Sie mit der manuellen Betriebszeit | Stellen Sie die Zeit auf 5 Minuten und die Messzeit für eine einzelne Probe auf 3 s ein |
| Erkennungszeit | 5–30 Min. (je nach Probengröße) |
| Anwendbares Instrument | Qubit (3.0/4.0); Mikroplatten-Reader mit der entsprechenden Wellenlänge |
| Anwendbare Szene | dsDNA-Quantifizierung; dsDNA-Quantifizierung beim Aufbau einer NGS-Bibliothek und schnelle Quantifizierung in einer konventionellen Bibliothek |
2. Welche Eigenschaften haben die von Yeasen gelieferten Qubit-Produkte?
Bevor wir die Eigenschaften der von
2.1 Wie funktioniert Qubit?
Das Prinzip von Qubit besteht darin, fluoreszierende Farbstoffe zu verwenden, die sich an DNA-Moleküle binden, um Fluoreszenz zu emittieren, und dann die Fluoreszenzintensität zu messen, um die Konzentration der Nukleinsäure zu ermitteln. Fluoreszierende Farbstoffe fluoreszieren nur, nachdem sie an den DNA-Doppelstrang gebunden haben, und die resultierende Fluoreszenz ist proportional zur DNA-Konzentration.
2.2 Beliebtes Produkt von Yeasen
Der 1×dsDNA HS Assay Kit für Qubit® (Cat#: 12642ES), entwickelt auf Basis der Das dsDNA HS Assay Kit für das Qubit®-Kit (Kat.-Nr.: 12640ES) ist eine gebrauchsfertige Qubit-Vormischlösung zum Vorverdünnen der konzentrierten Farbstoffe im Originalkit zu einer Arbeitslösung. Beim Betrieb müssen Sie nur die Arbeitsflüssigkeit mit der zu testenden Probe mischen, um die Probenkonzentration mit dem Qubit®-Fluorometer zu bestimmen, was bequemer zu verwenden ist.
2.3 Produktmerkmale
Einfach und leicht zu bedienen: gebrauchsfertige, vorgemischte Flüssigkeit, keine Vorbereitung der Arbeitsflüssigkeit erforderlich, einfache Bedienung, quantitative, mehr Zeitersparnis;
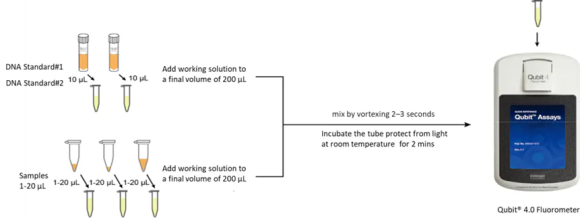
Abbildung 1. Die Prozesse des dsDNA HS Assay Kit für Qubit
Ultrahohe Empfindlichkeit: 64-fache Gradientenverdünnung, präzise Quantifizierung, 0,2–100 ng dsDNA hat eine gute lineare Beziehung;
Superspezifität: Spezifische Erkennung von dsDNA, hat eine ausgezeichnete Toleranz gegenüber einigen herkömmlichen Schadstoffen;
Schnelle Bindungsgeschwindigkeit des Farbstoffs: Die Sättigung kann innerhalb von 2 Minuten erreicht werden, sodass auf das schnelle Ablesen genauer quantitativer Ergebnisse keine Wartezeiten erforderlich sind;
Hohe Stabilität: Das Fluoreszenzsignal hielt 3 Stunden an und die quantitative Genauigkeit war nach 20 Tagen bei Raumtemperatur nicht beeinträchtigt.
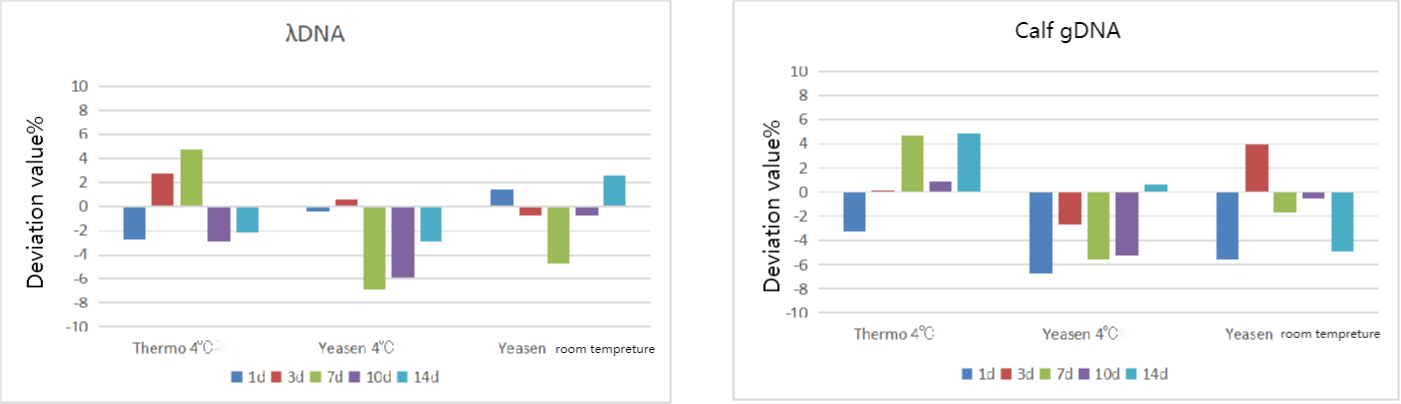
Abbildung 1. Der Produktstabilitätstest. Die Ergebnisse zeigen, dass es stabil nach 2-wöchiger Lagerung bei Raumtemperatur (die Abweichung zwischen gemessenem Wert und theoretischem Wert betrug <10%)
Hinweis: Es wurden zwei Konzentrationen von dsDNA ausgewählt und Thermo#Q33230 (gelagert bei 4 °C),
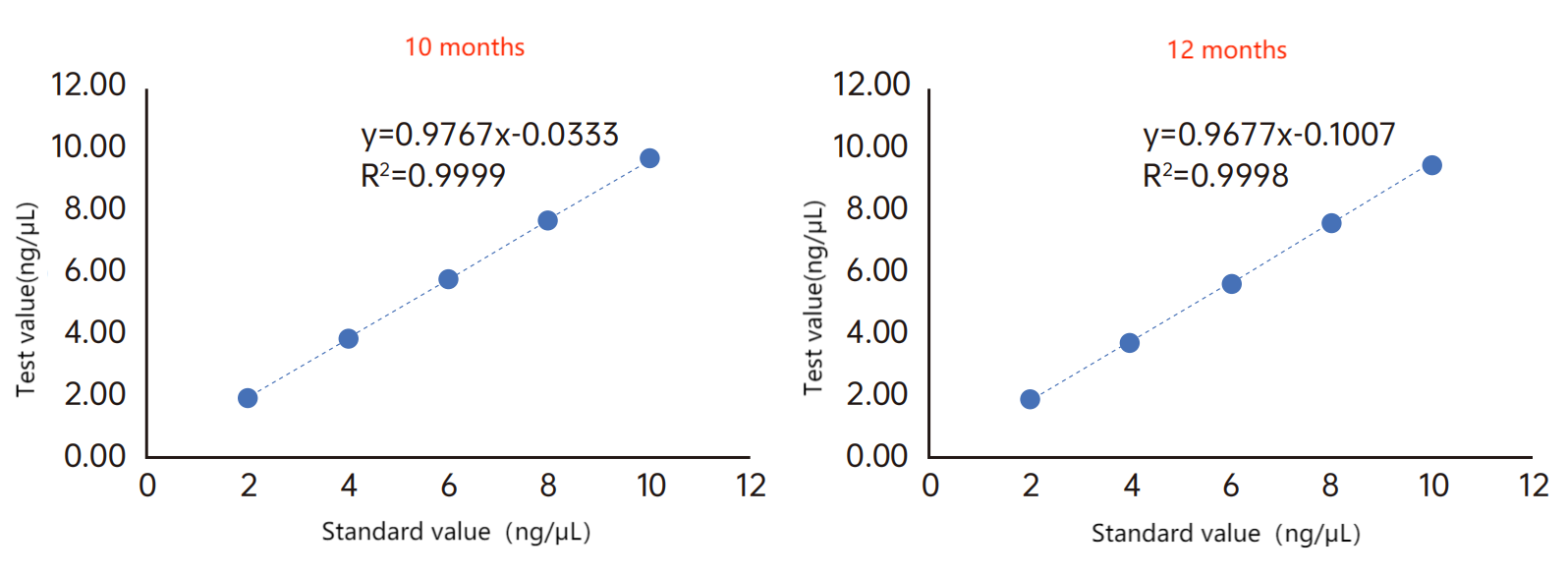
Abbildung 2. Produktstabilitätstest. Superstabilität, hohe Relevanz der Testergebnisse innerhalb der Gültigkeitsdauer
3. Wie wählt man Qubit für verschiedene Proben aus?
Qubit, schnelle Quantifizierung! Die Verwendung von Qubit zur Bibliotheksquantifizierung hat viele Vorteile. Sein Erkennungsprozess ist schnell und effektiv.Sequenzierungsergebnisse können für eine einzelne Probe innerhalb weniger Sekunden erhalten werden und die Konzentration kann direkt ausgegeben werden, was sehr intuitiv ist. Gleichzeitig ist es auch mit den meisten Enzymmarkierungsinstrumenten zur Batch-Probenquantifizierung kompatibel und ist die erste Wahl für die konventionelle Bibliotheksquantifizierung und dsDNA-Quantifizierung.
| Produktname | Katalog-Nr. | Größe | Anwendung |
| 1×dsDNA HS Assay Kit für Qubit | 12642ES | 100 t/500 t | dsDNA-Quantifizierung Bibliotheksquantifizierung |
| dsDNA HS Assay Kit für Qubit | 12640ES | 100 t/500 t | |
| ssDNA-Testkit | 12645ES | 100 t/500 t | ssDNA-Quantifizierung Bibliotheksquantifizierung |
