Wie wählen Sie Ihre NGS-Adapter aus?
Der NGS-Adapter, ein wesentlicher Bestandteil der Sequenzierungsbibliothek der nächsten Generation, spielt eine Rolle bei der Verbindung des getesteten DNA-Fragments und der Durchflusszelle (Sequenzierungschip). Die Effizienz der Verbindung ist ein wichtiger Faktor bei der Bestimmung der Qualität und Ausbeute der Bibliothek. Was ist also ein NGS-Adapter? Was sind die gängigen Arten von NGS-Adaptern? Und wie wählen Sie die richtigen NGS-Adapter für Ihre Sequenzierungsplattformen aus?
1. Was ist ein NGS-Adapter?
2. Welche Faktoren sollten Sie bei der Auswahl des NGS-Index berücksichtigen?
3. Was sind die gängigen Indextypen?
4. Was sind die gängigen Arten von NGS-Adaptern?
- UMI Adapter
- Komplette Adapter
- Unvollständige Adapter
- TN5-Adapter
5. Wie wählen Sie die richtigen NGS-Adapter für Ihre Sequenzierungsplattformen aus?
6. Zum Thema Lesen
1. Was ist ein NGS-Adapter?
Der NGS-Adapter, eine Reihe von Adaptern bei der Sequenzierung, ist eine kurze Nukleotidsequenz mit einer bekannten Sequenz. Er wird an beide Enden des Ziel-Nukleinsäurefragments ligiert. Während der Sequenzierung beginnt er mit der Sequenzierung, indem er mit der bekannten Sequenz auf der Durchflusszelle hybridisiert, um die Bibliothek mit dem Chip zu kombinieren. Wie ist also die Struktur des NGS-Adapters?
Am Beispiel der Illumina-Plattform kann ein NGS-Adapter in drei Teile unterteilt werden:
P5 und P7: Sequenz kombiniert mit P5- und P7-Enden auf der Durchflusszelle, um die Bibliothek auf dem Sequenzierungschip zu fixieren und so die Clusterreaktion durch Bridge-PCR zu erleichtern.
Rd1 SP und Rd2 SP (Read1/Read2-Sequenzierungsprimer): Bindungsbereiche von Sequenzierungsprimern, die die Position angeben, an der mit dem Lesen der Sequenz begonnen wird.
Index (auch als Barcode bekannt): eine bekannte synthetische Sequenz, die zur Unterscheidung verschiedener Proben bei der Sequenzierung der gemischten Bibliothek verwendet wird.
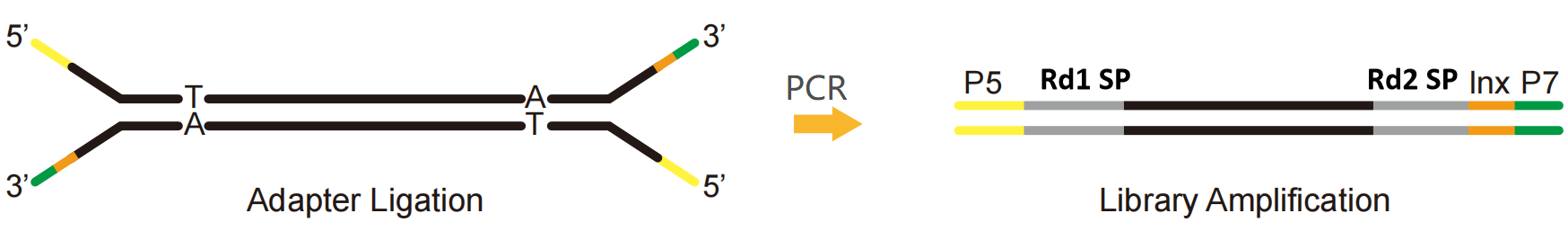
Feige. 1 Illumina-Plattform – Einzelindexbibliothek

Feige. 2 Single-End-Indexbibliothek der MGI-Plattform
Mit der Steigerung des Sequenzierungsdurchsatzes können mehrere Proben gleichzeitig sequenziert werden. Daher ist es besonders wichtig, unterschiedliche Proben voneinander zu unterscheiden. Wie bereits erwähnt, werden Indexsequenzen des NGS-Adapters verwendet, um verschiedene Proben beim Next-Generation-Sequencing (NGS) zu unterscheiden. Welche Faktoren sollten Sie bei der Auswahl des Index berücksichtigen? Bitte lesen Sie weiter...
2. Welche Faktoren sollten Sie bei der Indexauswahl berücksichtigen?
Der Index hat im Allgemeinen eine Länge von 6nt-18nt und wird je nach Anzahl der Indizes in einen Einzelindex und einen Doppelindex unterteilt. Der Doppelindex befindet sich an beiden Enden des zu testenden Fragments. Bei der Auswahl einer Indexkombination sollten das Basengleichgewicht und das Fluoreszenzgleichgewicht berücksichtigt werden.
Der Basissaldo bezieht sich auf den Saldo zwischen mehreren Indizes und nicht auf den Basissaldo in einem einzelnen Index. Er muss sowohl aus Basistypen als auch aus Basisverteilung betrachtet werden. Das Kombinationsprinzip besteht darin, dass die vier Basen A/T/C/G in derselben Indexgruppe enthalten sein müssen und der Anteil dieser vier Basen nahe beieinander liegt und jeweils etwa 25 % ausmacht.
Unter Fluoreszenzsignalbalance versteht man die Entscheidung, die Balance von Fluoreszenzsignalen sicherzustellen, wenn die Basisbalance nicht garantiert werden kann. Im 4-Kanal-Sequenzer der Illumina-Plattform ist dG/dT mit grüner Fluoreszenz und dC/dA mit roter Fluoreszenz markiert. Während der Sequenzierung müssen in jedem Zyklus sowohl grüne als auch rote Fluoreszenzsignale vorhanden sein, um eine erfolgreiche Sequenzierung sicherzustellen. Daher sollte bei der Auswahl des Index das Gleichgewicht zwischen dem grünen und dem roten Signal berücksichtigt werden.
3. Was sind die gängigen Indextypen?
Zu den gängigen Dualindizes zählen üblicherweise Unique Dual Index (UDI), Unique Dual Barcode (UDB) und Combined Dual Index (CDI), die Index-Hopping und Fehlzuweisungen deutlich reduzieren.
UDI&UDB: Die Indizes an beiden Enden entsprechen sich eins zu eins, sind in Gruppen angeordnet und können an beiden Enden gegenseitig überprüft werden.
Ein Stubby UDI Primer Kit für Illumina von
CDI: Die Indizes an beiden Enden können je nach Bedarf zu einer doppelseitigen Indexbibliothek kombiniert werden.
Ein 384 CDI Primer für Illumina, Set1-Set2, geliefert von
Um den Durchsatz und die Amplifikationseffizienz zu verbessern und die Sequenzierungskosten zu senken, führte Illumina die Clustertechnologie Array Flow Cell (PFCT) und Exclusive Amplification (ExAmp) für Novaseq und andere Hochdurchsatz-Sequenzer ein, verstärkte dabei jedoch unbeabsichtigt das Phänomen der Probenetiketten-Fehlpaarung und des Index-Hoppings.

Abb. 3 Verschiedene Illumina-Instrumentenmodelle verwenden eine ungemusterte oder eine gemusterte Durchflusszelle.
Um das Index-Hooping-Problem zu beheben, das bei Sequenzierungsplattformen wie HiSeq3000/4000, HiSeq X Series und NovaSeq auftritt, hat Illumina die Strategie vorgeschlagen, den Index an beiden Enden der Bibliothek zu platzieren, wodurch eine bilaterale Überprüfung durchgeführt und die nicht übereinstimmenden Adapter eliminiert werden können. Bei Verwendung der eindeutigen Indizes an beiden Enden wird die Index-Fehlerzuordnungsrate auf 0,01 % reduziert. Im Vergleich zur vorherigen herkömmlichen Methode zur Kombination von Indexpermutationsgruppen wird das Index-Hopping um zwei Größenordnungen reduziert.
Beim Aufbau einer PCR-freien Bibliothek steht ein Single-End-Indexadapter zur Verfügung. Die Label-Fehlpaarung wird hauptsächlich durch Sequenzierungsfehler verursacht. Insgesamt ist die Label-Fehlpaarungsrate gering (durchschnittlich 0,0004 %, bis zu 0,001 %). Beim Aufbau einer gezielten Capture-Bibliothek wird das Crosstalk-Problem jedoch verstärkt, da mehrere Schritte zu Label-Fehlpaarungen führen und normalerweise die UDI/UDB/CDI-Adapter verwendet werden.
4.Was sind die gängigen Arten von NGS-Adaptern?
Mit der Entwicklung der Sequenzierungstechnologie gibt es immer mehr Adaptertypen, wie z. B. Einzelindex-/Doppelindexadapter (wie in Abschnitt 3 erwähnt), UMI-Adapter, Transposaseadapter, vollständige/unvollständige Adapter usw., die für eine Vielzahl von Anwendungsszenarien geeignet sind. Dieser Teil sortiert diese Adapter systematisch, um Ihnen die Grundlage für die Adapterauswahl zu geben.
4.1 UMI-Adapter
Der Unique Molecular Identifier (UMI)-Adapter ist ein Spitzenwerkzeug für die Erkennung von Mutationen mit niedriger Frequenz und die absolute Quantifizierung. UMI ist eine zufällige synthetische Sequenz mit einer bekannten Sequenz. Sie kann als völlig zufällige Nukleotidkette, teilweise degenerierte Nukleotidkette oder feste Nukleotidkette gestaltet werden.Die Länge beträgt normalerweise 10 nt (einseitiger UMI) oder 5–8 nt (doppelseitiger UMI). Seine Funktion besteht darin, den Zustand von DNA-Fragmenten vor der Amplifikation einzufrieren, und jedes DNA-Molekül entspricht einem UMI. Daher kann es während der bioinformatischen Analyse DNA-Vorlagen aus verschiedenen Quellen unterscheiden, feststellen, welche Mutationen falsch positiv sind, die durch zufällige Fehler im Prozess der PCR-Amplifikation und -Sequenzierung verursacht wurden, und welche tatsächlich von Patienten getragen werden, um das Hintergrundrauschen herauszufiltern, die genaue Erkennung von Mutationen mit niedriger und extrem niedriger Frequenz zu realisieren und eine absolute Quantifizierung verschiedener DNA-Moleküle durchzuführen. Es wird häufig zur Erkennung von Mutationen mit niedriger Frequenz verwendet, insbesondere im Bereich der Tumorforschung.

Abb. 4 Schematische Darstellung des UMI-Adapters Struktur der Illumina-Plattform
4.2 Komplettadapter
Komplettadapter, ein notwendiges Produkt für eine PCR-freie Bibliothek, enthalten alle für Sequenzierungen erforderlichen Sequenzen wie P5, P7, RdS1 und RdS2 in der Illumina-Plattform, außerdem Indexsequenzen und UMI-Sequenzen gemäß den Anforderungen für die Sequenzierung. Mit den Komplettadaptern kann direkt sequenziert werden, ohne dass andere Adapter durch PCR eingeführt werden müssen. Komplettadapter können also zum Aufbau einer PCR-freien Bibliothek verwendet werden. Die PCR-freien Bibliotheken können PCR-Amplifikationsbias, Fehlerrate und Sequenzduplizierung reduzieren und so die Abdeckung einiger Regionen mit hohem GC oder hohem AT erhöhen, die in der Populationsgenomforschung weit verbreitet sind.
Ein komplettes Adapterprodukt für die Illumina-Plattform von
Ein komplettes Adapterprodukt für die MGI-Plattform von
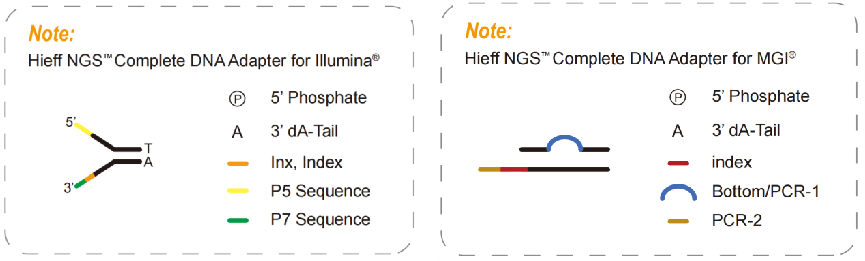
Abb. 5 Vollständiges Adapterdiagramm
4.3 Unvollständige Adapter
Unvollständige Adapter müssen nach der Adapterligation andere Sequenzen per PCR einführen, um einen vollständigen Adapter zu bilden. Sie zeichnen sich durch eine hohe Verbindungseffizienz und eine hohe effektive Bibliotheksrate aus. Der PCR-Prozess ist ein Anreicherungseffekt für die vollständige Bibliothek, um die Konzentration der effektiven Bibliothek sicherzustellen, und kann auch doppelendige Indizes und UMI-Sequenzen einführen.
4.4 Tn5-Adapter
Tn5-Adapter verbinden einen Teil der Adaptersequenz über die Restriktionsendonukleaseaktivität von Tn5 mit beiden Enden der DNA-Fragmente. Sie ermöglichen die gleichzeitige Durchführung von Fragmentierung und Adapterligation, um Zeit und Proben zu sparen. Abschließend werden der Rest der Linkersequenz, der Index, UMI und andere Sequenzen durch PCR eingeführt, um eine vollständige Bibliothek zu bilden. Sie kann zum Aufbau einer Cut&Tag-Bibliothek verwendet werden.
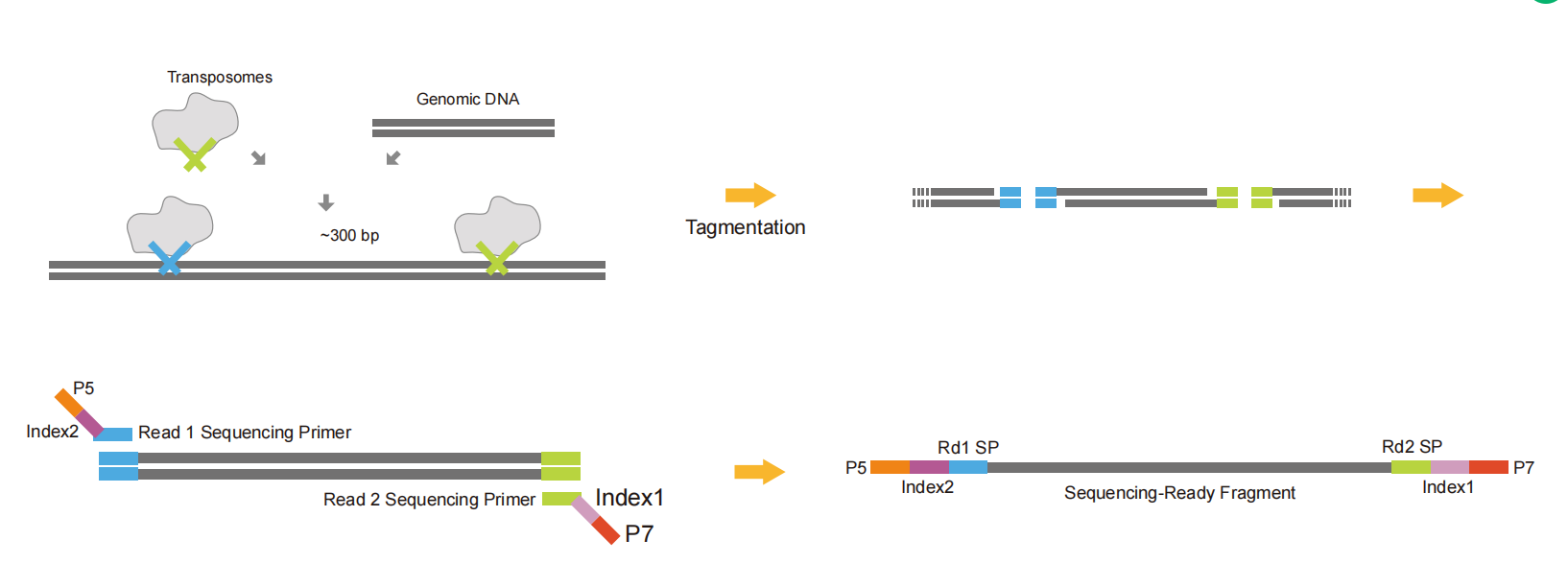
Abb. 6 Schematische Darstellung des Aufbaus der Tn5-Adapterbibliothek
5. Wie wählen Sie die richtigen NGS-Adapter für Ihre Sequenzierungsplattformen aus?
Derzeit gibt es zwei gängige Sequenzierungsplattformen, nämlich Illumina und MGI.
In Bezug auf die Illumina-Plattform gibt es von
Bei Komplett- und UDI-NGS-Adaptern müssen Sie sich keine Gedanken über Kupplungsprobleme machen, geeignet für Kunden, die Wert auf einfache Handhabung legen; CDI-NGS-Adapter haben weniger Schläuche und sind klein, was für Kunden geeignet ist, die sie einfach aufbewahren und transportieren möchten. Für PCR-freie Adapter müssen Komplett-NGS-Adapter verwendet werden.
Für Illumina
| InTube | Hieff NGS® DNA Lib Vorbereitung 384 CDI-Primer für Illumina, Set 1 (8*12, 96 Index) | 12412ES |
| Hieff NGS® DNA Lib Vorbereitung 384 CDI Primer für Illumina, Set 2 (8*12, 96 Index) | 12413ES | |
| Hieff NGS® RNA Lib Prep 384 CDI Primer für Illumina, Set 1 (96 Index) | 12414ES | |
| Hieff NGS® RNA Lib Prep 384 CDI Primer für Illumina, Set 1 (96 Index) | 12415ES | |
| In Platte | Hieff NGS® Stubby UDI Primer Kit für Illumina (1-384 Index) Set 1-4 | 12407ES |
| Hieff NGS® Stubby UDI Primer Kit für Illumina Satz 1(96-Well-Platte, 1-96 Index) Satz 1 | 12327ES | |
| Hieff NGS® Stubby UDI Primer Kit für Illumina Satz 2(96-Well-Platte, 97-192 Index) Satz 2 | 12328ES | |
| Hieff NGS® Stubby UDI Primer Kit für Illumina Satz 3(96-Well-Platte, 193-288 Index) Satz 3 | 12329ES | |
| Hieff NGS® Stubby UDI Primer Kit für Illumina Satz 4(96-Well-Platte, 289-384 Index) Satz 4 | 12330ES |
Für MGI
| Hieff NGS™ Duales UMI UDB-Adapterkit für MGI, Set1/Set2 | 96 Arten von Indizes | |
| Hieff NGS™ Komplettes Adapterkit für MGI, Set1/Set2/Set3 (Anfrage) | 13360ES | 8 Arten von Index, 41-48 |
| 13361ES | 16 Arten von Index, 57-72 | |
| 13362ES | 96 Indexarten, 1-96 |
Zum Thema Lesen
Wichtige Enzyme beim Aufbau der NGS-Bibliothek
Wie gut wissen Sie über NGS-bezogene Technologie?
Verschiedene Arten von Magnetkügelchen in NGS: DNA\RNA\mRNA-Magnetkügelchen
Quantifizierung der NGS-Bibliothek: Schnelles und genaues Qubit oder präzise qPCR? Alles erforderlich!
