Beschreibung
2× Hieff Canace™ AdvanceFast PCR Master Mix (mit Farbstoff) ist eine gebrauchsfertige 2× Vormischung mit Hieff Canace™ AdvanceFast High-Fidelity DNA-Polymerase, dNTPs und ein optimiertes Puffersystem, das bereits hinzugefügte Elektrophoreseindikatoren enthält. Der Premix enthält bereits hinzugefügte Elektrophoreseindikatoren, PCR-Produkte können direkt elektrophoretisch aufgetrennt werden, die Amplifikationsprodukte haben flache Enden. 2× Hieff Canace™ AdvanceFast PCR Master Mix (mit Farbstoff) bietet die Vorteile von Schnelligkeit und Einfachheit, hoher Sensitivität, hoher Spezifität, guter Stabilität usw. Das Reaktionssystem kann nur mit den Primern und Vorlagen hinzugefügt werden. Darüber hinaus enthält das Produkt auch ein spezielles Schutzmittel, sodass die Vormischung auch nach wiederholtem Einfrieren und Auftauen noch eine stabile Aktivität behält.
Ultrahohe Wiedergabetreue: Niedrige Fehlpaarungsrate bei mutationsanfälligen hohes GC-Gen;
Schnelle Geschwindigkeit: Die Geschwindigkeit ist so schnell wie 5 Sek./KB;
Breite Anwendbarkeit: Es kann zur Amplifikation von Genen mit 20–80 % GC-Gehalt verwendet werden und verträgt 20 % Blut und Mauslysate.
Gute Stabilität: 7 Tage bei 37 °C gelagert, ohne dass die Leistung beeinträchtigt wird;
Einfache Anwendung: Fügen Sie einfach Primer und Vorlagen zur Amplifikation hinzu, mit blauem Ladefarbstoff für die direkte Elektrophorese.
Technische Daten
| Kat.-Nr. | 10164ES01 | 10164ES03 | 10164ES08 |
| Größe | 250 μl | 1 ml | 5×1 ml |
Zahlen
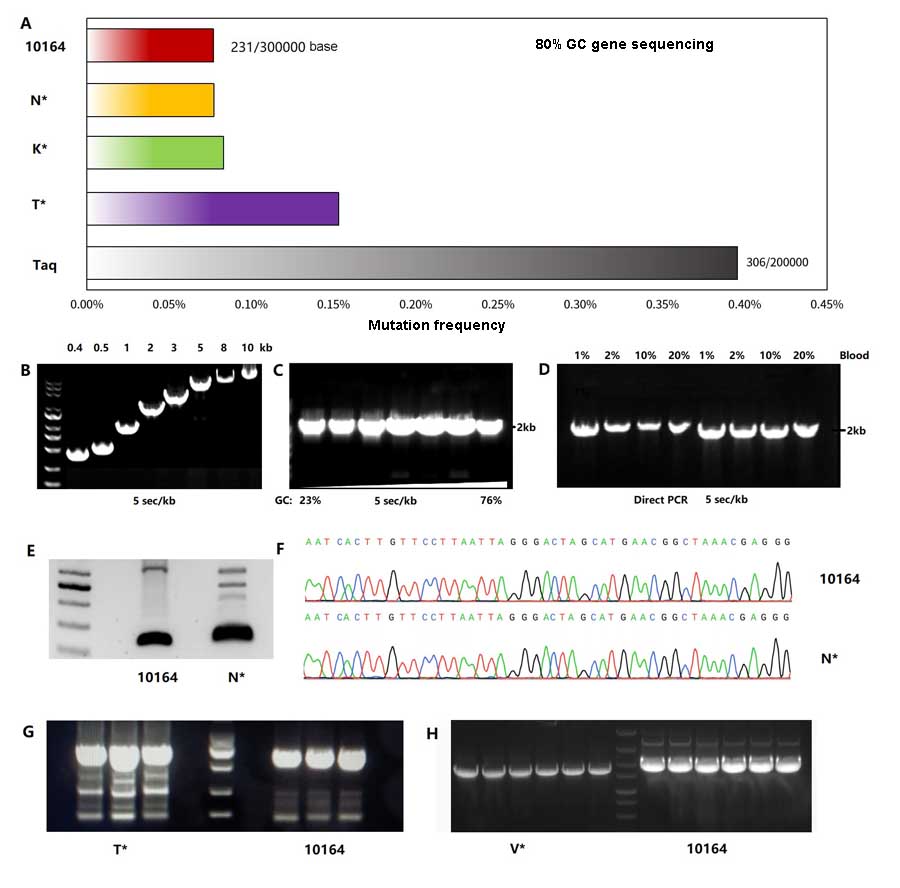
Abbildung: Hieff Canace™ AdvanceFast High-Fidelity DNA-Polymerase weist im Vergleich zu Produkten anderer Marken eine hohe Wiedergabetreue auf (A). Der Test wurde durchgeführt, indem die Mutationshäufigkeit von PCR-Produkten von 6 GC-reichen (ca. 80 %) Segmenten untersucht wurde, die aus den Genomen verschiedener Arten amplifiziert wurden. Die durch Hieff Canace™ AdvanceFast High-Fidelity DNA-Polymerase amplifizierten PCR-Produkte wurden in Vektoren geklont und 100 ausgewählte Einzelklone für jede Sequenz wurden einer Sanger-Sequenzierung unterzogen.
Hieff Canace™ AdvanceFast High-Fidelity DNA-Polymerase verfügt über eine hohe Amplifikationsgeschwindigkeit (B), eine hohe GC-Amplifikationskompatibilität (C) und Blutverträglichkeit (D). Ergebnisse aus der PCR und Sequenzierung von Kunden zeigen, dass die Hieff Canace™ AdvanceFast High-Fidelity DNA-Polymerase gute Leistungen bei PCR-Ausbeute und Spezifität (EH) aufweist.

Lagerung
Dieses Produkt sollte 1 Jahr lang bei -25–15 °C gelagert werden.
Anweisungen
- Empfohlene PCR-Reaktionssysteme.
| Komponenten | Volumen (μL) | Finale Konzentration |
| 2× Hieff Canace™ AdvanceFast PCR Master Mix (mit Farbstoff)* | 25 | 1× |
| Vorlage** | X | - |
| Nach vorne Grundierung (10 μmol/l)*** | 2 | 0,4 μmol/l |
| Umkehren Grundierung (10 μmol/l) | 2 | 0.4 μmol/l |
| ddH2O | Bis zu 50 | - |
Tabelle 1 PCR-Reaktionssystem
*In 1× Vormischungen mit 2 mM Mg2+ und 200 μM dNTPs.
**Empfohlener Bereich 10–200 ng, Upload-Volumenbereich der cDNA-Probe nicht mehr als 1/10 des Reaktionssystems, empfohlen 1–2,5 μL.
***Die endgültige Primerkonzentration im PCR-Reaktionssystem liegt zwischen 0,2 und 1 μM, empfohlen werden 0,4 μM.
- Reaktionsprogramm.
| Zyklus Schritt | Temperatur. | Zeit | Zyklen |
| Erste Denaturierung | 98℃ | 30 Sek. | 1 |
| Denaturierung | 98℃ | 10 Sek. | 30-35 |
| Glühen* | 60℃ | 5 Sek. | |
| Verlängerung** | 72℃ | 5–10 Sek./KB | |
| Finale Verlängerung | 72℃ | 2 Minuten | 1 |
Tabelle 2 PCR-Reaktionsprogramm
*Empfohlene Temperatur: 60°C, ein Temperaturgradient kann eingestellt werden, um die optimale Temperatur für das Primer-Annealing zu finden. Die empfohlene Annealing-Zeit ist auf 5 Sekunden eingestellt und kann von 5-30 Sekunden angepasst werden. Eine zu lange Annealing-Zeit kann zu diffusen Amplifikationsprodukten auf dem Gel führen.
**Verlängerungszeit: Empfohlen 5 Sek./kb, kann bei Bedarf auch auf 10 Sek./kb verlängert werden.
Hinweise
- Dieses Produkt ist nur für Forschungszwecke bestimmt.
- Bitte arbeiten Sie zu Ihrer Sicherheit mit Laborkitteln und Einweghandschuhen.
Unterlagen:
Zitate und Referenzen:
[1] Dong W, Zhu Y, Chang H, et al. Ein SHR-SCR-Modul spezifiziert das Schicksal von Hülsenfruchtkortikalzellen, um die Nodulation zu ermöglichen. Natur. 2021;589(7843):586-590. doi:10.1038/s41586-020-3016-z(WENN:42.779)
[2] Sun L, Yan Y, Lv H, et al. Rapamycin zielt auf STAT3 ab und beeinflusst c-Myc, um das Tumorwachstum zu unterdrücken. Cell Chem Biol. 2022;29(3):373-385.e6. doi:10.1016/j.chembiol.2021.10.006(WENN:8.116)
[3] Ye M, Xiong L, Dong Y, et al. Die potenzielle Rolle des Methionin-Aminopeptidase-Gens PxMetAP1 in einem kosmopolitischen Schädling für die Toxintoleranz von Bacillus thuringiensis. Int J Mol Sci. 2022;23(21):13005. Veröffentlicht am 27. Oktober 2022. doi:10.3390/ijms232113005(IF:6.208)
[4] He Y, Zhou J, Fu R, et al. Die Anwendung von DNA-HRP-funktionalisierten AuNP-Sonden zur kolorimetrischen Erkennung von Zitrus-assoziierten Alternaria-Genen. Talanta. 2022;237:122917. doi:10.1016/j.talanta.2021.122917(WENN:6.057)
[5] Guo L, Yang W, Huang Q, et al. Selenocysteinspezifische Massenspektrometrie enthüllt gewebespezifische Selenoproteome und Kandidaten-Selenoproteine. Cell Chem Biol. 2018;25(11):1380-1388.e4. doi:10.1016/j.chembiol.2018.08.006(WENN:5.592)
[6] Dou W, Zhu Q, Zhang M, Jia Z, Guan W. Screening und Bewertung der starken endogenen Promotoren in Pichia pastoris. Fakten über Mikrobenzellen. 2021;20(1):156. Veröffentlicht am 9. August 2021. doi:10.1186/s12934-021-01648-6(WENN:5.328)
[7] Xiong Y, Yi Y, Wang Y, Yang N, Rudd CE, Liu H. Ubc9 interagiert mit dem TCR-Adapter SLP-76 und SUMOyliert ihn für die NFAT-Transkription in T-Zellen. J Immunol. 2019;203(11):3023-3036. doi:10.4049/jimmunol.1900556(WENN:4.718)
[8] Huang L, Xiang M, Ye P, Zhou W, Chen M. Beta-Catenin fördert die durch Makrophagen vermittelte akute Entzündungsreaktion nach einem Herzinfarkt. Immunol Cell Biol. 2018;96(1):100-113. doi:10.1111/imcb.1019(WENN:4.557)
[9] Wang X, Du A, Yu G, Deng Z, He X. Guanidin-N-Methylierung durch BlsL ist abhängig von der Acylierung von Beta-Amin-Arginin in der Biosynthese von Blasticidin S. Front Microbiol. 2017;8:1565. Veröffentlicht am 22. August 2017. doi:10.3389/fmicb.2017.01565(WENN:4.076)
[10] Yu P, Zhou L, Yang WT, et al. Vergleichende Mitogenomanalysen decken Mitogenommerkmale und phylogenetische Implikationen der Unterfamilie Cobitinae auf. BMC Genomics. 2021;22(1):50. Veröffentlicht am 14. Januar 2021. doi:10.1186/s12864-020-07360-w(WENN:3.969)
[11] Liu G, Liu Y, Niu B, et al. Genetische Mutation von TRPV2 induziert Angst durch Verringerung der GABA-B R2-Expression im Hippocampus. Biochem Biophys Res Commun. 2022;620:135-142. doi:10.1016/j.bbrc.2022.06.079(WENN:3.575)
[12] Tan KS, Zhang Y, Liu L, et al. Molekulare Klonierung und Charakterisierung eines atypischen Butyrylcholinesterase-ähnlichen Proteins im Zebrafisch. Comp Biochem Physiol B Biochem Mol Biol. 2021;255:110590. doi:10.1016/j.cbpb.2021.110590(WENN:2.231)
Zahlung und Sicherheit
Ihre Zahlungsinformationen werden sicher verarbeitet. Wir speichern weder Kreditkartendaten noch Zugriff auf Ihre Kreditkarteninformationen.
Anfrage
Sie können auch mögen
FAQ
Das Produkt ist nur für Forschungszwecke bestimmt und nicht für die therapeutische oder diagnostische Anwendung bei Menschen oder Tieren. Produkte und Inhalte sind durch Patente, Marken und Urheberrechte von
Für bestimmte Anwendungen sind möglicherweise zusätzliche geistige Eigentumsrechte Dritter erforderlich.