Beschreibung
Produktbeschreibung
Das Hieff NGS™ Ultima Dual-mode RNA Library Prep Kit ist ein Kit zum Aufbau einer vollständigen RNA-Sequenzierungsbibliothek für die Sequenzierungsplattformen Illumina® und MGI®, das Reagenzien zur RNA-Fragmentierung, Reagenzien zur Reversen Transkription, konventionelle und strangspezifische ds-cDNA-Synthesereagenzien sowie Reagenzien zur Bibliotheksverstärkung enthält. Die Sequenzierungsbibliothek kann aufgebaut werden, gefolgt vom mRNA-Reinigungskit oder dem rRNA-Entfernungskit. Das Zweistrangsynthesemodul ist mit zwei Puffern ausgestattet, um den Bedarf an einer konventionellen oder strangspezifischen Bibliothek zu decken. Unter ihnen wird dTTP im strangspezifischen Zweistrangsynthesepuffer durch dUTP ersetzt, sodass dUTP dem zweiten Strang der cDNA hinzugefügt werden kann. Die in diesem Kit verwendete hochpräzise DNA-Polymerase kann die DNA-Vorlage, die Uracil enthält, nicht verstärken, wodurch Strangspezifität erreicht wird. Alle bereitgestellten Reagenzien wurden einer strengen Qualitätskontrolle und Funktionsüberprüfung unterzogen, wodurch die Stabilität und Reproduzierbarkeit des Bibliotheksaufbaus in höchstem Maße sichergestellt wird.
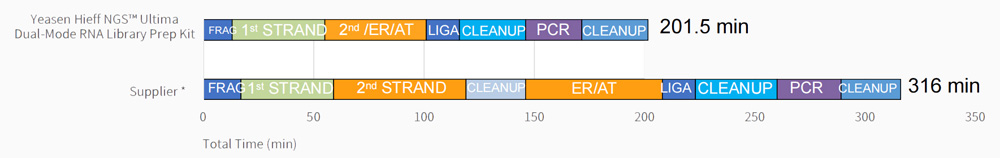
Arbeitsablauf
Produktkomponenten
| Komponenten | 12308ES24 | 12308ES96 | |||
| 12308-A | 2× Frag/Prime-Puffer | 250 μl | 930 μl | ||
| 12308-B | 1. Strang Enzymmischung | 48 μl | 192 μl | ||
| 12308-C | Strangspezifitätsreagenz | 150 μl | 580 μl | ||
| 12308-D | 2. Strangpuffer (dNTP) | 720 μl | 2×1440 μL | ||
| 12308-E | 2. Strangpuffer (dUTP) | 720 μl | 2×1440 μL | ||
| 12308-F | 2. Strang Enzym Master Mix | 120 μl | 480 μl | ||
| 12308-G | Ligationsverstärker | 720 μl | 2×1440 μL | ||
| 12308-H | Neue T4 DNA Ligase | 120 μl | 480 μl | ||
| 12308-I | 2×Super Canace® II Hi-Fi-Mix | 600 μl | 2×1200 μL | ||
| 12308-K | Nukleasefreies H2O | 300 μl | 1000 μl | ||
Hinweis: Dieses Kit ist sowohl mit Illumina- als auch mit MGI-Plattformen kompatibel, aber zusätzliche Illumina oder MGI Primer Mix (Kat.-Nr. 13335 Primer Mix für Illumina und Kat.-Nr. 13334 Primer Mix für MGI ) Ist erforderlich.
Versand und Lagerung
Die Komponenten des Hieff NGS™ Ultima Dual-mode mRNA Library Prep Kit in Box I werden mit Kühlakkus geliefert und können ein Jahr lang bei 2–8 °C gelagert werden.
Die Komponenten des Hieff NGS™ Ultima Dual-mode mRNA Library Prep Kit in Box II werden mit Trockeneis geliefert und können ein Jahr lang bei -20 °C gelagert werden.
Vorsichtsmaßnahmen
1 Bedienung
1.1 Tragen Sie zu Ihrer Sicherheit und Gesundheit beim Umgang mit diesem Produkt persönliche Schutzausrüstung (PSA), wie Laborkittel und Einweghandschuhe. Dieses Produkt ist NUR für Forschungszwecke bestimmt!
1.2 Komponenten bei Zimmertemperatur auftauen. Durch mehrmaliges Auf- und Abschütteln gründlich mischen, kurz abzentrifugieren und zur Verwendung auf Eis legen.
1.3 Es wird empfohlen, jede Schrittreaktion in einem Thermocycler mit beheiztem Deckel durchzuführen. Der Thermocycler sollte vor der Verwendung auf die eingestellte Temperatur vorgeheizt werden.
1.4 Es ist notwendig, dass die Verbrauchsmaterialien frei von RNase-Verunreinigungen sind und der Versuchsbereich regelmäßig gereinigt wird.
1.5 Unsachgemäße Handhabung kann sehr wahrscheinlich zu Aerosolverunreinigungen führen, die die Genauigkeit des Ergebnisses beeinträchtigen. Eine obligatorische physische Isolierung der PCR-Reaktionsmischbereiche und der PCR-Produktreinigungstestbereiche wird empfohlen. Ausgestattet mit Geräten wie Spezialpipetten für den Bibliotheksaufbau.
2 Adapterligatur
2.1 Es stehen Illumina- oder MGI-Langadapter-Kits (Adapter mit Strichcode) und Kurzadapter-Kits zur Auswahl, je nach den Anforderungen der Kunden beim Experimentieren.
2.2 Es wird empfohlen, hochwertige, kommerzielle Adapter auszuwählen. Wenn Sie sich für selbst hergestellte Adapter entscheiden, vertrauen Sie diese bitte einem Unternehmen mit Erfahrung in der NGS-Primersynthese an und weisen Sie auf die Notwendigkeit einer strengen Kontaminationskontrolle hin. Darüber hinaus wird empfohlen, die DNA-Annealing-Lösung in einer Reinraumbank vorzubereiten und jedes Mal nur einen Adaptertyp zu verwenden, um Kreuzkontaminationen zu vermeiden.
2.3 Tauen Sie die Adapter bitte auf Eis oder bei 4°C auf, bei Betrieb bei Raumtemperatur sollte die Labortemperatur 25°C nicht überschreiten, um eine Denaturierung der Adapter zu vermeiden.
2.4 Die Konzentration des Adapters wirkt sich direkt auf die Ligationseffizienz und die Bibliotheksausbeute aus. Das dem Kit hinzugefügte Adaptervolumen ist auf 5 μl festgelegt. Es wird empfohlen, die Adapter mit 0,1 × TE-Puffer zu verdünnen. Die verdünnten Adapter können 48 Stunden lang bei 4 °C gelagert werden. Tabelle 1 listet die empfohlene Adaptermenge für unterschiedliche Mengen an Eingangs-RNA auf.
Tabelle 1-1 Die empfohlene Illumina-Adaptermenge für verschiedene Eingangs-RNA
| Eingabe Gesamt-RNA | Illumina Adapterbestandskonzentration |
| 10 ng | 1 μM |
| 100 ng | 1,5 μM |
| 500 ng | 3 μM |
| ≥1 μg | 5 μM |
Tabelle 1-2 Die empfohlene MGI®-Adaptermenge für verschiedene Eingangs-RNA
| Eingabe Gesamt-RNA | MGI Adapterbestandskonzentration |
| 100-499 ng | 2 μM |
| 500-4000 ng | 5 μM |
*Die Adapternutzung kann je nach Art der Gesamt-RNA-Proben und Eingabemenge angepasst werden.
3 Bibliotheksverstärkung
3.1 Auf der Grundlage der DNA-Polymerase der ersten Generation weist die hochpräzise DNA-Polymerase im Kit eine stark verbesserte Amplifikationsgleichmäßigkeit auf und weist keinen Amplifikationsbias auf.
3.2 Wenn ein indizierter Adapter (auch als langer Adapter oder großer Y-Adapter bezeichnet) an die Ziel-DNA ligiert wird, kann die in diesem Kit enthaltene Primermischung zur Amplifikation verwendet werden; wenn ein „kurzer Adapter“ oder „kleiner Y-Adapter“ zur DNA-Ligation verwendet wird, werden Indexprimer zur Amplifikation benötigt.
3.3 Die Anzahl der Amplifikationszyklen sollte streng kontrolliert werden. Unzureichende Amplifikation kann zu einer geringen Bibliotheksausbeute führen; Überamplifikation kann zu erhöhter Verzerrung, Fehlern, doppeltem Lesen, chimären Produkten und der Ansammlung von Expansionsmutationen führen. Tabelle 2 listet die empfohlenen Zyklenzahlen für die PCR-Amplifikation auf.
Tabelle 2 Die empfohlene Anzahl von Zyklen zur Generierung einer RNA-Bibliothek*
| Eingabe Gesamt-RNA | Anzahl der Zyklen | |
| Nicht verseilt | Gestrandet | |
| 10 ng | 15 | 15 |
| 100 ng | 14 | 14 |
| 500 ng | 12 | 13 |
| 1 μg | 11 | 12 |
Hinweis:*Die Ausbeute der Bibliothek hängt nicht nur von der Eingangsmenge und der Anzahl der Amplifikationszyklen ab sondern auch von der Qualität der Proben, den Fragmentierungsbedingungen und den Sortierbedingungen beeinflusst. Wählen Sie beim Aufbau der Bibliothek die am besten geeigneten Bedingungen entsprechend der tatsächlichen Situation aus.
4 Bead-basierte DNA-Reinigung und Größenauswahl
4.1 Es gibt mehrere Schritte im Bibliotheksaufbauprozess, die magnetische Perlen zur DNA-Reinigung erfordern. Wir empfehlen Hieff NGS™ DNA Selection Beads (Yeasen Cat#12601) oder AMPure® XP magnetische Perlen (Beckman Cat#A63880) zur DNA-Reinigung und Größenauswahl.
4.2 Die magnetischen Perlen sollten vor der Verwendung auf Raumtemperatur gebracht werden, da sonst die Ausbeute abnimmt und der größenselektierende Effekt beeinträchtigt wird.
4.3 Die magnetischen Perlen sollten vor der Verwendung durch Vortexen oder Pipettieren gut gemischt werden.
4.4 Saugen Sie die Perlen beim Übertragen des Überstands nicht ab, selbst kleinste Mengen der Perlen können die folgenden Reaktionen beeinträchtigen.
4.5 Der 80 %ige Ethanol muss frisch zubereitet werden, da sonst die Rückgewinnungseffizienz beeinträchtigt wird.
4.6 Die Magnetkügelchen sollten vor dem Eluieren des Produkts bei Raumtemperatur getrocknet werden. Bei unzureichender Trockenheit können Ethanolrückstände leicht nachfolgende Reaktionen beeinträchtigen; bei übermäßiger Trockenheit reißen die Magnetkügelchen und die Reinigungsausbeute wird verringert. Normalerweise reicht es aus, die Kügelchen 3–5 Minuten bei Raumtemperatur trocknen zu lassen, damit sie vollständig trocknen.
4.7 Bei Bedarf können die gereinigten oder nach Größe selektierten und im TE-Puffer eluierten DNA-Proben 1–2 Wochen bei 4 °C oder einen Monat bei -20 °C gelagert werden.
5 Analyse der Bibliotheksqualität
5.1 Normalerweise kann die Qualität der erstellten Bibliothek durch Längenverteilung und Konzentrationsbestimmung bewertet werden.
5.2 Erkennung der Bibliothekskonzentration: Methoden basierend auf Fluoreszenzfarbstoffen für doppelsträngige DNA, wie Qubit®, PicoGreen® usw.; absolute Quantifizierung basierend auf qPCR.
5.3 Auf Spektraldetektion basierende Methoden wie NanoDrop® usw. sind für die Erkennung von Bibliothekskonzentrationen nicht anwendbar.
5.4 qPCR wird zur Ermittlung der Bibliothekskonzentration empfohlen: Mit Qubit®, PicoGreen® und anderen Methoden, die auf Fluoreszenzfarbstoffen für doppelsträngige DNA basieren, kann nicht effektiv zwischen Produkten unterschieden werden, die an einem Ende an Adapter ligiert sind, Produkten, die nicht an beiden Enden an Adapter ligiert sind, und anderen unvollständigen doppelsträngigen Produkten. Die absolute Quantifizierung von qPCR basiert auf dem Prinzip der PCR-Amplifikation, die nur die vollständige Bibliothek des Adapters an beiden Enden der Probe quantifiziert (die Bibliothek, die sequenziert werden kann), und die Interferenz von nicht sequenzierbaren Bibliotheken ausschließt, die weder an einem noch an einem doppelseitigen Ende an den Adapter ligiert sind.
5.5 Die Erkennung der Bibliothekslängenverteilung kann mit dem Agilent Bioanalyzer 2100 und anderen Geräten durchgeführt werden, die auf dem Prinzip der Kapillarelektrophorese oder Mikrofluidik basieren.
Unterlagen:
12308ES-Hieff NGS™ Ultima Dual-Mode RNA-Bibliotheksvorbereitungskit-Ver.EN20230327.pdf
Zitate und Referenzen:
[1] Entschlüsselung des Darmmikrobioms von Graskarpfen durch einen Multi-Omics-Ansatz
M Li, H Liang, H Yang, Q Ding, R Xia, J Chen, W Zhou… - Mikrobiom, 2024 IF:19.4
[2] Gepooltes CRISPR-Screening identifiziert P-Bodies als Repressoren der epithelial-mesenchymalen Transition bei Krebs
L Fang, L Zhang, M Wang, Y He, J Yang, Z Huang… – Krebsforschung, 2024 IF:12.7
[3] Klotho-abgeleitetes Peptid 1 hemmt die zelluläre Seneszenz in der fibrotischen Niere, indem es die Klotho-Expression über posttranskriptionelle Regulation wiederherstellt
X Zhang, L Li, H Tan, X Hong, Q Yuan, FF Hou… - Theranostik, 2024 IF:12.4
[4] Exoskelett‐partiell beschichtete Stammzellen zur Wiederherstellung des infarktbedingten Myokards
H He, Y Yuan, Y Wu, J Lu, X Yang, K Lu… – Advanced Materials, 2023 IF: 29.4
[5] Genomische Innovation und regulatorische Neuverdrahtung während der Evolution der Baumwollgattung Gossypium
M Wang, J Li, Z Qi, Y Long, L Pei, X Huang… - Nature Genetics, 2022 IF:41.379
[6] MTMR3-Risikoallele verstärken die durch Toll Like Receptor 9 induzierte IgA-Immunität bei IgA-Nephropathie
Y Wang, T Gan, S Qu, L Xu, Y Hu, L Liu, S Shi, J Lv… – Kidney International, 2023 IF:19.6
[7] Aufgeteilte Komplementierung von Basiseditoren, um Off-Target-Editierungen zu minimieren
X Xiong, K Liu, Z Li, FN Xia, XM Ruan, X He, JF Li - Naturpflanzen, 2023 IF:18.6
[8] Künstlich erzeugte bakterielle Außenmembranvesikel, die onkolytische Adenoviren einkapseln, steigern die Wirksamkeit der Krebsvirotherapie durch eine Verstärkung der Tumorzellautophagie.
W Ban, M Sun, H Huang, W Huang, S Pan… - Nature Communications, 2023 IF:16.6
[9] Eine Resistenz der Krebszellen gegen IFNγ kann über eine erhöhte Aktivität des Doppelstrangbruch-Reparaturweges entstehen
T Han, X Wang, S Shi, W Zhang, J Wang, Q Wu… - Cancer Immunology Research, 2023 IF: 12.0
[10] Kombinationstherapie basierend auf einem dualen biomimetischen Nano-Delivery-System zur Überwindung der Cisplatinresistenz beim hepatozellulären Karzinom
Y Huang, Q Kou, Y Su, L Lu, X Li, H Jiang… – Journal of Nanobiotechnology, 2023 IF: 10.9
[11] PIAS3 fördert die Ferroptose durch Regulierung von TXNIP über den TGF-β-Signalweg im hepatozellulären Karzinom
W Bao, J Wang, K Fan, Y Gao, J Chen - Pharmakologische Forschung, 2023 IF:9.3
[12] Identifizierung von RNA-bindenden Proteintargets mit HyperTRIBE in Saccharomyces cerevisiae
W Piao, C Li, P Sun, M Yang, Y Ding, W Song… – International Journal of Molecular Science, 2023 IF:5.6
[13] Mikrobiota reguliert Lebenszyklusübergänge und Nematozytendynamik bei Quallen
S Peng, L Ye, Y Li, F Wang, T Sun, L Wang, W Hao… - Iscience, 2023 IF: 5.08
[14] Transkriptom- und miRNA-Profile enthüllen regulatorisches Netzwerk und Schlüsselregulatoren der sekundären Xylembildung in der „84K“-Pappel
H Wang, P Zhao, Y He, Y Su, X Zhou… - International Journal of Molecular Science, 2023 IF:5.6
Zahlung und Sicherheit
Ihre Zahlungsinformationen werden sicher verarbeitet. Wir speichern weder Kreditkartendaten noch Zugriff auf Ihre Kreditkarteninformationen.
Anfrage
Sie können auch mögen
FAQ
Das Produkt ist nur für Forschungszwecke bestimmt und nicht für die therapeutische oder diagnostische Anwendung bei Menschen oder Tieren. Produkte und Inhalte sind durch Patente, Marken und Urheberrechte von
Für bestimmte Anwendungen sind möglicherweise zusätzliche geistige Eigentumsrechte Dritter erforderlich.

