Beschreibung
Hieff NGSTM EvoMax RNA Library Prep Kit (dUTP) ist ein vorgemischt, Actinomycin D kostenlos und strangspezifisch gesamt RNA-Sequenzierungsbibliothek Vorbereitung Kit kompatibel mit Illumina- und MGI-Plattformen. Dieses Produkt ist in zwei Ausführungen erhältlich: Rohr oder Dichtungssatz und Dieses Kit ist mehr komfortabel ob durch automatisierte Liquid Handling Device oder manuelle Manipulation . Das Kit enthält Reagenzien zur RNA-Fragmentierung, Reagenzien zur Reversen Transkription, Reagenzien zur strangspezifischen ds-cDNA-Synthese und Reagenzien zur Bibliotheksamplifikation. Es kann mit dem mRNA-Reinigungskit oder dem rRNA-Entfernungskit verknüpft werden, um mRNA- oder lncRNA-Forschung betreiben. Dieses Produkt optimiert das Reverse-Transkriptionsmodul, um eine Bibliothek mit hoher Kettenspezifität ohne Actinomycin D zu erhalten, was die Sicherheit der Experimentatoren in höherem Maße gewährleistet. Alle Reagenzien wurden einer strengen Qualitätskontrolle und Funktionsvalidierung unterzogen, um die Stabilität und Wiederholbarkeit der Bibliothek zu maximieren Vorbereitung.
Technische Daten
| Kat.-Nr. | 12340ES24/12340ES96/12340ES97/12340ES98 |
| Größe | 24 T / 96 T / 96 T (Automatik) / 96 T (Platte) |
Komponenten
| Komponenten Nr. | Name | 12340ES24 | 12340ES96 | 12340ES97 (Automatisierung) | 12340ES98 (Platte )** |
| 12340-A | Frag/Prime-Puffer | 450 μl | 2×900 μL | 2×1064 μL | 8×266 μL |
| 12340-B | 1. Reaktionsmodul 2.0 | 192 μl | 768 μl | 960 μl | 8×120 μL |
| 12340-C | 2. Reaktionsmodul (dUTP) | 840 μl | 3×1120 μL | 3×1280 μL | 8×480 μL |
| 12340-D | Ligationsreaktionsmodul | 840 μl | 3×1120 μL | 3×1280 μL | 8×480 μL |
| 12340-E | 2×Super Canace® II High-Fidelity-Mix | 600 μl | 2×1200 μL | 2×1360 μL | 8×340 μL |
| * | Grundierungsmischung* | / | / | / | / |
Hinweis: * Die Bezeichnung zeigt an, dass die Komponente nicht im Kit enthalten ist. Primer-Mix ist erforderlich, wenn die kompletten Adapter verwendet werden, andernfalls ist es nicht't. Dieses Kit ist mit Illumina- und MGI-Plattformen kompatibel, benötigt jedoch zusätzliche Primer-Mischung (Kat.-Nr. 13334 Primer-Mischung für MGITM und Kat.-Nr. 13335 Primer Mix für Illumina®) für die Illumina®- oder MGI-Plattform, wenn die kompletten Adapter verwendet werden.
Figur
Das Layout der Plattenreagenzgruppe.

Abbildung 1: Reagenzienlayout des Sealing-Plate-Bibliothekskits

Abbildung 2. Vereinfachte Komponenten von EvoMax RNA-Bibliothek-Vorbereitungskit.
Lagerung
Dieses Produkt sollte bei -25~-15℃ gelagert werden für 1 Jahre.
Anweisungen
1. Die empfohlene Zugabemenge in einem 20 μL-System beträgt 0,1 bis 1 Einheiten (U), und die Eingabemenge kann basierend auf den tatsächlichen Ergebnissen angepasst werden.
2. Je nach den Anforderungen des Experiments kann die Endkonzentration von dUTP zwischen 0,2 und 0,6 mM eingestellt werden, und 0,2 mM dTTP können selektiv hinzugefügt werden.
3. Die Reaktionszeit bei 25~37℃ kann je nach Versuchsbedarf innerhalb von 5 bis 10 Minuten angepasst werden.
Hinweise
1. Betrieb
1) Bitte operieren Sie mit Laborkittel und Einweghandschuhen,für Ihre Sicherheit.
2) Die Komponenten bei Zimmertemperatur auftauen. Durch mehrmaliges Auf- und Abdrehen gründlich mischen, kurz abzentrifugieren und zur Verwendung auf Eis legen.
3) Es wird empfohlen, jede Schrittreaktion in einem Thermocycler mit beheiztem Deckel durchzuführen. Der Thermocycler sollte vor der Verwendung auf die eingestellte Temperatur vorgeheizt werden.
4) Es ist notwendig, dass die Materialien frei von RNase-Verunreinigungen sind und der Versuchsbereich regelmäßig gereinigt wird. ThermoFishers RNAZapTM Zur Entfernung einer RNase-Kontamination wurde ein hochwirksames Nukleinsäureentfernungsspray empfohlen.
5) Unsachgemäße Handhabung kann sehr wahrscheinlich zu Aerosolverunreinigungen führen, die die Genauigkeit des Ergebnisses beeinträchtigen. Eine obligatorische physische Isolierung der PCR-Reaktionsmischbereiche und der PCR-Produktreinigungstestbereiche wird empfohlen. Ausgestattet mit Geräten wie Spezialpipetten für den Bibliotheksaufbau.
6) Dieses Reagenz ist für den einmaligen Gebrauch bestimmt. Eine Mehrfachverwendung ist strengstens untersagt.
7) Dieses Produkt ist nur für Forschungszwecke bestimmt.
2. Adapter-Ligation
1) Kunden können je nach ihren experimentellen Anforderungen zwischen langen Adapter-Kits (Adapter mit Strichcode) und kurzen Adapter-Kits von Illumina oder MGI wählen.
2) Es wird empfohlen, hochwertige, kommerzielle Adapter auszuwählen. Wenn Sie sich für selbst hergestellte Adapter entscheiden, vertrauen Sie diese bitte einem Unternehmen mit Erfahrung in der NGS-Primersynthese an und weisen Sie auf die Notwendigkeit einer strengen Kontaminationskontrolle hin. Darüber hinaus wird empfohlen, die DNA-Annealing-Lösung in einer Reinraumbank vorzubereiten und jedes Mal nur einen Adaptertyp zu verwenden, um Kreuzkontaminationen zu vermeiden.
3) Tauen Sie die Adapter bitte auf Eis oder bei 4°C auf, bei Betrieb bei Raumtemperatur sollte die Labortemperatur 25°C nicht überschreiten, um eine Denaturierung der Adapter zu vermeiden.
4) Die Konzentration des Adapters wirkt sich direkt auf die Ligationseffizienz und die Bibliotheksausbeute aus. Das dem Kit hinzugefügte Adaptervolumen ist auf 5 μl festgelegt. Es wird empfohlen, die Adapter mit 0,1 × TE-Puffer zu verdünnen. Die verdünnten Adapter können 48 Stunden lang bei 4 °C gelagert werden. Tabelle 1 listet die empfohlene Adaptermenge für unterschiedliche Mengen an Eingangs-RNA auf.
Tabelle 1-1 Die empfohlene Illumina-Adaptermenge für verschiedene Eingangs-RNA
| Eingabe Gesamt-RNA | Illumina® Adapter Stammkonzentration |
| 10 ng | 1 μM |
| 100 ng | 1,5 μM |
| 500 ng | 3 μM |
| ≥1 μg | 5 μM |
Tabelle 1-2 Die empfohlene MGI-Adaptermenge für verschiedene Eingangs-RNA
| Eingabe Gesamt-RNA | MGI® Adapter Stammkonzentration |
| 10~99 ng | 1 μM |
| 100~499 ng | 2 μM |
| 500~4000 ng | 5 μM |
* Die Adapternutzung kann je nach Art der Gesamt-RNA-Proben und Eingangsmenge angepasst werden
3.Bibliothekserweiterung
1) Auf der Grundlage der DNA-Polymerase der ersten Generation weist die hochpräzise DNA-Polymerase im Kit eine stark verbesserte Amplifikationsgleichmäßigkeit auf und weist keinen Amplifikationsbias auf.
2) Die Anzahl der Amplifikationszyklen muss streng kontrolliert werden. Unzureichende Amplifikation kann zu einer geringen Bibliotheksausbeute führen; Überamplifikation kann zu erhöhter Verzerrung, Fehlern, doppeltem Lesen, chimären Produkten und der Ansammlung von Expansionsmutationen führen. Tabelle 2 listet die empfohlenen Zyklenzahlen für die PCR-Amplifikation auf.
3) Die in Tabelle 2 empfohlene Anzahl an Zyklen kann die überwiegende Mehrheit der Anforderungen an die Bibliotheksvorbereitung erfüllen. Wenn Ihre Probe von schlechter Qualität ist (z. B. FFPE-Proben mit starker Degradation), kann die Anzahl der Zyklen je nach tatsächlicher Situation angemessen erhöht werden. Beachten Sie, dass mRNA je nach Art und Gewebe unterschiedlich ist. Die Anzahl der Amplifikationszyklen sollte angepasst werden.
Tabelle 2 Eingabe Gesamt-RNA-Volumen und empfohlene Amplifikationszyklen Tabelle *
| Eingabe Gesamt-RNA | Anzahl der Zyklen |
| 10 ng | 16 |
| 100 ng | 14 |
| 500 ng | 12 |
| 1 μg | 11 |
[Hinweis]: *Die Ausbeute der Bibliothek hängt nicht nur von der Eingangsmenge und der Anzahl der Amplifikationszyklen ab, sondern wird auch von der Qualität der Proben, den Fragmentierungsbedingungen und den Sortierbedingungen beeinflusst. Wählen Sie beim Aufbau der Bibliothek die am besten geeigneten Bedingungen entsprechend der tatsächlichen Situation aus.
4. Bead-basierte DNA-Reinigung und Größenauswahl
1) Es gibt mehrere Schritte im Bibliotheksaufbauprozess, die magnetische Perlen zur DNA-Reinigung erfordern. Wir empfehlen Hieff NGS™ DNA Selection Beads (Yeasen Cat#12601) oder AMPure® XP magnetische Perlen (Beckman Cat#A63880) zur DNA-Reinigung und Größenauswahl.
2) Die magnetische Perle sollte vor dem Gebrauch auf Raumtemperatur gebracht werden, sonst Die Ausbeute verringert sich und der Größenauswahleffekt wird beeinträchtigt.
3) Die magnetischen Perlen sollten vor der Verwendung durch Vortexen oder Pipettieren gut gemischt werden.
4) Saugen Sie die Perlen beim Übertragen des Überstands nicht ab, selbst kleinste Mengen der Perlen können die folgenden Reaktionen beeinträchtigen.
5) Der 80-prozentige Ethanol muss frisch zubereitet werden, da sonst die Rückgewinnungseffizienz beeinträchtigt wird.
6) Die Magnetkügelchen sollten vor dem Eluieren des Produkts bei Raumtemperatur getrocknet werden. Bei unzureichender Trockenheit können Ethanolrückstände leicht nachfolgende Reaktionen beeinträchtigen; bei übermäßiger Trockenheit reißen die Magnetkügelchen und die Reinigungsausbeute wird reduziert. Normalerweise reicht es aus, die Kügelchen 3–5 Minuten bei Raumtemperatur trocknen zu lassen, damit sie vollständig trocknen.
7) Bei Bedarf können die gereinigten oder nach Größe selektierten und im TE-Puffer eluierten DNA-Proben 1–2 Wochen bei 4 °C oder einen Monat bei -20 °C gelagert werden.
5.Analyse der Bibliotheksqualität
Im Allgemeinen kann die Qualität der erstellten Bibliothek durch Konzentrationserkennung und Längenverteilungserkennung bewertet werden.
6. Sonstiges Material
1) mRNA-Anreicherungskit: Hieff NGS® mRNA Isolation Master Kit V2 (Yeasen Cat # 12629).
2) rRNA-Entfernungskit: Hieff NGS® MaxUp Human rRNA Depletion Kit (rRNA & ITS / ETS) (Yeasen Cat # 12257) oder anderes rRNA-Entfernungskit.
3) RNA-Reinigung von magnetischen Perlen: Hieff NGS® RNA Cleaner (Yeasen Cat # 12602) oder andere gleichwertige Produkte.
4) DNA-gereinigte magnetische Perlen: Hieff NGS® DNA Selection Beads (Yeasen Cat # 12601) oder AMPure® XP Beads (A63880) oder andere gleichwertige Produkte.
5) RNA-Qualitätskontrolle: Agilent 2100 Bioanalyzer RNA 6000 Nano/Pico Chip oder andere gleichwertige Produkte.
6) Adapter: Komplettadapter für Illumina®-Verwendung (Kat.-Nr. 13519-13520 oder gleichwertig) oder Komplettadapter für MGI®-Verwendung (Kat.-Nr. 13360-13362 oder gleichwertig).
7. Qualitätskontrolle der Bibliothek
Agilent 2100 Bioanalyzer DNA 1000 Chip / High Sensitivity Chip oder andere gleichwertige Produkte; Reagenzien zur Bibliotheksquantifizierung.
8. Sonstige Materialien
Wasserfreier Ethanol, steriles Reinstwasser, Pistolenkopf mit geringer Adsorption, PCR-Röhrchen, Magnetrahmen, PCR-Instrument usw.
Flussdiagramm

Abbildung 1: Prozess des Aufbaus einer RNA-Bibliothek
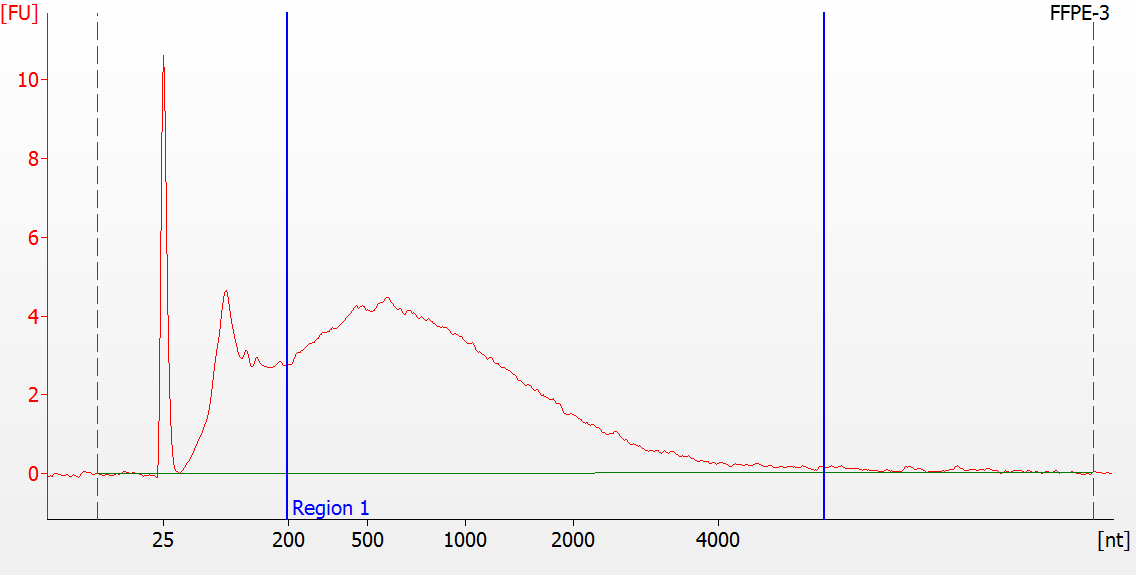
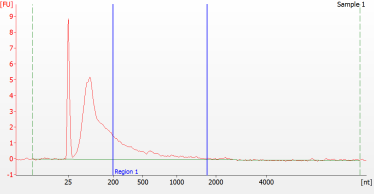
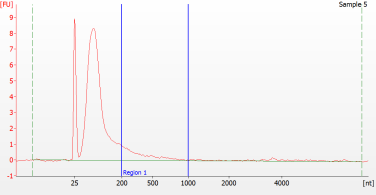
RNA-Bibliotheksvorbereitung für FFPE-Proben unterschiedlicher Qualität
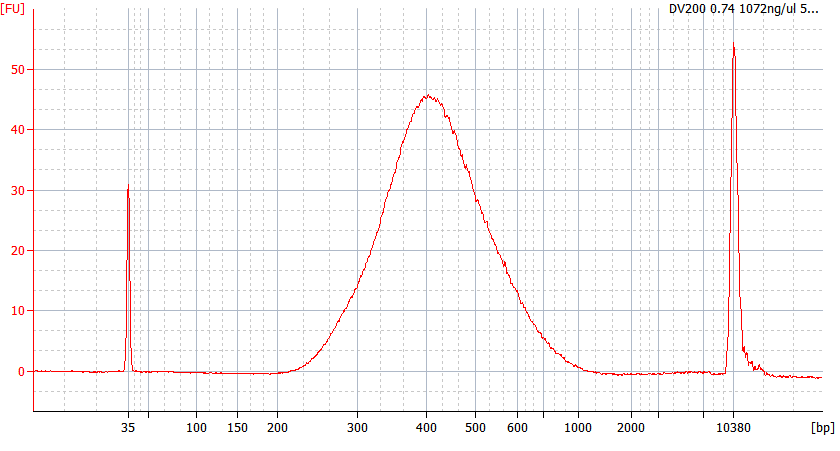
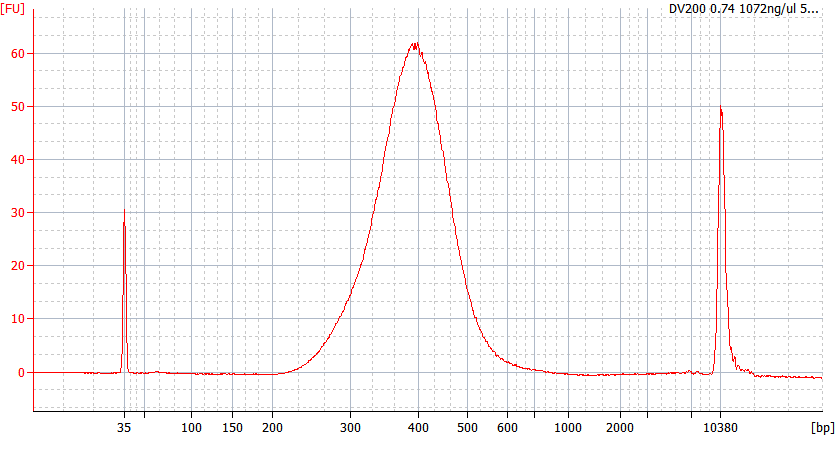
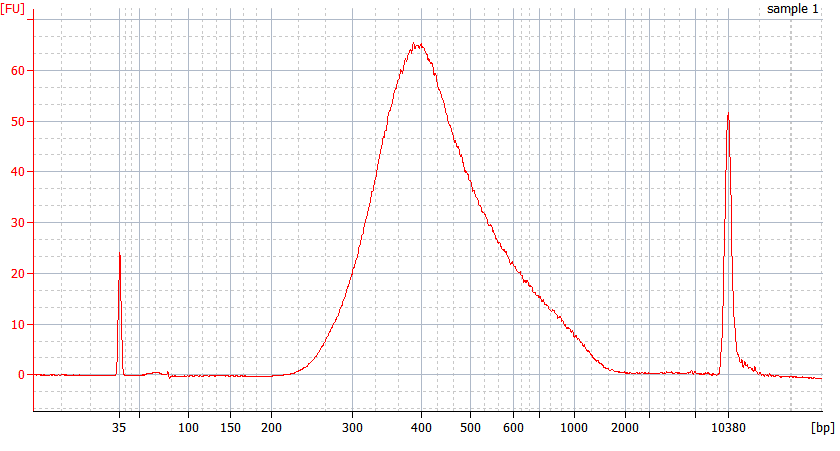
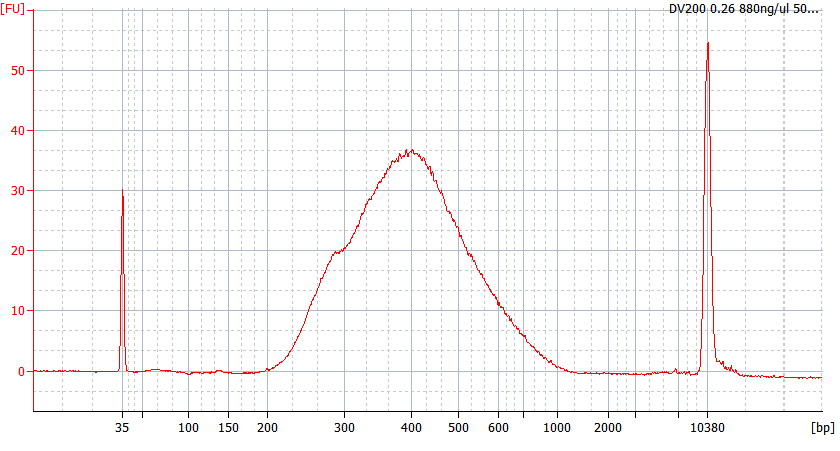
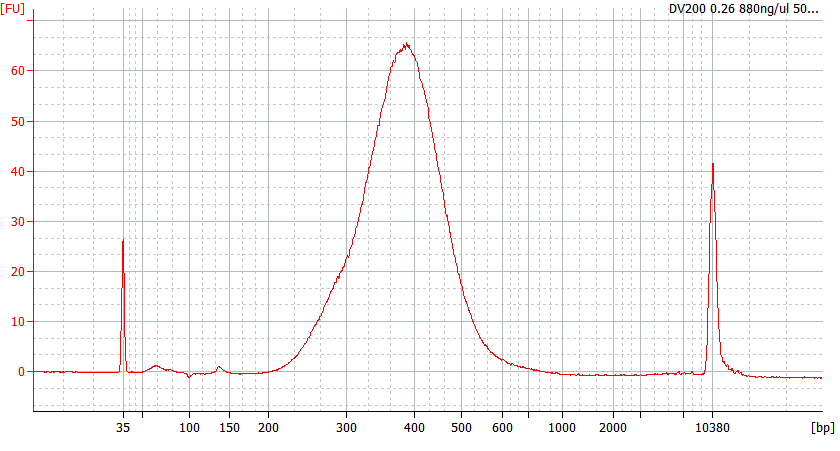
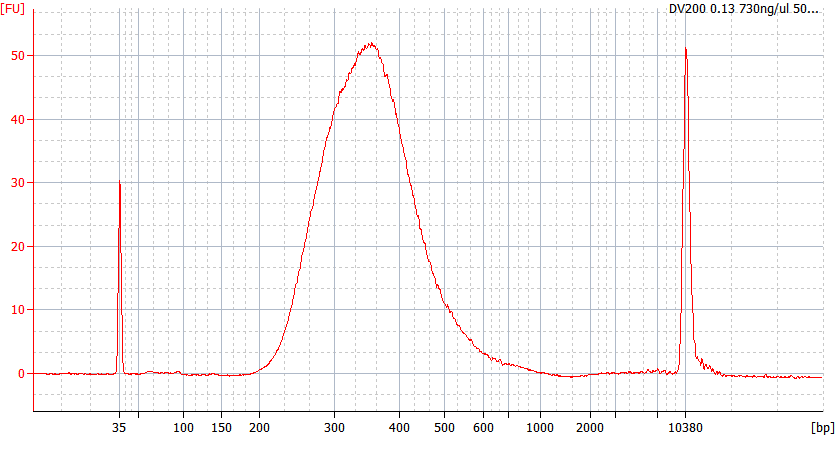
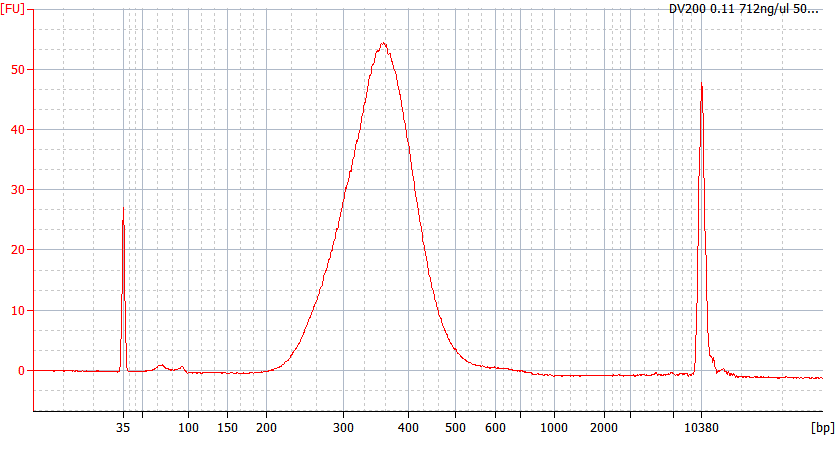
Für stark degradierte FFPE-RNA (DV 200 < 50 %) und Proben mit geringer Eingangsmenge empfehlen wir nach der Adapterligation ein Doppelreinigungsprotokoll, um den Bibliotheksverlust zu reduzieren.
Spitzenmuster von FFPE-Proben unterschiedlicher Qualität
Zahlung und Sicherheit
Ihre Zahlungsinformationen werden sicher verarbeitet. Wir speichern weder Kreditkartendaten noch Zugriff auf Ihre Kreditkarteninformationen.
Anfrage
Sie können auch mögen
FAQ
Das Produkt ist nur für Forschungszwecke bestimmt und nicht für die therapeutische oder diagnostische Anwendung bei Menschen oder Tieren. Produkte und Inhalte sind durch Patente, Marken und Urheberrechte von
Für bestimmte Anwendungen sind möglicherweise zusätzliche geistige Eigentumsrechte Dritter erforderlich.