Beschreibung
Das Hieff NGS™ OnePot Pro DNA Library Prep Kit V4 ist ein enzymatisches Bibliotheksvorbereitungskit der nächsten Generation, das für die Verwendung mit Illumina entwickelt wurde und MGI-Hochdurchsatz-Sequenzierungsplattformen. Im Vergleich zu herkömmlichen Methoden zur Bibliotheksvorbereitung verwendet dieses Kit hochwertige Fragmentierungsenzyme, wodurch aufwändige Ultraschallprozesse überflüssig werden. Die Fragmentierungs- und Endreparaturmodule werden in einem einzigen Schritt kombiniert, wodurch sowohl der Zeit- als auch der Kostenaufwand für die Bibliotheksvorbereitung erheblich reduziert werden. Dieses Kit eignet sich für eine Vielzahl von Probentypen, darunter Pflanzen- und Tiergenome, mikrobielle Genome und mehr, mit einem Probeneingabebereich von 1 ng bis 1 μg. Die enzymatische Fragmentierung gewährleistet einheitliche Fragmentgrößen über verschiedene Arten hinweg mit minimalen Abweichungen zwischen den Arten. Darüber hinaus kann dieses Kit mit Illumina kombiniert werden oder MGI Adapter und Primer für die Sequenzierung auf Illumina- oder MGI-Hochdurchsatzplattformen.
Technische Daten
| Kat.-Nr. | 12972ES08 / 12972ES24 / 12972ES96 |
| Größe | 8 T / 24 T / 96 T |
Komponenten
| Komponenten Nr. | Name | 12972ES08 | 12972ES24 | 12972ES96 |
| 12972-A | 80 μL | 240 μL | 960 μL | |
| 12972-B | Smearase™ Enzym 4.0 | 80 μL | 240 μL | 960 μL |
| 12972-C | Ligationsverstärker 4.0 | 240 μl | 720 μL | 3×960 μL |
| 12972-D | Schnell DNA-Ligase 4.0 | 80 μL | 240 μL | 2×480 μL |
| 12972-E | 2×Ultima HF-Verstärkungsmix | 200 μl | 600 μL | 3×800 μL |
Lagerung
Dieses Produkt sollte bei -25~-15℃ für 1 gelagert werden Jahr.
Hinweise
1. Über die Operation
1. Bitte operieren Sie mit Laborkittel und Einweghandschuhen,zu Ihrer Sicherheit.
2. Komponenten bei Raumtemperatur auftauen. Nach dem Auftauen, durch Vortexen gründlich mischen, das Röhrchen kurz zentrifugieren und zur späteren Verwendung auf Eis legen.
3. Beim Vorbereiten der Reaktionslösung für jeden Schritt wird empfohlen, sie mit einer Pipette gut zu vermischen oder vorsichtig zu schütteln. Kräftiges Schütteln kann zu einer Verringerung der Bibliotheksausgabe führen.
4. Es wird dringend empfohlen, gefilterte Pipettenspitzen zu verwenden, um Kreuzkontaminationen zu vermeiden. Achten Sie darauf, die Pipettenspitzen zu wechseln, wenn Sie verschiedene Proben verarbeiten.
5. Unsachgemäße Handhabung kann sehr wahrscheinlich zu Aerosolverunreinigungen führen, die die Genauigkeit des Ergebnisses beeinträchtigen. Eine obligatorische physische Isolierung der PCR-Reaktionsmischbereiche und der PCR-Produktreinigungstestbereiche wird empfohlen. Ausgestattet mit Geräten wie Spezialpipetten für den Bibliotheksaufbau.Führen Sie eine Routinereinigung für jeden Bereich durch, indem Sie die Oberflächen mit 0,5 % Natriumhypochlorit oder 10 % Bleichmittel abwischen.
6. Dieses Produkt ist nur für Forschungszwecke bestimmt.
2. DNA-Fragmentierung1. Das Kit ist kompatibel mit 100 pg - 1000 ng Eingangs-DNA. Es wird dringend empfohlen, qualitativ hochwertige Eingangs-DNA mit A260/A280 = 1,8-2,0 zu verwenden.
2. Nachfolgende Experimente könnten beeinträchtigt werden, wenn hohe Konzentrationen von Salzen wie dem Metallchelatbildner mit der Eingangs-DNA eingeführt wurden. Wir empfehlen, die DNA-Probe in ddH2O zur Fragmentierung.
3. Bitte beachten Sie die Tabelle 6 für die Fragmentierungszeit von Standard-DNA-Proben.Das Kit weist eine geringe Fragmentierungsneigung auf und bietet eine gleichmäßige GC-Abdeckung für DNA-Proben mit einem breiten Spektrum an GC-Zusammensetzungen. Bitte passen Sie die Fragmentierungszeit entsprechend Ihren experimentellen Anforderungen an.
4. Für eine genaue Fragmentierung bereiten Sie die Reaktion bitte auf Eis vor.
3. Adapterligatur
1. Kunden können je nach ihren experimentellen Anforderungen zwischen langen Adapter-Kits (Adapter mit Strichcode) und kurzen Adapter-Kits von Illumina oder MGI wählen.
2. Es wird empfohlen, hochwertige, kommerzielle Adapter auszuwählen. Wenn Sie sich für selbst hergestellte Adapter entscheiden, vertrauen Sie diese bitte einem Unternehmen mit Erfahrung in der NGS-Primersynthese an und weisen Sie auf die Notwendigkeit einer strengen Kontaminationskontrolle hin. Darüber hinaus wird empfohlen, die DNA-Annealing-Lösung in einer Reinraumbank vorzubereiten und jedes Mal nur einen Adaptertyp zu verwenden, um Kreuzkontaminationen zu vermeiden.
3. Tauen Sie die Adapter bitte auf Eis oder bei 4°C auf, bei Betrieb bei Raumtemperatur sollte die Labortemperatur 25°C nicht überschreiten, um eine Denaturierung der Adapter zu vermeiden.
4. Die Qualität und Konzentration der Adapter wirken sich direkt auf die Ligationseffizienz und die Bibliotheksausbeute aus. Eine zu hohe Adapterkonzentration begünstigt die Bildung von Adapterdimeren, während eine zu geringe Adapterkonzentration die Ligationsrate und die Bibliotheksausbeute verringert. Entsprechende Verdünnungen mit TE-Puffer je nach DNA-Eingangsmenge bei Verwendung von Adaptern.Tabelle 2 -3 listet die empfohlenen Adapter-Verdünnungsmethoden für unterschiedliche Input-DNA-Mengen bei Verwendung dieses Kits auf.
Tabelle 1 Die empfohlene Illumina-Adaptermenge für verschiedene Eingangs-DNA
| DNA-Eingabe | Adapterverdünnung (Adaptervolumen: Gesamtvolumen) | Konzentration |
| 1 ng | 30-Falten (1: 30) | 0,5 μM |
| 10 ng | 7.5-Falten (1: 7.5) | 2 μM |
| 100 ng | 3-Falten (1: 3) | 5 μM |
| 1000 ng | 1.5-Falten (1: 1.5) | 10 μM |
Tabelle 2 Die empfohlene MGI-Adaptermenge für verschiedene Eingangs-DNA
| DNA-Eingabe | Adapterverdünnung (Adaptervolumen: Gesamtvolumen) | Konzentration |
| 1 ng | 20-Falten (1: 20) | 0,5 μM |
| 10 ng | 5-Falten (1: 5) | 2 μM |
| 100 ng | 2-Falten (1: 2) | 5 μM |
| 1000 ng | unverdünnt | 10 μM |
4. DNA-Reinigung und Größenauswahl auf Perlenbasis
1. Der Schritt zur Auswahl der DNA-Fragmentgröße kann entweder nach der Adapterligation oder nach der Bibliotheksamplifikation durchgeführt werden.
2. Es wird empfohlen, die Größenauswahl direkt nach der Adapterligation durchzuführen, wenn die eingegebene DNA-Menge mehr als 50 ng beträgt; Andernfalls führen Sie die Größenauswahl bitte nach der Verstärkung durch.
3. Der Ligation Enhancer enthält eine hohe Konzentration an PEG, was die genaue Größenauswahl erheblich beeinträchtigen kann. Wenn die Größenauswahl direkt nach der Adapterligatur durchgeführt werden soll, wird daher dringend empfohlen, vor der Größenauswahl einen Perlenreinigungsschritt hinzuzufügen. Der Schritt zur Größenauswahl kann direkt durchgeführt werden, wenn er vor der Endreparatur/dA-Tailing oder nach die Bibliotheksverstärkung.
4. Die magnetischen Perlen sollten vor der Verwendung auf Raumtemperatur gebracht werden, da sich sonst die Ausbeute verringert und der Größenauswahleffekt beeinträchtigt wird.
5. Die magnetischen Perlen sollten vor der Verwendung durch Vortexen oder Pipettieren gut gemischt werden.
6. Saugen Sie die Perlen beim Übertragen des Überstands nicht ab, selbst kleinste Mengen der Perlen können die folgenden Reaktionen beeinträchtigen.
7. Der 80-prozentige Ethanol muss frisch zubereitet werden, da sonst die Rückgewinnungseffizienz beeinträchtigt wird.
8. Für eine genaue Größenauswahl wird empfohlen, mit einem Volumen von mehr als 100 μL zu beginnen. Wenn das Volumen geringer ist, wird empfohlen, das Volumen mit ultrareinem Wasser auf 100 μL zu bringen.
9. Die Magnetkügelchen sollten vor dem Eluieren des Produkts bei Raumtemperatur getrocknet werden. Bei unzureichender Trockenheit können Ethanolrückstände leicht nachfolgende Reaktionen beeinträchtigen; bei übermäßiger Trockenheit reißen die Magnetkügelchen und die Reinigungsausbeute wird reduziert. Normalerweise reicht es aus, die Kügelchen 3–5 Minuten bei Raumtemperatur trocknen zu lassen, damit sie vollständig trocknen.
10. Bei Bedarf werden die gereinigten oder nach Größe selektierten DNA-Proben in 0,1× TE-Puffer kann 1–2 Wochen bei 4 °C oder einen Monat bei -20 °C gelagert werden.
5. Bibliothekserweiterung
1. Ob eine Bibliotheksamplifikation durchgeführt werden soll oder nicht, hängt von der Menge der eingegebenen DNA, den Adaptertypen, den Sequenzierungsdatenanwendungen usw. ab. Der Amplifikationsschritt ist erforderlich, wenn Teiladapter verwendet werden. Bei Verwendung von Adaptern voller Länge wird eine Amplifikation empfohlen, wenn die eingegebene DNA <200 ng ist; andernfalls ist eine Amplifikation nicht erforderlich.
2. Die Anzahl der Amplifikationszyklen sollte streng kontrolliert werden. Unzureichende Amplifikation kann zu einer geringen Bibliotheksausbeute führen; Überamplifikation kann zu erhöhter Verzerrung, Fehlern, doppeltem Lesen und chimären Produkten führen. Tabelle 3 listet empfohlene Zykluszahlen auf, die auf eine Bibliotheksausbeute von 1 μg abzielen.
Tabelle 3 Die empfohlene Anzahl von Zyklen zur Erzeugung 1.000 ng Bibliotheksausbeute
| DNA-Eingabe | Anzahl der Zyklen, die zur Erzeugung von 1 μg Bibliotheksausbeute erforderlich sind |
| 1000 ng | 2 - 4 |
| 500 ng | 2 - 4 |
| 250 ng | 4 - 6 |
| 100 ng | 5 - 7 |
| 50 ng | 7 - 9 |
| 1 ng | 12 - 14 |
Notiz:
1.Tabelle 3 zeigt die Anzahl der Loop-Parameter bei Verwendung hochwertiger Input-DNA-Tests von etwa 200 bp. Die FFPE-DNA-Qualität variiert stark, und bei schlechter DNA-Qualität oder großer Bibliothekslänge muss die Anzahl der Zyklen entsprechend erhöht werden, um ausreichend Bibliotheken zu erhalten.
2.ICHWenn beim Erstellen der Bibliothek eine Größenauswahl erforderlich ist, wird eine höhere Zyklenzahl für die Bibliotheksamplifikation empfohlen; andernfalls wird eine niedrigere Zyklenzahl empfohlen.
3.ICHBei Verwendung unvollständiger Adapter müssen mindestens 2 Zyklen verstärkt werden, um einen vollständigen Adapter zu bilden.
6. Analyse der Bibliotheksqualität
1. Die Qualität der erstellten Bibliotheken wird im Allgemeinen durch Messen der Konzentrationen und Größenverteilungen analysiert.
2. Die Konzentrationen der Bibliotheken können mit fluoreszenzbasierten Methoden wie Qubit und PicoGreen oder qPCR gemessen werden.
3. Es wird NICHT empfohlen, absorptionsbasierte Quantifizierungsmethoden wie NanoDrop zu verwenden.
4. Es wird empfohlen, die qPCR-Methode zur Quantifizierung der Bibliothek zu verwenden: Fluoreszenzbasierte Methoden wie Qubit und PicoGreen können die unvollständigen dsDNA-Strukturen (Inserts ohne Adapter oder mit nur einem der mit Adapter ligierten Enden) nicht von den vollständigen Bibliotheken unterscheiden. Die qPCR-Methode amplifiziert und misst nur die vollständigen Bibliotheken, deren beide Enden mit Adaptern ligiert sind (die sequenzierbaren Bibliotheken) und bietet somit eine genauere Messung zum Laden.
5. Die Größenverteilung von Bibliotheken kann mit Agilent Bioanalyzer oder anderen Geräten analysiert werden, die auf den Prinzipien der Kapillarelektrophorese oder Mikrofluidik basieren.
7. Andere Materialien
1. Magnetkügelchen zur DNA-Reinigung: Hieff NGSTM DNA-Selektionsperlen (Yeasen Cat#12601) oder AMPure® XP Beads (A63880) oder andere gleichwertige Produkte.
2. Adapter: Komplettadapter für Illumina: Yeasen Kat.-Nr.: 13519-13520; 384 Dual-CDI-Zündhütchen: Ja Katze Nr. 12412 ~ Katze Nr. 12413; 384 Unique Dual Index (UDI)-Primer: Yeasen Katze#12327~Katze Nr. 13330; UMI UDI-Adapter: Ja Katze Nr. 13370 ~ Katze Nr. 13371; Kompletter Adapter für MGI: Yeasen Cat#13360-13362. DNA Primer Mix:Katze#12190 oder Katze#12191.
3. Qualitätsanalyse der Bibliothek: Agilent 2100 Bioanalyzer DNA 1000 Chip/High Sensitivity Chip oder andere gleichwertige Produkte; quantitative Reagenzien für die Bibliothek.
4. Andere Materialien: absoluter Ethanol, steriles Reinstwasser, Pipettenspitzen mit geringer Retention, PCR-Röhrchen, Magnetständer, Thermocycler usw.
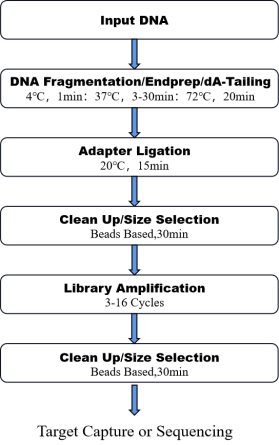
8. Workflow
Abbildung 1.Der Workflow von Ein Topf Pro DNA Bibliotheks-Vorbereitungskit
Zahlung und Sicherheit
Ihre Zahlungsinformationen werden sicher verarbeitet. Wir speichern weder Kreditkartendaten noch Zugriff auf Ihre Kreditkarteninformationen.
Anfrage
Sie können auch mögen
FAQ
Das Produkt ist nur für Forschungszwecke bestimmt und nicht für die therapeutische oder diagnostische Anwendung bei Menschen oder Tieren. Produkte und Inhalte sind durch Patente, Marken und Urheberrechte von
Für bestimmte Anwendungen sind möglicherweise zusätzliche geistige Eigentumsrechte Dritter erforderlich.