Beschreibung
Ribonuklease R (RNase R) aus Escherichia coliist eine magnesiumabhängige 3′→5′ Exoribonuklease, die dürfen verdaut alle linearen RNAs, aber keine Lariat- oder zirkulären RNA-Strukturen. Die meisten zellulären RNAs werden vollständig von RNase R verdaut, mit Ausnahme von tRNAs, 5S-RNA und Intron-Lariats. RNase R wird zur Anreicherung von circRNAs in biologischen Proben, zur Identifizierung von intronischen Lariat-Sequenzen, zur Identifizierung von exonischen circRNAs und zum Studium des alternativen Spleißens verwendet.
Merkmale
Garantierte Wirksamkeit und Qualität durch ISO 13485-Zertifizierung.
Erhältlich in Großpackungen oder maßgeschneiderten Konfigurationen.
Baut lineare RNA effektiv ab, während Lariat-Loops und zirkuläre RNAs erhalten bleiben.
Die selektive Verdauung linearer RNAs verbessert die Isolierung zirkulärer RNAs für die Proteinproduktion oder den Aufbau intronenbasierter cDNA-Bibliotheken.
Angebot an RNAse R in GMP-Qualität
Spezifikationen
| Quelle | Ein rekombinanter Escherichia coli Stamm, der das RNAse R-Gen trägt Escherichia coli |
| Optimale Temperatur | 37℃ |
| Speicherpuffer | 50 mM Tris-HCl, 100 mM NaCl, 0,1 mM EDTA, 0,1 % Triton® X-100, 1 mM DTT, 50% Glycerin, pH 7,5 bei 25℃ |
| Einheitendefinition | Eine Einheit wandelt 1 μg Poly-r(A) in säurelöslichen Nukleotiden in 10 Minuten bei 37°C in 20 mM Tris-HCl (pH 8,0), 100 mM KCl und 0,1 mM MgCl um.2. |
Anwendung
RNase R dient als grundlegende Komponente bei der zirkulären RNA-Synthese (circRNA). CircRNA verspricht eine Verbesserung der pharmazeutischen Stabilität und Biostabilität. Mit ihren kovalent geschlossenen Strukturen widerstehen circRNAs RNA-Exonukleasen. [1,2] RNase R findet breite Anwendung in verschiedenen Studien, darunter alternatives Spleißen, Intron-cDNA-Produktion und die Isolierung von Spleißintermediaten und Lariats.
Komponenten
| Komponenten Nr. | Name | 14615ES25 | 14615ES72 | 14606ES80 |
| 14615 | RNase R (GMP-Qualität, 20 U/μL) | 25 μl | 250 μl | 1 ml |
Lagerung
Dieses Produkt sollte 2 Jahre lang bei -25–15 °C gelagert werden.
Anweisungen
Bereiten Sie das folgende Reaktionssystem in einem sterilen Mikrozentrifugenröhrchen vor
| Komponente | Volumen |
| 10×RNase R-Reaktionspuffer | 2 μL |
| RNA-Probe | 1 μg |
| RNase R (20 U/μL) | 2-4 HE |
| RNase-freies ddH2O | Bis zu 20 μL |
Inkubieren bei 37 ℃ für 10 - 30 Minuten.
Zahlen
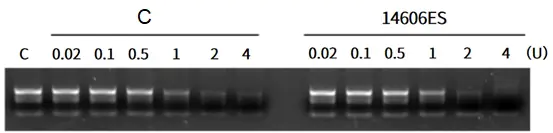
Abbildung 1: Lineare RNA-Moleküle können durch Yeasen RNAse R verdaut werden.
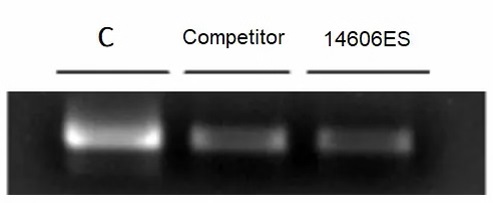
Abbildung 2: CircRNA kann nicht durch Yeasen RNAse R verdaut werden.
Hinweise
- Die Aktivität der RNase R erfordert 0,1-1,0 mM Mg2+;
- Mit der Erhöhung der RNA kann die Reaktionszeit verlängert und die Enzymmenge entsprechend erhöht werden;
- Der EDTA-Gehalt in RNA-Proben kann die Aktivität von RNase R beeinflussen.
- Eine Inkubation bei 70 °C für 10 Minuten kann das Enzym inaktivieren.
Referenz
- Qu L, Yi Z, Shen Y, et al. Zirkuläre RNA-Impfstoffe gegen SARS-CoV-2 und neue Varianten. Cell. 2022;185(10):1728-1744.e16. doi:10.1016/j.cell.2022.03.044
- Chen L, Wang C, Sun H et al. Die bioinformatische Toolbox zur Entdeckung und Analyse von circRNA. Brief Bioinform. 2021;22(2):1706-1728. doi:10.1093/bib/bbaa001
Zahlung und Sicherheit
Ihre Zahlungsinformationen werden sicher verarbeitet. Wir speichern weder Kreditkartendaten noch Zugriff auf Ihre Kreditkarteninformationen.
Anfrage
Sie können auch mögen
FAQ
Das Produkt ist nur für Forschungszwecke bestimmt und nicht für die therapeutische oder diagnostische Anwendung bei Menschen oder Tieren. Produkte und Inhalte sind durch Patente, Marken und Urheberrechte von
Für bestimmte Anwendungen sind möglicherweise zusätzliche geistige Eigentumsrechte Dritter erforderlich.

