Beschreibung
Hieff NGSTM UCF.ME DNA-Selektionskügelchen werden nach dem SPRI-Prinzip (Solid Phase Reverse Immobilization) hergestellt. und ist für die DNA-Reinigung und Größenauswahl bei der Vorbereitung von Next Generation Sequencing (NGS)-Bibliotheken anwendbar. Hieff NGSTM UCF.ME DNA Selection Beads sind mit verschiedenen DNA- und RNA-Bibliotheksvorbereitungskits kompatibel. Dieses Produkt wird in einer ultrareinen Anlage mit einem hohen Sauberkeitsgrad hergestellt und kann für den DNA-Reinigungsschritt bei Krankheitserregererkennungsprozessen verwendet werden.
Technische Daten
| Kat.NO. | 17266ES03 /17266ES75 |
| Größe | 1 ml /450 ml |
Komponenten
| Komponenten Nr. | Name | 17266ES03 | 17266ES75 |
| 17266 | Hieff NGS UCF.ME DNA-Selektionsperlen | 1 ml | 450 ml |
Lagerung
Dieses Produkt sollte gelagert werden bei 2~8℃ für 18 Monate.
Versand und Lagerung
Die Perlen werden mit Kühlakkus versendet und können ein Jahr lang bei 2°C–8°C gelagert werden.
Anweisungen
- 1. Vorbereitung
Äquilibrieren Sie die Selektionskügelchen vor der Verwendung mindestens 30 Minuten lang bei Raumtemperatur.
- 2. Auswahl der DNA-Größe
Der Vorgangsablauf zur Größenauswahl ist in Abbildung 1 dargestellt und das Protokoll lautet wie folgt.
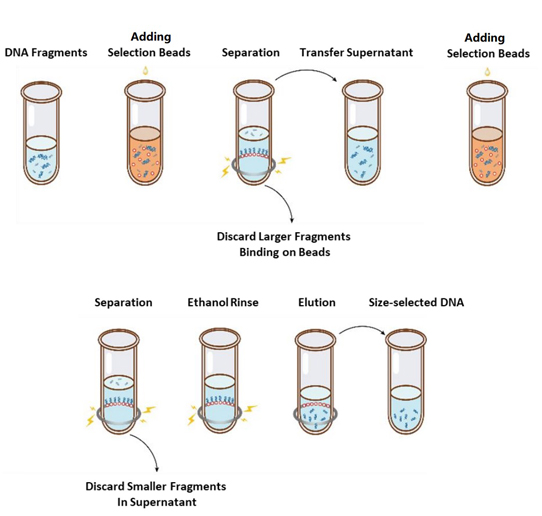
Abbildung 1. Das Flussdiagramm der DNA-Größenauswahl
2.1 Mischen Sie die Perlen vor jedem Gebrauch gründlich durch Vortexen oder Auf- und Abpipettieren.
2.2 Fügen Sie der Probe die erste Runde Selektionsperlen hinzu (siehe Tabelle 1). Mischen Sie gründlich, indem Sie die Probe mindestens 10 Mal auf dem Vortex-Rührer oder durch Auf- und Abpipettieren auf- und abpumpen.
2.3 5 Minuten bei Raumtemperatur inkubieren.
2.4 Zentrifugieren Sie das Röhrchen kurz und stellen Sie es auf einen Magnetständer. Wenn die Lösung klar ist (ca. 5 min), geben Sie den Überstand in ein neues PCR-Röhrchen.
2.5 Fügen Sie der Probe aus Schritt 2.4 gemäß Tabelle 1 die zweite Runde Selektionskügelchen hinzu. Mischen Sie gründlich, indem Sie mindestens 10 Mal auf und ab verwirbeln oder pipettieren.
2.6 5 Minuten bei Raumtemperatur inkubieren.
2.7 Zentrifugieren Sie das Röhrchen kurz und stellen Sie es auf einen Magnetständer. Wenn die Lösung klar ist (ca. 5 min), saugen Sie den Überstand ab und entsorgen Sie ihn.
2.8 Belassen Sie das Röhrchen im Magnetständer und geben Sie 200 μl frisch zubereiteten 80%igen Ethanol hinzu, ohne die Perlen zu beschädigen. Inkubieren Sie es 30 Sekunden lang bei Raumtemperatur. Saugen Sie das Ethanol ab und entsorgen Sie es.
2.9 Wiederholen Sie Schritt 2.8 einmal, also insgesamt zwei Mal.
2.10 Restliches Ethanol mit 10-µL-Pipettenspitzen entfernen. Das Röhrchen im Magnetständer lassen und die Selektionsperlen bei geöffnetem Deckel lufttrocknen, bis Risse sichtbar werden (ca. 5 Min.).
Hinweis: Trocknen Sie die Selektionsperlen nicht zu stark. Dies kann zu einer geringeren DNA-Zielausbeute führen.
2.11 Entfernen Sie das Röhrchen vom Magnetständer. Fügen Sie eine entsprechende Menge ddH2O (≥20 µL) hinzu und mischen Sie gründlich durch Vortexen oder mindestens 10-maliges Auf- und Abpipettieren.
2.12 5 Minuten bei Raumtemperatur inkubieren.
Zentrifugieren Sie das Röhrchen kurz und stellen Sie es auf den Magnetständer. Wenn die Lösung klar ist (ca. 5 Minuten), geben Sie 20 μL des Überstands in ein neues Röhrchen.
- 3. Empfohlene Bedingungen für die Auswahl der DNA-Größe
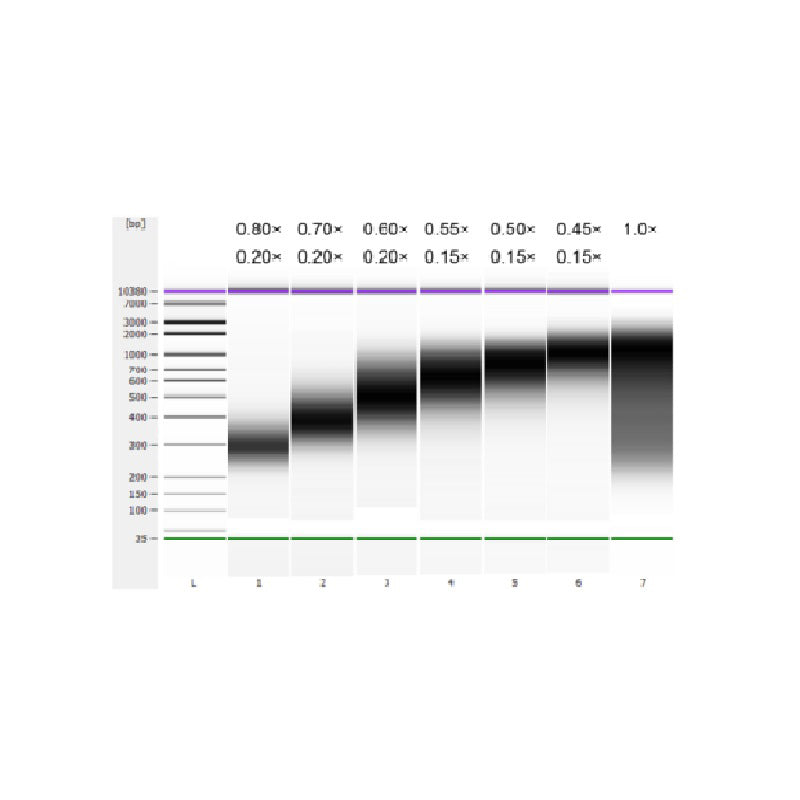
Die Kalbsthymus-DNA wurde durch Ultraschallbehandlung fragmentiert, um ein Fragment von 100–1.000 bp herzustellen, und es wurden zwei Runden der Größenauswahl gemäß Tabelle 1 durchgeführt. Die Ergebnisse wurden mit einem Agilent 2100 Bioanalyzer analysiert (Abbildung 2).
Tabelle 1. Empfohlene Bedingungen für die Auswahl der DNA-Größe
| Länge des DNA-Fragments | 250-350 bp | 320-420 bp | 450–550 bp | 550-700 bp | 700-900 bp | 800–1.000 Basispunkte |
| Perlenverhältnis: DNA für die 1. Runde | 0,80× | 0,70× | 0,60× | 0,55× | 0,50× | 0,45× |
| Perlenverhältnis: DNA für die 2. Runden | 0,20× | 0,20× | 0,20× | 0,15× | 0,15× | 0,15× |
Hinweis: „ד in der Tabelle gibt das Volumen der DNA-Probe an. Wenn beispielsweise die Insertlänge der Bibliothek 250 bp beträgt und das DNA-Probenvolumen 100 μL beträgt, beträgt das Volumen der in der ersten Sortierrunde verwendeten Magnetkügelchen 0,80 × 100 μL = 80 μL; das Volumen der in der zweiten Sortierrunde verwendeten Magnetkügelchen beträgt 0,20 × 100 μL = 20 μL.
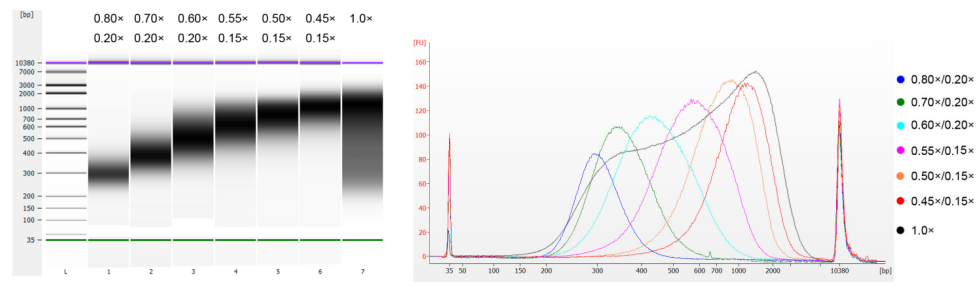
Abbildung 2. Agilent 2100 hochempfindliches DNA-Chip-Elektropherogramm
Hinweise:1. Tragen Sie zu Ihrer Sicherheit und Gesundheit bei Operationen bitte Laborkittel und Einweghandschuhe.
Zahlung und Sicherheit
Ihre Zahlungsinformationen werden sicher verarbeitet. Wir speichern weder Kreditkartendaten noch Zugriff auf Ihre Kreditkarteninformationen.
Anfrage
Sie können auch mögen
FAQ
Das Produkt ist nur für Forschungszwecke bestimmt und nicht für die therapeutische oder diagnostische Anwendung bei Menschen oder Tieren. Produkte und Inhalte sind durch Patente, Marken und Urheberrechte von
Für bestimmte Anwendungen sind möglicherweise zusätzliche geistige Eigentumsrechte Dritter erforderlich.