¿Qué hacer cuando los resultados de qPCR son extraños?
Los experimentos de qPCR pueden parecer fáciles y, a veces, difíciles. Una vez que se completa el diseño del cebador, se agregan muestras de qPCR paso a paso como PCR y el experimento se puede completar en la máquina. Existen varios problemas comunes en los experimentos de qPCR por el método de tinción y, dadas las posibles causas y soluciones, esperamos que pueda usarlas para resolver los problemas en los experimentos de qPCR.
1. ¿Qué es la qPCR?
2. Curva de amplificación qPCR anormal
3. Curva de fusión de qPCR anormal
4. Información para pedidos
5. Productos relacionados
6. Respecto a la lectura
1. ¿Qué es la qPCR?
Desde que se inventó la técnica de reacción en cadena de la polimerasa (PCR), la PCR es probablemente la técnica más utilizada en biología molecular debido a su simplicidad, bajo costo, confiabilidad, rapidez y alta sensibilidad. La qPCR es una tecnología desarrollada por PCR technology. Durante el proceso de amplificación de ADN, el método de detección de la cantidad total de productos después de cada ciclo de PCR con colorantes fluorescentes no solo tiene la rapidez y sensibilidad de la PCR, sino que también tiene más especificidad y alta, monitoreo en tiempo real, cuantificación repetible y precisa, y otras ventajas. La qPCR es una tecnología que utiliza un dispositivo de amplificación de PCR cuantitativa en tiempo real, es decir, un instrumento qPCR, para monitorear los productos de amplificación de ácidos nucleicos en tiempo real durante el proceso de PCR. La qPCR realiza el análisis cuantitativo de la plantilla de partida a través del análisis del valor Ct y la curva estándar. En 1992, el japonés Higuchi propuso por primera vez la "tecnología de PCR cuantitativa fluorescente en tiempo real". En 1996, la American Biological Company lanzó el primer instrumento de PCR cuantitativa fluorescente del mundo, que se compone de un sistema de ciclo térmico de amplificación de PCR, un sistema óptico de detección de fluorescencia y un software de computadora y aplicación. Puede monitorear productos de amplificación de ácidos nucleicos en tiempo real a través de tintes fluorescentes o sondas fluorescentes. , a través de la relación de función matemática, combinada con el software para el análisis de resultados, se realiza el cálculo de la cantidad de plantilla inicial de la muestra a analizar. Por lo tanto, la tecnología de PCR cuantitativa fluorescente en tiempo real ha sido ampliamente utilizada. Los métodos de etiquetado fluorescente para qPCR se dividen en método de mosaico de tinte fluorescente basado en el método de tinte SYBR Green I, método de sonda fluorescente basado en el método de sonda Taqman (Sonda Ciclista, Bracon Molecular, etc.), método de cebador de tinte extintor.
Los experimentos de qPCR pueden parecer fáciles y, a veces, difíciles. Una vez completado el diseño del cebador, se añaden muestras de qPCR paso a paso como en la PCR y el experimento se puede completar en la máquina. Sin embargo, hay muchos pequeños detalles que necesitan atención, como que no quede líquido en la pared de la tubería, que no haya burbujas en la tubería y que sea necesario minimizar el contacto directo entre la pared de la tubería, la cubierta de la tubería, etc., para reducir los errores experimentales o los resultados experimentales extraños. Xiaoyi ha recopilado varios problemas comunes en los experimentos de qPCR mediante el método de tinción y ha dado las posibles causas y soluciones, que esperamos que pueda utilizar para resolver los problemas en los experimentos de qPCR.
2. Curva de amplificación qPCR anormal
2.1 Anormalidades en la fase basal del gráfico logarítmico de la curva de amplificación
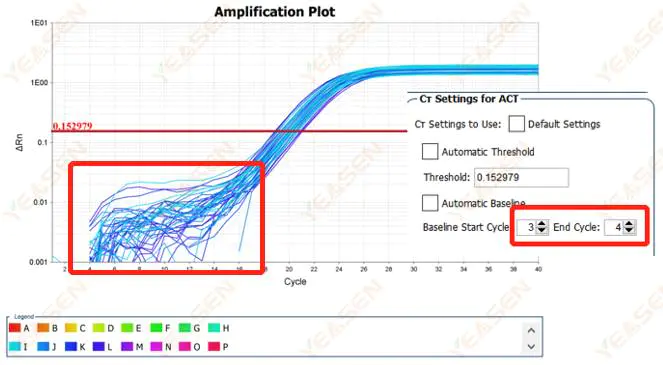
Figura 1. Gráfica de amplificación
Posibles causas:
Configuración de línea base incorrecta.
Soluciones:
Se recomienda aumentar el valor del punto final de la línea base.El diagrama de ejemplo de ajuste es el siguiente.

Figura 2. Gráfica de amplificación
2.2 Segmentación de la curva de amplificación del gráfico logarítmico
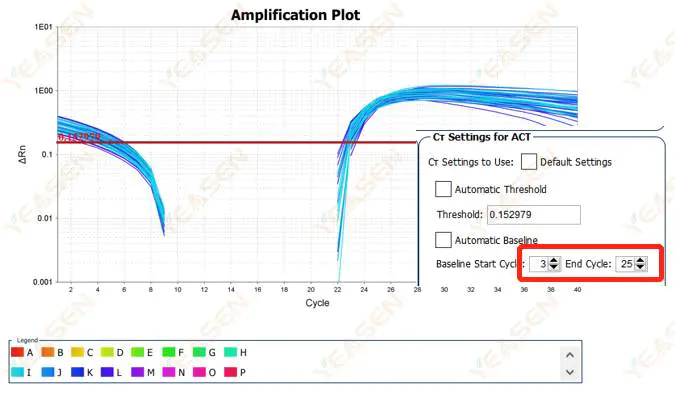
Figura 3. Gráfica de amplificación
Posibles causas:
La línea base está establecida demasiado alta.
Soluciones:
Se recomienda reducir el valor final de la línea base. El diagrama de ejemplo de ajuste es el siguiente.
2.3 Gráfico de linealidad de la curva de amplificación no suave
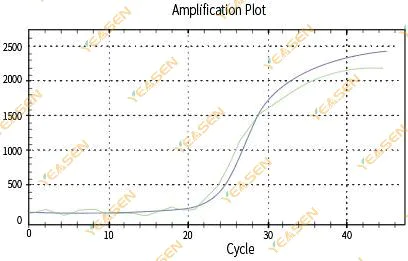
Figura 4. Gráfica de amplificación
Posibles causas:
1) El tubo de reacción de PCR no estaba bien tapado y la solución de reacción tuvo fugas.
2)Solución de reacción de PCR con pared colgante.
3) El instrumento no está calibrado (incluida la calibración automática o la calibración ROX).
4) El sistema tiene muchos inhibidores, lo que produce una fluorescencia inestable.
5)El uso excesivo del instrumento da como resultado una recolección de fluorescencia inestable.
Soluciones:
1) Presione firmemente la cubierta de la tubería.
2) Mezcle bien el reactivo y colóquelo con cuidado en el instrumento cuantitativo después de la centrifugación completa.
3)Calibrar el instrumento.
4) Mejorar la pureza del ARN y seleccionar reactivos de transcripción inversa adecuados.
2.4 Gráfico lineal de curva amplificada en zigzag en una meseta
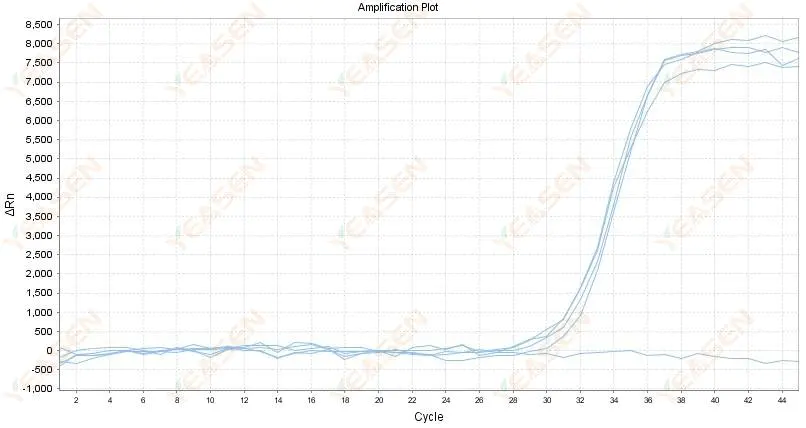
Figura 5. Gráfica de amplificación
Posibles causas:
1) La pureza del ARN es pobre y contiene muchas impurezas.
2) El instrumento se utiliza durante demasiado tiempo.
Soluciones:
1) Reextraer ARN de alta calidad.
2) Diluir la plantilla de ARN para reducir la concentración de impurezas.
3) Calibrar el instrumento.
2.5 El gráfico lineal de la curva de amplificación no logra alcanzar una meseta
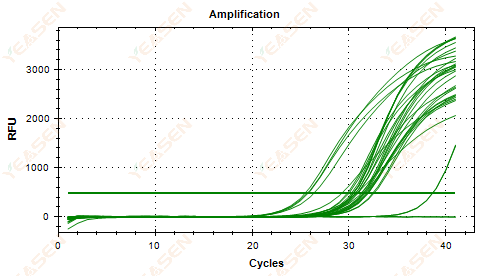
Figura 6. Gráfica de amplificación
Posibles causas:
1) Baja concentración de plantilla (valor Ct alrededor de 35).
2) Muy pocos ciclos de amplificación.
3) Baja eficiencia de amplificación del reactivo (Ct pequeño, pero también incapaz de alcanzar la meseta).
Soluciones:
1) Aumentar la concentración de la plantilla.
2) Aumentar el número de ciclos.
3) Aumento de Mg2+ concentración.
2.6 Curva de amplificación Trazado lineal Meseta de hundimiento
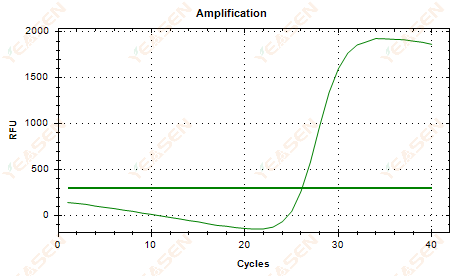
Figura 7. Gráfica de amplificación
Posibles causas:
1) Presencia de degradación (degradación del producto de amplificación, degradación de SYBR).
2) La tapa del tubo no está bien cubierta y el reactivo se volatiliza.
3) La concentración de ADNc es demasiado alta (si el valor Ct es demasiado pequeño, el umbral de fluorescencia se eleva y la flacidez es más grave).
4) Burbujas de aire en el tubo, luego desaparecen.
Soluciones:
1) Mejorar la pureza del sistema.
2) Reducir la cantidad de ADNc (diluir la plantilla).
3) Disminuir el valor del punto final de la línea base.
2.7 Valor grande de Ct en la curva de amplificación
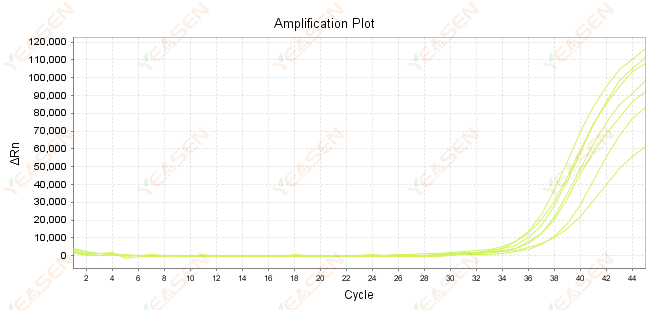
Figura 8. Gráfica de amplificación
Posibles causas:
1) Baja cantidad de plantillas.
2) Baja eficiencia de amplificación.
3) El fragmento de PCR es demasiado largo.
4) Presencia de inhibidores en el sistema de reacción.
Soluciones:
1) Reducir los tiempos de dilución o aumentar la cantidad de plantilla para que el valor Ct se sitúe entre 15 y 30 tanto como sea posible.
2) Optimice las condiciones de reacción, pruebe un procedimiento de amplificación de tres pasos o rediseñe los cebadores.
3) La longitud del producto de PCR está diseñada para estar entre 100 y 150 pb, no se recomienda superar los 300 pb.
4) Reproducción de plantillas de mayor pureza.
2.8 Mala repetibilidad de las curvas de amplificación
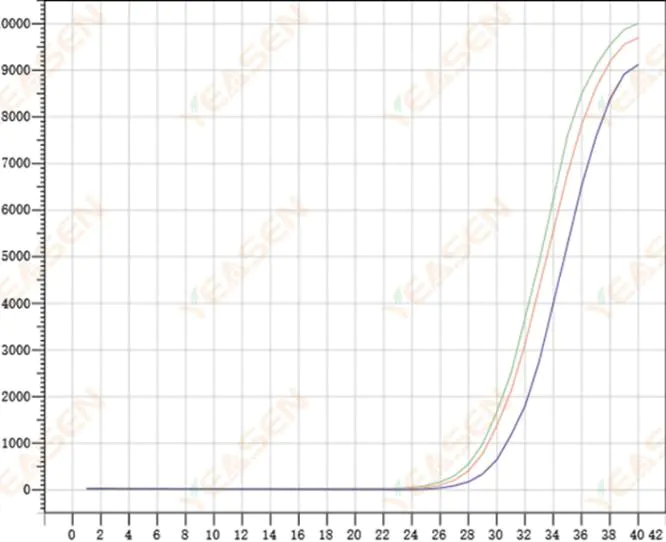
Figura 9. Gráfica de amplificación
Posibles causas:
1) Grandes errores de picos.
2) Los reactivos y los sistemas no están bien mezclados.
3) Bajo número de copias de ADNc.
4) No se utilizó calibración Rox.
Soluciones:
1) Calibración de pipetas.
2) Mezcla completa del sistema de reacción.
3) La baja concentración de plantilla, la repetibilidad deficiente, 4-6 orificios compuestos y 1-2 desviaciones de valores mayores se pueden descartar de forma adecuada.
4) Lo mejor es utilizar la calibración Rox. Si el reactivo utilizado no contiene Rox, el colorante de referencia debe seleccionarse como Ninguno.
2.9 Curvas de amplificación aleatorias
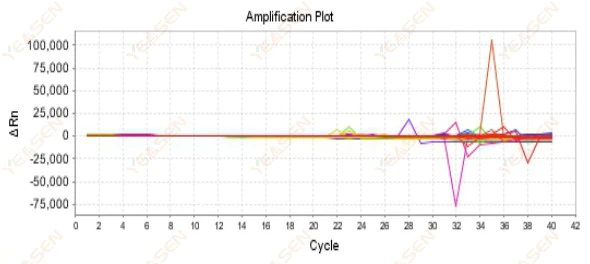
Figura 10. Gráfica de amplificación
Posibles causas:
El desajuste entre la concentración de Rox y el modelo.
Soluciones:
Cambie la configuración del tinte de referencia en el instrumento de ROX a NINGUNO y vea si la curva de amplificación vuelve a la normalidad.
2.10 Pico NTC
2.10.1 Ct>35, valor Tm de la curva de fusión <80℃
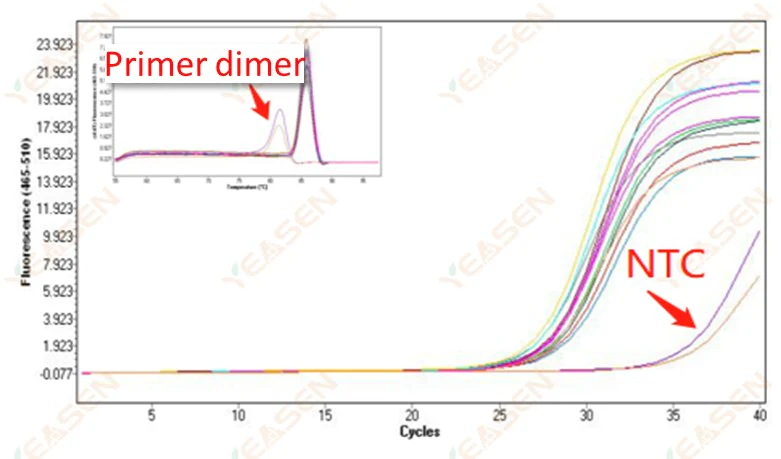
Figura 11. Gráfica de amplificación
Posibles causas:
Resultados de dimerización de cebadores.
Soluciones:
Primers optimizados.
2.10.Valor 2 Ct <35, la curva de fusión NTC y la forma del pico de la curva de fusión del gen se superponen
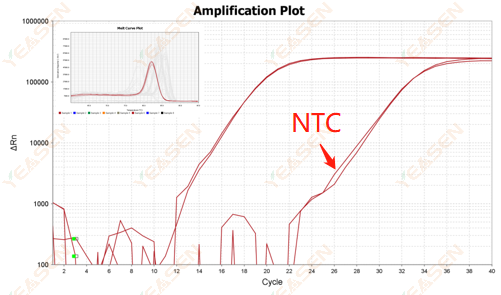
Figura 12. Gráfica de amplificación
Posibles causas:
Contaminación del sistema de reacción.
Soluciones:
Comprueba las fuentes de contaminación una por una.
3. Curva de fusión de qPCR anormal
3.1 Curva de fusión de pico único pero no pronunciada
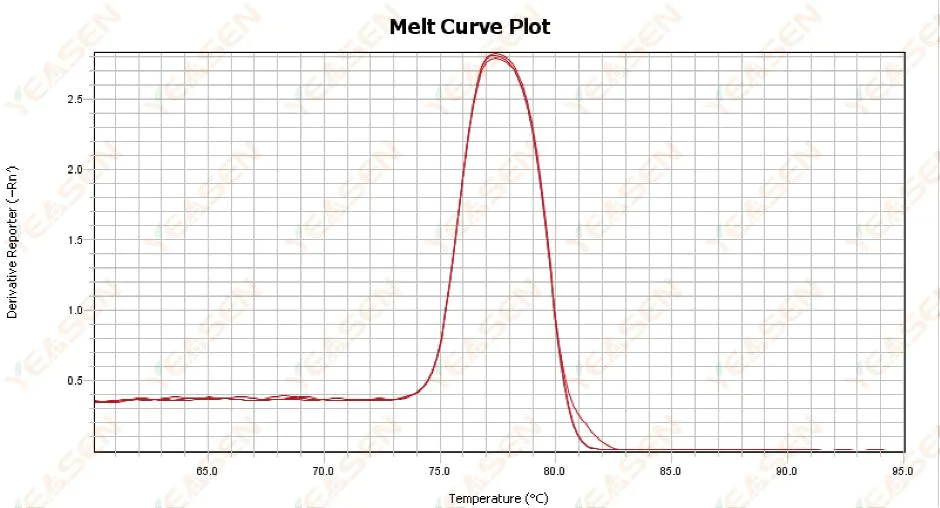
Figura 13. Gráfica de la curva de fusión
Posibles causas:
1) Está relacionado con la composición del reactivo y el modelo del instrumento.
2) Presencia de amplificación no específica de fragmentos de tamaño similar.
Soluciones:
1) El intervalo de temperatura desde el inicio hasta el final del pico no es superior a 7 °C. Los resultados se consideran utilizables, es decir, de un solo pico.
2) Se realizó una electroforesis de agarosa de alta concentración (por ejemplo, 3% de agarosa) para ayudar en la determinación.
3.2 La curva de fusión tiene doble pico y la temperatura máxima más baja es anterior a los 80 °C.
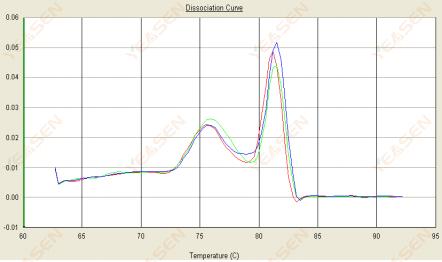
Figura 14. Curva de disociación
Posibles causas:
Presencia de dímeros de cebadores.
Soluciones:
Aumente la temperatura de recocido, disminuya la concentración de cebadores o rediseñe los cebadores.
3.3 La curva de fusión tiene doble pico y el pico inferior Tm es después de 80 °C.
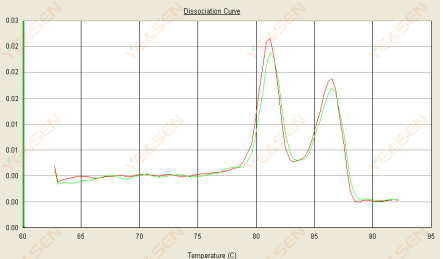
Figura 15. Curva de disociación
Posibles causas:
1) La baja especificidad del cebador conduce a la amplificación de productos no específicos.
2) contaminación del ADNg.
Soluciones:
1) Verifique la especificidad del cebador y rediseñe si es deficiente.
2) Confirmación mediante control negativo NRC, si lo hubiera, para volver a preparar la plantilla.
3.4 Picos de curva de fusión desordenados
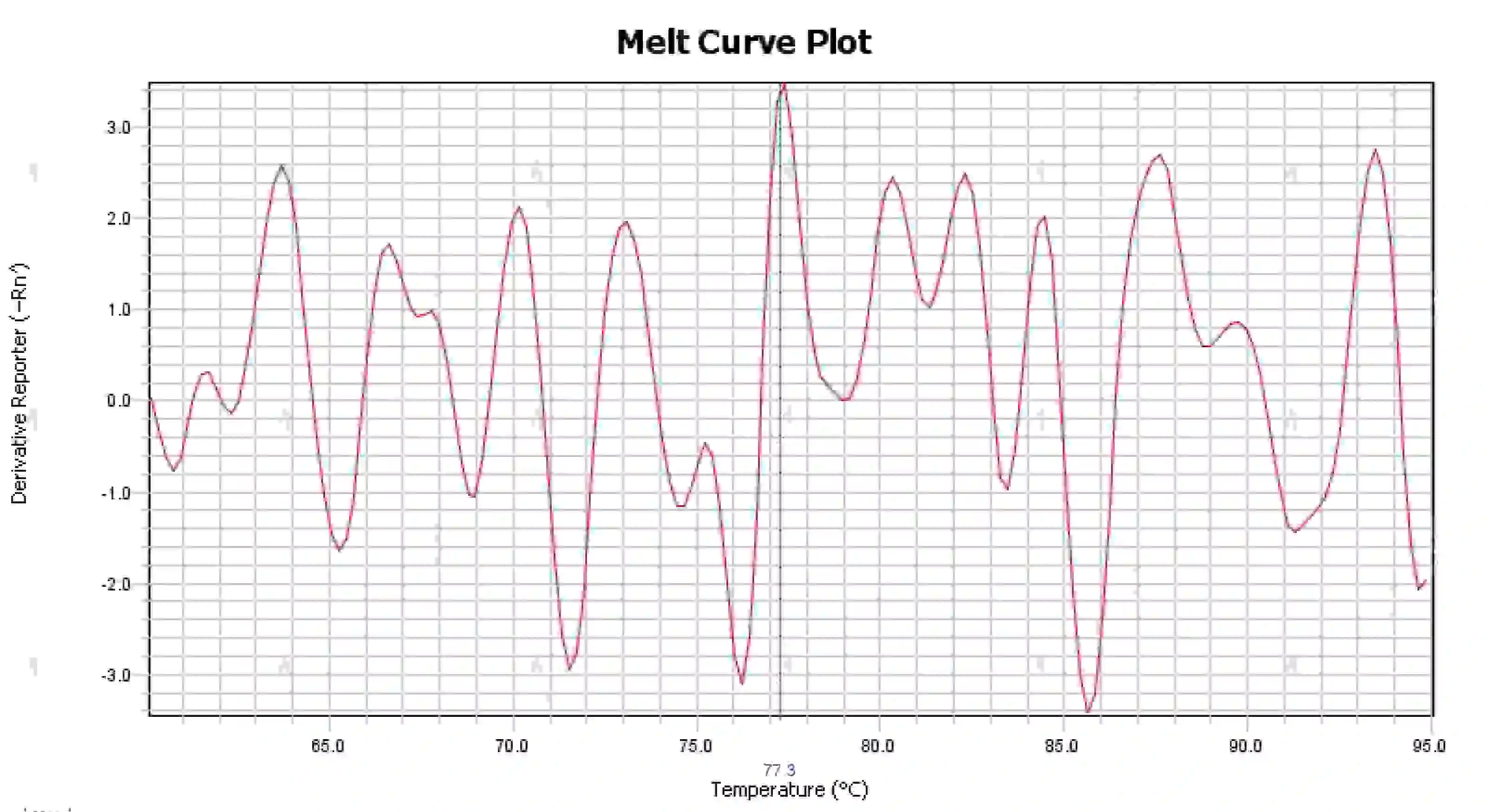
Figura 16. Gráfica de la curva de fusión
Posibles causas:
1) Contaminación del sistema de reacción.
2) La exposición de los reactivos a la luz brillante o a altas temperaturas provoca la falla del reactivo.
3) El instrumento no ha sido calibrado durante mucho tiempo.
4) Desajuste entre consumibles e instrumentos.
Soluciones:
1) Al combinar los resultados de NTC y NRC para confirmar la contaminación, se recomienda excluir la contaminación del agua, los cebadores, las enzimas y el medio ambiente uno por uno.
2) Se recomienda realizar experimentos comparativos con nuevos reactivos.
3) Se recomienda realizar un mantenimiento periódico de la calibración del instrumento.
4) Confirmar los requisitos del instrumento correspondiente para consumibles.
3.5 Picos espurios en el extremo delantero de la curva de fusión
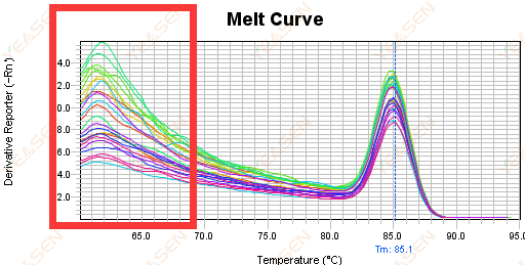
Figura 17. Gráfico de curva de fusión
Posibles causas:
La concentración de Rox no coincide con el modelo.
Soluciones:
Se recomienda cancelar la corrección de Rox para ver si la curva de fusión es normal.
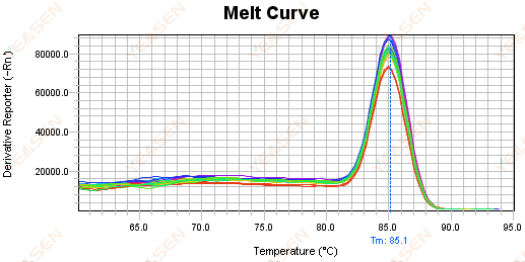
Figura 18. Gráfica de la curva de fusión
4. Información para pedidos
Tabla 1. Información para pedidos
| Nombre del producto | Código del producto | Especificación |
| Mezcla maestra para qPCR Hieff Unicon™ Universal Blue (basada en colorante) | 11184ES03 | 1 ml |
| 11184ES08 | 5×1 ml | |
| 11184ES50 | 50 x 1 ml | |
| 11184ES60 | 100×1 ml |
5. Productos relacionados
Los productos proporcionados por Yeasen son los siguientes.
Tabla 2. Productos relacionados
| Posicionamiento del producto | Nombre del producto | Código del producto |
| Extracción de ADN genómico y transcripción inversa en un solo paso | Hifair™V SuperMix de digestión RT-gDNA de un solo paso para qPCR (Consulta) | 11142ES |
| Mezcla maestra de alta sensibilidad (con paso de eliminación de ADN genómico) | SuperMix de síntesis de ADNc de primera hebra Hifair™ III para qPCR (digestor de ADNg plus) | 11141ES |
| Kit de alta sensibilidad (con paso de eliminación de ADN genómico) | Kit de síntesis de ADNc de primera hebra Hifair™ III (digestor de ADNg plus) (Consulta) | 11139ES |
| Mezcla maestra cuantitativa ordinaria (método de sonda) | Mezcla maestra de sonda TaqMan qPCR Hieff™ (Consulta) | 11205ES |
6. Respecto a la lectura
DNasa I y sus aplicaciones en biomedicina
Guía de selección de la transcriptasa inversa
