DNasa I y sus aplicaciones en biomedicina
La desoxirribonucleasa I (DNasa I) es una endonucleasa, cuya aplicación no es solo para mantener la integridad del ARN sino también para el análisis de la huella de ADN, la generación de bibliotecas de ADN aleatorias, la reducción de la adhesividad en lisados celulares o extractos de proteínas, etc. En una palabra, la DNasa I se puede utilizar en casi cualquier aplicación que requiera escisiones enzimáticas del ADN. A continuación se presenta una introducción detallada a la DNasa I y su aplicación específica.
1. ¿Qué es la DNasa I?
2. DNasa I para la preparación de extracción de ARN sin ADN
3. DNasa I para la transcripción in vitro para eliminar el ADN molde
4. DNasa I para la eliminación del ARNr
5. DNasa I para el etiquetado del ADN
6. Otras aplicaciones
7. Guía de selección de productos de DNasa I
1. ¿Qué es la DNasa I?
Desoxirribonucleasa I (DNasa I) es una endonucleasa no específica que puede digerir ADN monocatenario o bicatenario, presente en diferentes tejidos y fluidos corporales. Puede hidrolizar enlaces fosfodiéster para producir mono y oligodesoxinucleótidos que contienen un grupo 5'-fosfato y un grupo 3'-OH. El rango de pH de trabajo óptimo de la DNasa I es 7-8, su actividad depende de Ca2+ y puede ser activada por iones metálicos divalentes como Mn2+, Mg2+, Zn2+, etc. En presencia de Mg2+, la DNasa I corta aleatoriamente cualquier sitio de ADN bicatenario; en presencia de Mn2+, la DNasa I puede cortar ADN bicatenario en el mismo sitio para formar un extremo romo o extremos pegajosos de 1-2 nucleótidos.
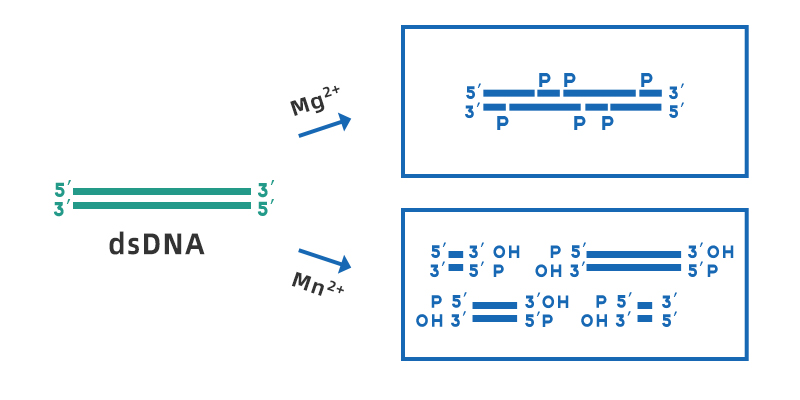
Figura 1. Diagrama esquemático de la escisión del dsADN por la DNasa I en presencia de Mg2+ y Mn2+.
Aunque las escisiones de la DNasa I se consideran generalmente escisiones no específicas, es más probable que actúe sobre ciertos fragmentos de secuencia, como la región del surco menor, y es más propensa a escindir secuencias de purina-pirimidina. Sin embargo, cuando la DNasa I actúa sobre dsADN heterogéneo, se escindirán las cuatro bases y el efecto sobre una base específica no será más de 3 veces mayor que el de otras bases.
2. DNasa I para la preparación de extracción de ARN sin ADN
En los experimentos biológicos, el primer paso es preparar el ácido nucleico para estudiar las distintas funciones del ARN. Sin embargo, dado que el ADN y el ARN suelen liberarse juntos durante el proceso de lisis celular, la interferencia entre ambos no se puede evitar sin importar qué solución de extracción se utilice, por lo que se deben utilizar enzimas específicas para eliminar la interferencia. Para una extracción de ARN de alta calidad, se utiliza la DNasa I para eliminar el ADN residual de la muestra.
La DNasa I puede degradar el ADN bicatenario y monocatenario en oligonucleótidos y nucleótidos simples, y el ADN en el producto de preparación del ARN puede degradarse de manera efectiva. Luego, la DNasa I se inactiva mediante calentamiento con un tampón de parada. Durante el proceso de calentamiento, se puede abrir la estructura de horquilla de la molécula de ARN, lo que facilita la entrada directa del ARN en el proceso de transcripción inversa.
La calidad del ARN afectará directamente a los datos experimentales en gran medida. En general, los residuos de ADN genómico no se pueden evitar por completo durante la extracción de ARN, por lo tanto, generalmente se recomienda tratar las muestras de ARN con DNasa I para digerir el ADN genómico residual antes de realizar aplicaciones posteriores (por ejemplo, análisis de expresión de ARNm, análisis de transcriptoma, etc.). El paso de digestión del ADN genómico se puede realizar durante la extracción de ARN, después de la extracción de ARN o antes de la transcripción inversa del ARN.Según el posicionamiento del producto, los productos que ofrece Yeasen son los siguientes:
Tabla 1: Lista de productos relacionados con la eliminación de ADN o la extracción de ARN o antes de la transcripción inversa
| Posicionamiento del producto | Nombre del producto | Gato # |
| Extracción de ARN | Reactivo de extracción de ARN total TRIeasy™ [preguntar] | 10606ES |
| 19221ES | ||
| Kit de ARN MolPure™ Plant Plus [preguntar] | 19292ES | |
| Kit de ADN/ARN viral MolPure™ [preguntar] | 19321ES | |
| Eliminación de ADN genómico | 10325ES | |
| Transcripción inversa | SuperMix de síntesis de ADNc de primera hebra Hifair™Ⅲ para qPCR (digestor de ADNg plus) | 11141ES |
| PCR cuantitativa | 11184ES |
3. DNasa I para la transcripción in vitro para eliminar el ADN molde
La transcripción in vitro (IVT) utiliza principalmente ADN como plantilla, además de los sustratos y tampones correspondientes para obtener ARN mediante la transcripción in vitro. En los experimentos de transcripción in vitro, las ARN polimerasas como T7, T3 y SP6 se utilizan comúnmente para la síntesis de ARN. El ARN sintetizado puede tener residuos de ADN. La eliminación de los residuos de ADN es beneficiosa para el desarrollo de experimentos posteriores. Por ejemplo, en la etapa de desarrollo de la vacuna de ARNm, la eliminación de residuos es un paso crítico, que puede reducir la dificultad de la purificación posterior y aumentar la pureza del producto. La plantilla de ADN se elimina típicamente utilizando ADNasa I recombinante (libre de ARNasa).Según el proceso de síntesis de ARNm, los productos proporcionados por Yeasen son los siguientes:
| Proceso de síntesis del ARNm | Nombre del producto | Gato# |
| Preparación de plantillas | ADN polimerasa de alta fidelidad Hieff Canace™ Plus [preguntar] | 10148ES |
| 10922ES | ||
| 10125ES | ||
| FuniCut™BsaI [preguntar] | 15005ES | |
| FuniCut™ XbaI [preguntar] | 15033ES | |
| BspQI[consultar] [preguntar] | 16215ES | |
| Transcripción in vitro | 10623ES | |
| 10624ES | ||
| ARN polimerasa T7 (50 U/μL)[preguntar] | 10618ES | |
| Solución de conjunto NTP (ATP, CTP, UTP, GTP, 100 mM cada uno) | 10133ES | |
| 10620ES | ||
| 10621ES | ||
| Eliminar la plantilla de ADN | 10611ES | |
| modificación del ARNm | Enzima de protección contra la vaccinia con ARNm UCF.ME™ de grado GMP | 10614ES |
| 10612ES | ||
| 10132ES | ||
| 10619ES | ||
| Purificación de ARNm | 12602ES |
4. DNasa I para la eliminación del ARNr
In vivo, el ARNr es muy abundante y muy conservador, lo que tiene poca importancia para obtener información biológica, por lo que a menudo se elimina primero en la construcción y secuenciación de la biblioteca de ARN.En la actualidad, el método de eliminación del ARNr es principalmente la digestión con ARNasa H. Los pasos principales de la eliminación del ARNr basada en enzimas se muestran en la figura 2: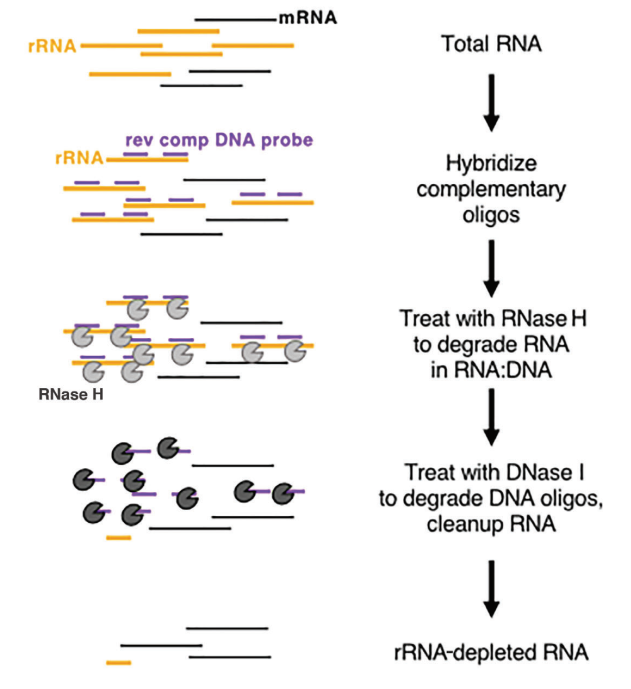
Figura 2: Diagrama esquemático del principio de depleción de ARNr basada en enzimas (Baldwin, A. et al. 2021, Current Protocols)
En primer lugar, se extrae el ARN total, luego se hibrida la sonda de ADN monocatenario con el ARNr, se diseña y sintetiza una sonda de ADN monocatenario específica para el ARNr, luego se utiliza la ARNasa H para degradar el ARNr hibridado y se utiliza la ADNasa I para degradar la sonda de ADN. Por último, se deja la plantilla de ARN que no es ARNr. Los productos relacionados con la eliminación del ARNr proporcionados por Yeasen son los siguientes:
| Proceso de síntesis del ARNm | Nombre del producto | Gato# |
| Humano/Ratón/Rata Agotamiento del ARNr | Alto NGS™ Kit de depleción de ARNm MaxUp (humano/ratón/rata) MaxUp [preguntar] | 12253ES |
| Agotamiento del ARNr en plantas | 12254ES | |
| Eliminación de ARN ribosómico y regiones 45S ITS/ETS del ARN total humano | Alto NGS™ Kit de depleción de ARNr humano MaxUp (ARNr e ITS/ETS) | 12257ES |
| Degradación del ARNr | 12906ES | |
| Degradación de la sonda de ADN | 10325ES |
5.DNasa I para el etiquetado del ADN
La traducción de nick es uno de los métodos de marcaje de sondas de ácido desoxirribonucleico más utilizados en el laboratorio. Este método utiliza varias actividades enzimáticas de la ADN polimerasa I para incorporar trifosfatos de desoxirribonucleósidos marcados en cadenas de ADN recién sintetizadas. De este modo, se sintetizan sondas de ADN marcadas de manera uniforme para una alta actividad específica. Las características de la traducción de nick son sondas rápidas, simples, deliberadas, de alta especificidad y marcadas de manera uniforme, que son adecuadas para ADN de doble cadena más largo.El método se realiza mediante la acción combinada de la ADNasa I y la ADN polimerasa I de E. coli. Los pasos principales del marcaje de ADN por traducción de mellas se muestran en la figura 3:
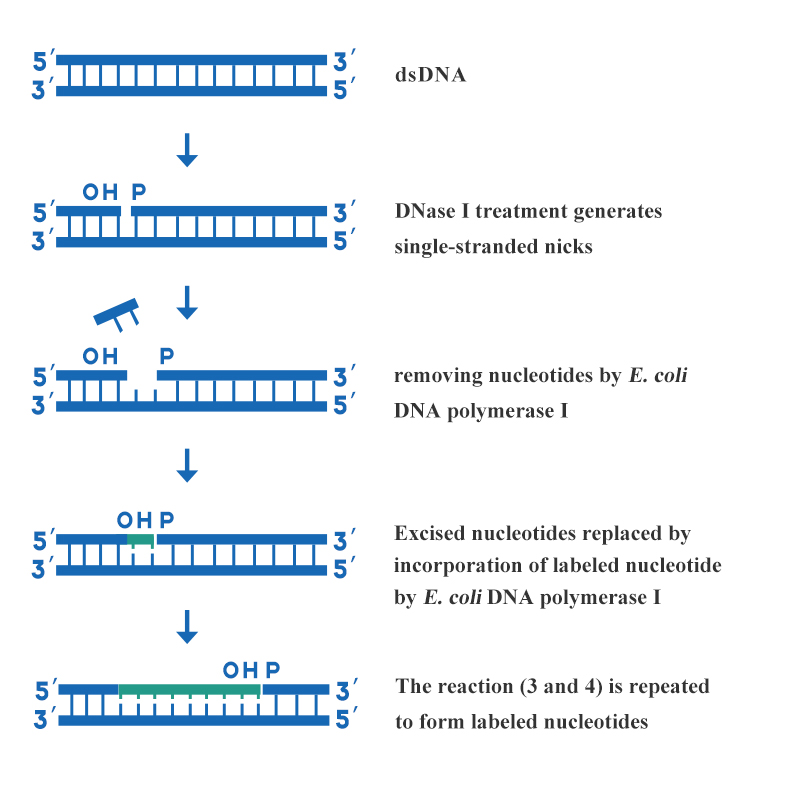
Figura 3: Diagrama esquemático del etiquetado de ADN por traducción de mellas
Se utiliza una concentración adecuada de DNasa I para crear varios huecos monocatenarios en cada hebra del ADN bicatenario que se va a marcar, y el extremo hidroxilo 3' se forma en el hueco. Se utiliza la actividad exonucleasa 5'→3' de la ADN polimerasa I de E. coli para cortar un nucleótido del extremo 5' del corte y, al mismo tiempo, la actividad polimerasa 5'→3' de la ADN polimerasa I de E. coli introduce un nucleótido marcado con el extremo 3' del hueco para repararlo. A medida que el hueco se mueve a lo largo de la hebra de ADN, los nucleótidos marcados se incorporan a la hebra recién sintetizada.Los productos relacionados con el etiquetado de ADN proporcionados por Yeasen son los siguientes:
| Posicionamiento del producto | Nombre del producto | Gato# |
| Común | Desoxirribonucleasa I (DNasa I) del páncreas bovino [preguntar] | 10607ES/10608ES |
| Libre de ARNasa | 10325ES | |
| E. coli fuente | 12903ES |
6. Otras aplicaciones
Las anteriores son varias aplicaciones de uso común. Otras aplicaciones de la DNasa I incluyen las siguientes, como el ensayo de huella de DNasa I y los sitios hipersensibles a la DNasa I. El ensayo de huella de DNasa I es un método de detección que puede identificar con precisión los sitios de unión de las proteínas de unión al ADN en el ADN. Cuando una proteína se une a un fragmento de ADN, puede proteger el sitio de unión de ser dañado por la DNasa I y los fragmentos de ADN quedarán atrás después de la digestión enzimática ("huella"), y se puede determinar su secuencia. En la imagen del gel, no hay bandas donde el ADN se una a la proteína. Para leer más haga clic en el enlace.Los sitios hipersensibles a la DNasa I se refieren a las escisiones en un pequeño número de sitios específicos cuando la cromatina se trata con niveles bajos de DNasa I y estos sitios específicos se denominan sitios hipersensibles a la DNasa I. El principio es que cuando un gen está en un estado transcripcionalmente activo, la cromatina que contiene el gen es significativamente más sensible a la degradación por DNasa que la región inactiva. Para leer más, haga clic en el enlace.
7. Guía de selección de productos de DNasa I
Yeasen Biotechnology (Shanghai) Co., Ltd., fundada en 2014, es una empresa de alta tecnología dedicada a la I+D y producción de materias primas enzimáticas y anticuerpos antigénicos. Sus productos incluyen enzimas, proteínas y anticuerpos de diagnóstico molecular utilizados en productos farmacéuticos, pruebas de seguridad alimentaria, cría, justicia y otras industrias. Nos comprometemos a proporcionar a los clientes del campo de las ciencias biológicas productos y servicios de alta calidad. Las pautas de compra de productos para DNasa I son las siguientes:| Nombre del producto (Cat.#) | Posicionamiento del producto | Aplicaciones recomendadas |
| DNasa I de páncreas bovino (CAT#10607,10608)[preguntar] | Se eliminó la ARNasa, no se detectó | Se utiliza principalmente en la investigación de proteínas: eliminación de ADN de preparaciones de proteínas. |
| DNasa I recombinante (libre de ARNasa)(CAT#10325) | Libre de ARNasa, para investigación | Ideal para una variedad de aplicaciones: eliminación de ADN de preparaciones de ARN y proteínas como bibliotecas de ADNc sensibles a la ARNasa o preparación de muestras para experimentos de RT-PCR. |
| Desoxirribonucleasa I (DNasa I) de grado GMP UCF.ME™(CAT#10611) | Libre de ARNasa, grado farmacéutico GMP. | Ideal para una variedad de aplicaciones: eliminación de ADN de preparaciones de ARN y proteínas como bibliotecas de ADNc sensibles a la ARNasa o preparación de muestras para experimentos de RT-PCR. |
Respecto a la lectura:
Reactivos de grado GMP para la síntesis in vitro de ARNm
Los principios de la huella de ADNasa I y sus aplicaciones biomédicas
Referencias
1. Baldwin A, Morris AR, Mukherjee N. Un método fácil, rentable y escalable para agotar el ARN ribosómico humano para RNA-seq[J]. Protocolos actuales, 2021.
2. Song C, Zhang S, Huang H. Elección de un método adecuado para la identificación de orígenes de replicación en genomas microbianos [J]. Frontiers in Microbiology, 2015, 6:1049.
