Trabajar en ambas líneas facilita el análisis de la expresión de miRNA
La longitud del miRNA maduro es de tan solo 20 nt aproximadamente, por lo general, el cebador directo cubrirá toda su longitud o incluso tendrá una parte sobrante, y el cebador inverso no tendrá dónde colocarse. La solución actual del mercado es aumentar la longitud de la transcripción inversa durante la transcripción inversa. Los métodos comunes incluyen el método de cola y el método de tallo-bucle.
1. Introducción
2. Productos recomendados
3. Lista de productos relacionados
1. Introducción
Los microARN (miARN) son ARN pequeños monocatenarios con un tamaño de aproximadamente 21-23 bases, que se generan a partir de precursores de ARN monocatenarios con una estructura de horquilla de aproximadamente 70-90 bases después del procesamiento de la enzima Dicer. El miARN se une principalmente de manera específica a la región no codificante del extremo 3' del ARNm del gen diana y regula el proceso de traducción del ARNm del gen diana postranscripcional, bloqueando así la traducción del ARNm y promoviendo la degradación del ARNm, lo que resulta en una disminución de la expresión de proteínas. El papel de los miARN está involucrado en la aparición y el desarrollo de varias enfermedades cardiovasculares como la ontogenia, la diferenciación tisular, la proliferación celular y la apoptosis.
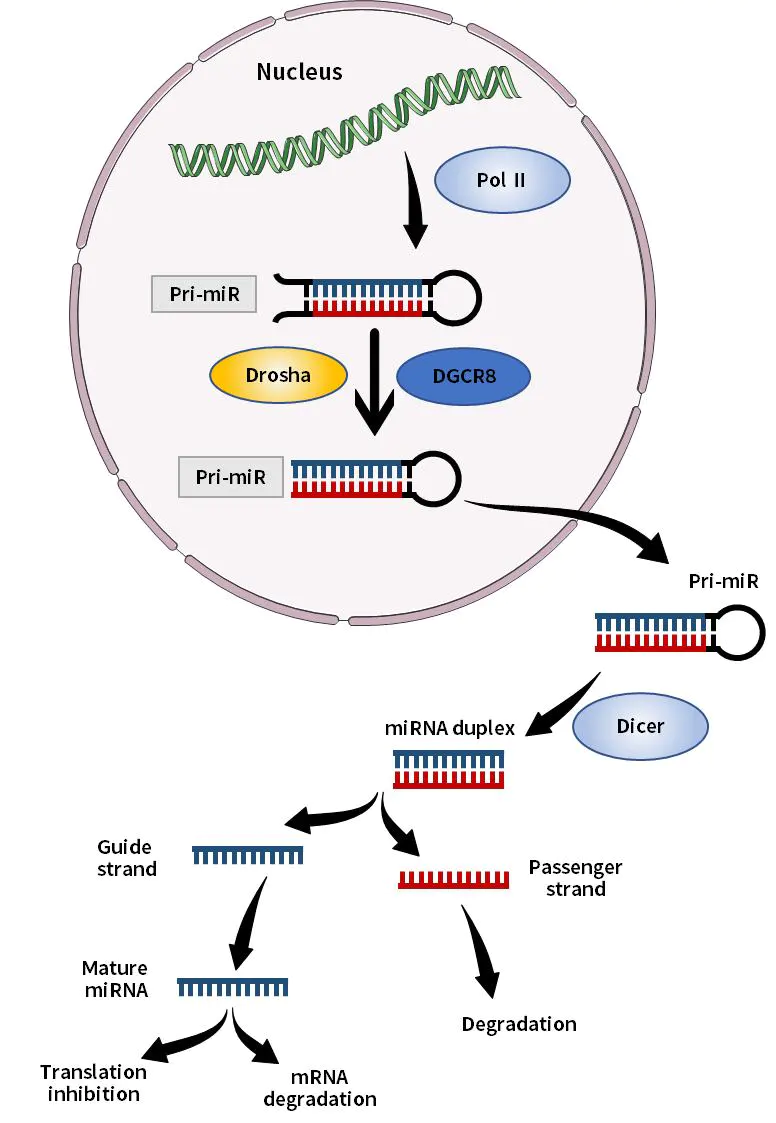
Figura 1. Diagrama esquemático de la síntesis de miRNA in vivo
La longitud del miRNA maduro es de tan solo 20 nt aproximadamente, por lo general, el cebador directo cubrirá toda su longitud o incluso tendrá una parte sobrante, y el cebador inverso no tendrá dónde colocarse. La solución actual del mercado es aumentar la longitud de la transcripción inversa durante la transcripción inversa. Los métodos comunes incluyen el método de cola y el método de tallo-bucle.
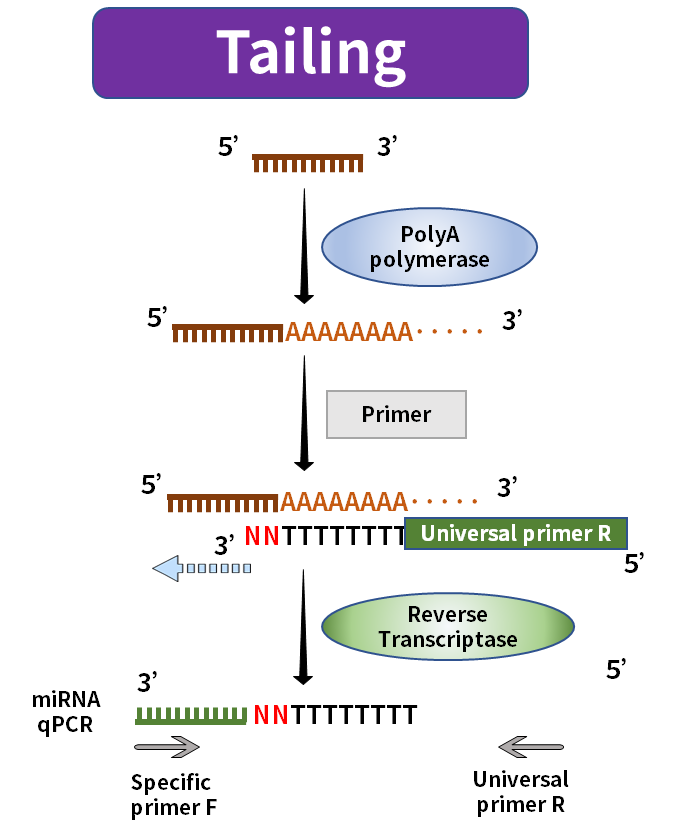
Figura 2. Síntesis de miRNA mediante el método de cola
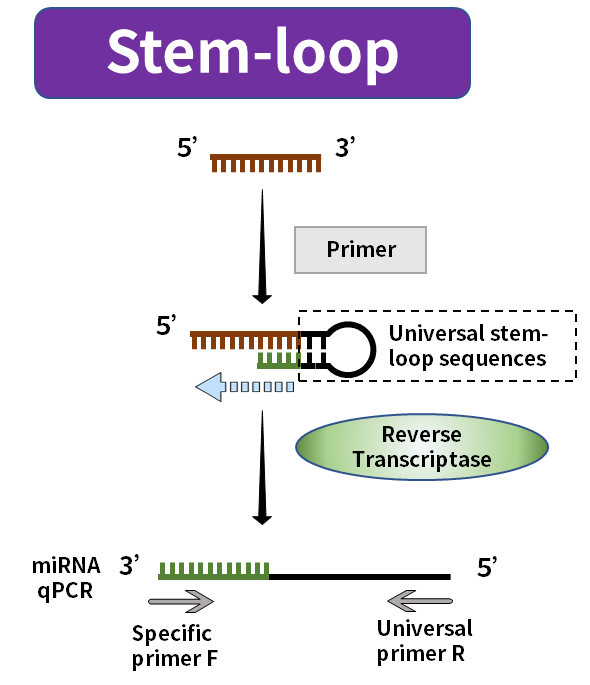
Figura 3. Síntesis de miRNA mediante el método de tallo-bucle
El método de cola utiliza la polimerasa PolyA para añadir una cola Poly(A) al miRNA maduro y utiliza Oligo-dT con la secuencia universal como cebador de transcripción inversa para sintetizar el miRNA alargado correspondiente a la primera cadena de ADNc. El método de tallo-bucle utiliza la secuencia universal plegada en la estructura de tallo-bucle como cebador para sintetizar la primera cadena del ADNc correspondiente al miRNA alargado. La estructura de tallo-bucle se abrirá a una temperatura adecuada durante el proceso de amplificación posterior. y se combina con los cebadores de amplificación pertinentes.
Tabla 1.Comparación entre cola y tallo-bucle
| Categoría | Cola | Tallo-bucle |
| Aplicabilidad | Adecuado para estudiar varios miRNA diferentes simultáneamente | Adecuado para la detección cuantitativa precisa de unos pocos miRNA en una muestra. |
| Ventaja | Una única transcripción inversa puede detectar múltiples miRNA con alto rendimiento | Solo se puede detectar un miRNA a la vez mediante transcripción inversa, con una fuerte especificidad. |
| Sistema | Junto con el miRNA involucrado, transcripción inversa en el mismo sistema de reacción con alta precisión | Los miRNA involucrados internamente se separan y se transcriben de manera inversa en los dos sistemas, lo que es propenso a errores. |
| Especificidad | ★★★★☆ | ★★★★★ |
| Sensibilidad | ★★★★★ | ★★★★☆ |
| Tiempo | ★★★★☆ | ★★★★★ |
2. Productos recomendados
2.1 Combinación cuantitativa de inversión de cola de miRNA de Yeasen (11148ES y 11171ES)
Figura 4. Kit de síntesis de ADNc de primera cadena de miRNA 11148ES-Hifair™
El kit adopta el método de cola para la transcripción inversa del ADNc de primera cadena de miRNA. El tampón de RT de miRNA 2×Hifair™ en el producto contiene todas las materias primas y los cebadores para la reacción de cola de miRNA y la reacción de transcripción inversa, y ha sido cuidadosamente optimizado. Puede garantizar que los procesos de modificación de poli(A) y transcripción inversa del extremo 3' de miRNA se lleven a cabo de manera eficiente y simultánea.
Figura 5. Mezcla maestra universal qPCR SYBR 11171ES -Hieff™ miRNA
Una nueva generación de reactivos de detección cuantitativa por PCR de fluorescencia premezclados desarrollados especialmente para la detección cuantitativa de miRNA, que contienen un colorante de referencia pasivo ROX especial, adecuado para todos los instrumentos de qPCR, sin necesidad de ajustar la concentración de ROX en diferentes instrumentos, solo en la reacción de preparación La amplificación se puede realizar agregando cebadores y plantillas al sistema. La ADN polimerasa adopta una polimerasa de inicio en caliente modificada químicamente y coopera con un sistema de tampón especial, lo que hace que la especificidad y la sensibilidad de la reacción sean más altas y puede cuantificar con precisión en un rango más amplio.
2.2 Rendimiento del producto
2.2.1 Compatible con instrumentos qPCR de todas las plataformas

Figura 6. Resultados compatibles con diferentes instrumentos de qPCR
Utilizando el ARN total de células 293T como plantilla, se realizó la transcripción inversa con el kit de síntesis de ADNc de primera hebra de miRNA Hifair™ (Cat# 11148ES).El ADNc obtenido se diluyó 10 veces y luego se utilizó Hieff™ miRNA Universal qPCR SYBR Master Mix (Cat# 11171ES) para detectar los genes de referencia internos de hsa-miR-let-7b-5p, hsa-miR-let-7c-5p y U6, respectivamente.
2.2.2 Alta tasa de detección, hasta 10 pg
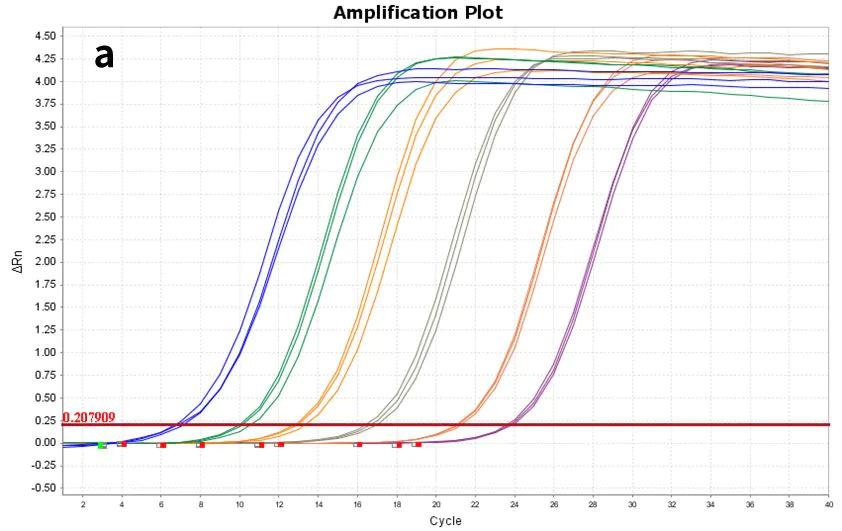
Figura 7. Gráfica de amplificación
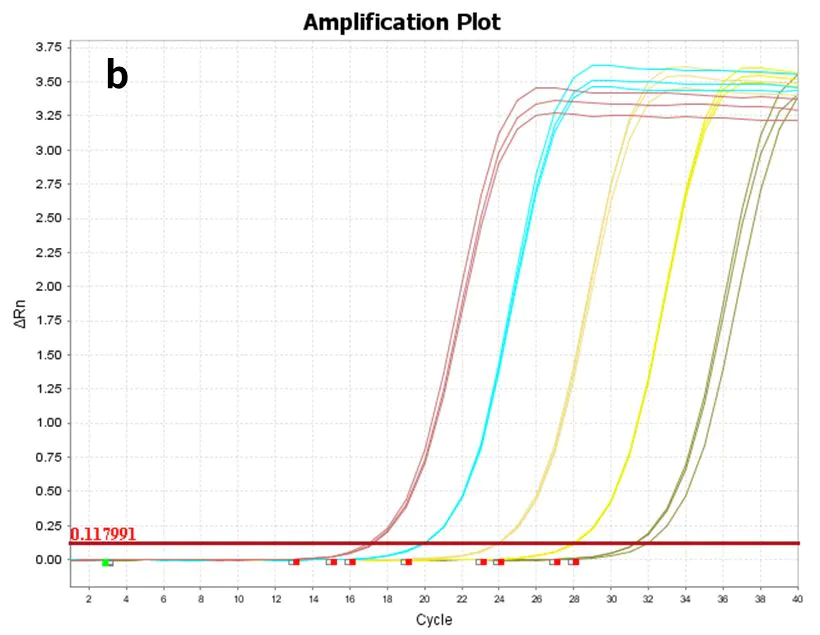
Figura 8. Gráfica de amplificación
Utilizando hsa-miR-let-7e-5p(a) sintético y ARN total de células 293T(b) como plantillas, diluidos a los siguientes gradientes, respectivamente: 60 copias a 606 copias (6 gradientes) y 10 pg-100 ng (5 gradientes), se transcribió de forma inversa con el kit de síntesis de ADNc de primera hebra de miRNA Hifair™ (n.° de cat. 11148ES) y el ADNc resultante se detectó con la mezcla maestra universal de qPCR SYBR de miRNA Hieff™ (n.° de cat. 11171ES) para la expresión del gen hsa-miR-let-7e -5p.
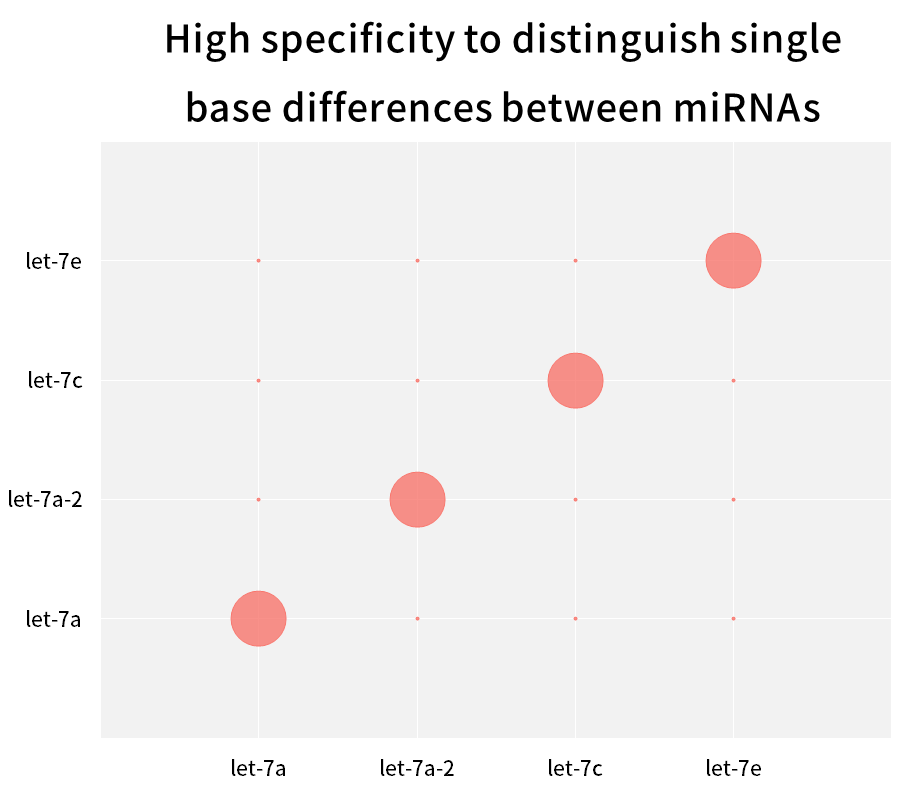
2.2.3 Alta especificidad, capaz de distinguir diferencias de bases individuales entre miRNAs de la misma familia
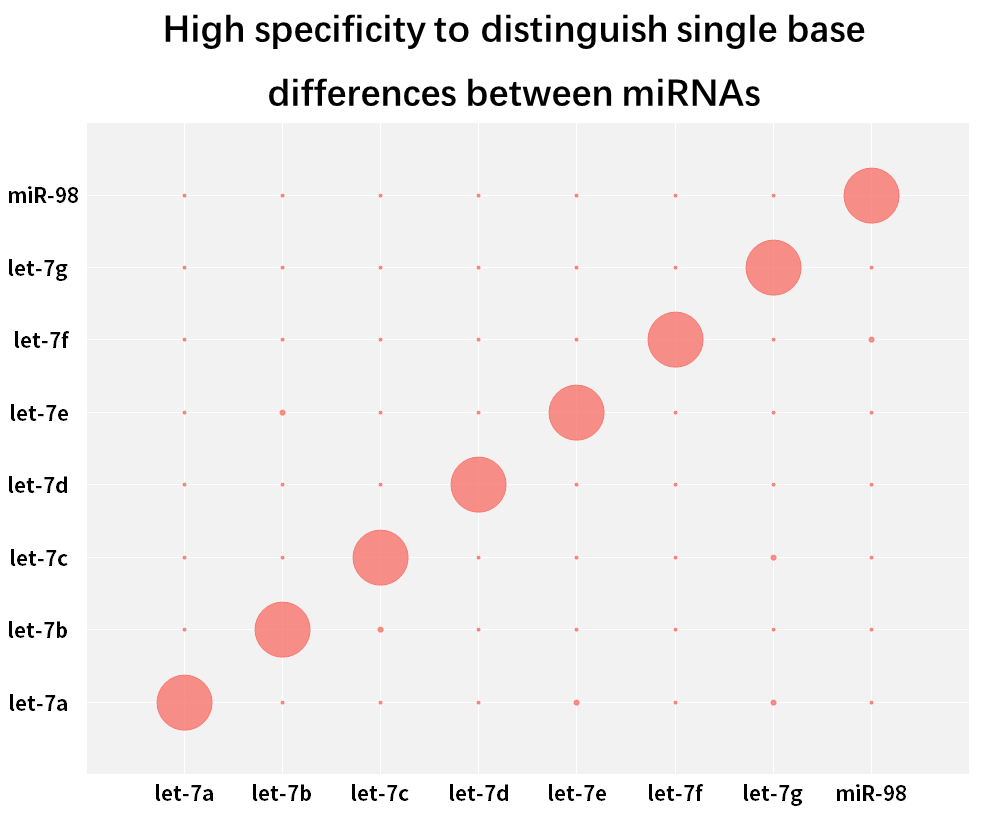
Figura 9. Distinguir diferencias de bases individuales entre los miRNA
La familia humana hsa-let-7 tiene múltiples miRNA, con pocas bases que difieren entre sí, o incluso con una sola diferencia de bases. Los miRNA sintéticos para cada isoforma Let-7 (hsa-miR-let-7a~Let-7i, has-miR-98) se transcribieron de forma inversa en ADNc utilizando el kit de síntesis de ADNc de primera hebra de miRNA Hifair™ (n.° de cat. 11148ES), utilizando la mezcla maestra Hieff™ miRNA Universal qPCR SYBR (n.° de cat. 11171ES) para amplificar estos miRNA por separado con diferentes cebadores y calcular la tasa de coincidencia utilizando la ecuación 2-ΔCT x 100 %. Los resultados muestran que los miembros de la familia de subtipos estrechamente relacionados se pueden distinguir de manera efectiva.
2.2.4 Excelente eficiencia de amplificación
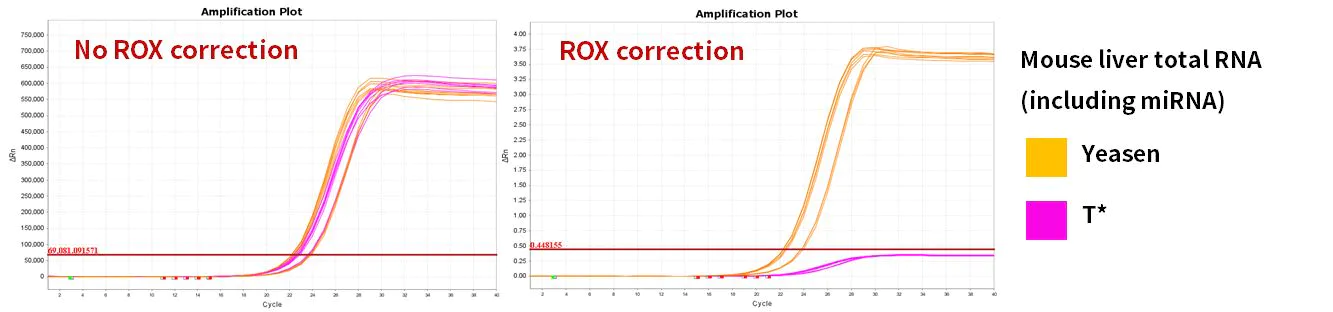
Figura 10. Eficiencia de amplificación
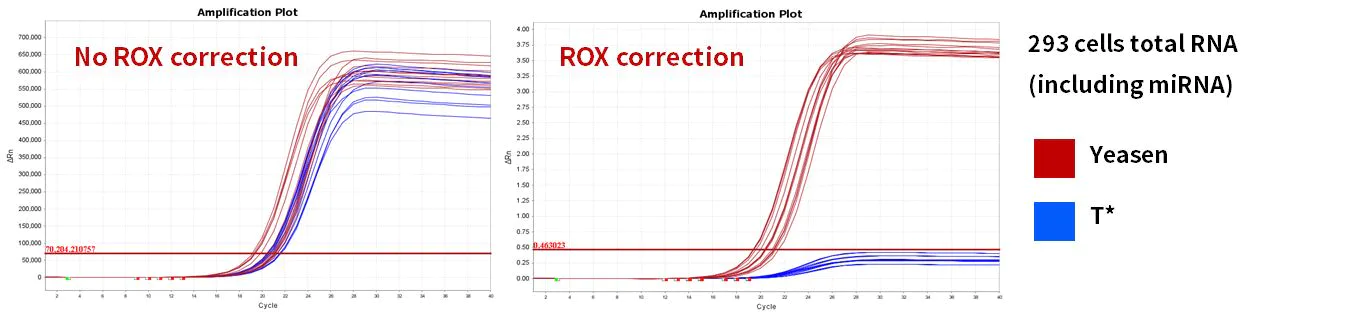
Figura 11. Eficiencia de amplificación
Los genes miR-let-7b-5p, miR-let-7c-5p, miR-let-7e-5p y miR-let-7f-5p se amplificaron utilizando células 293T humanas y ARN total de hígado de ratón como plantillas. Los resultados mostraron que, en comparación con productos similares, el kit de síntesis de ADNc de primera hebra de miRNA Hifair™ (n.° de cat. 11148ES) coincidió con el sistema de reacción de la mezcla maestra universal para qPCR SYBR de miRNA Hieff™ (n.° de cat. 11171ES) con un rendimiento excelente.
2.2.5 Excelente estabilidad

Figura 12. Análisis de estabilidad del producto a 37 ℃ durante 7 días

Figura 13. Análisis de resultados después de 50 ciclos de congelación y descongelación.
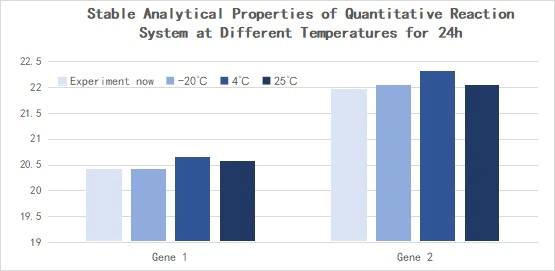
Figura 14. Propiedades analíticas estables del sistema de reacción cuantitativa a diferentes temperaturas durante 24 h.
La transcripción inversa se realizó con el kit de síntesis de ADNc de primera hebra de miRNA Hifair™ (n.° de cat. 11148ES) utilizando el ARN total de células 293T como plantilla, y el ADNc obtenido se diluyó 10 veces y se detectó con la mezcla maestra Hieff™ miRNA Universal qPCR SYBR (n.° de cat. 11171ES). Expresión de hsa-miR-let-7b-5p, hsa-miR-let-7c-5p, hsa-miR-let-7e-5p. △Ct<0,5.
2.3 Combinación cuantitativa de inversión de tallo-bucle de miRNA de Yeasen (11139ES y 11170ES)
Figura 15. Kit de síntesis de ADNc de primera cadena 11139ES-Hifair™ III (digestor de ADNg plus)
El kit fue desarrollado con base en la transcriptasa inversa Hifair™ III.La velocidad de síntesis de ADNc por enzima es rápida y la estabilidad térmica mejora considerablemente. Al mismo tiempo, la enzima mejora la afinidad con la plantilla y es adecuada para la transcripción inversa de una pequeña cantidad de plantillas y genes de bajo número de copias. La capacidad de la transcriptasa inversa Hifair™ III para sintetizar ADNc de longitud completa también se ha mejorado, lo que permite la síntesis de ADNc de hasta 19,8 kb.

Figura 16. Mezcla maestra universal qPCR SYBR 11170ES-Hieff™ miRNA
Este producto es una mezcla maestra especial para la cuantificación de miRNA mediante el método de fluorescencia quimérica SYBR Green I. Debido a que las secuencias de miRNA son cortas y las secuencias de miRNA de la misma familia suelen ser muy similares, la especificidad es extremadamente necesaria durante la cuantificación. Este producto se basa en la ADN polimerasa de inicio en caliente, con tampón optimizado, que puede reducir en gran medida la amplificación no específica; al mismo tiempo, el colorante de referencia ROX especial hace que la mezcla maestra sea adecuada para todos los instrumentos de qPCR, y no es necesario ajustar ROX en diferentes instrumentos. La amplificación se puede realizar agregando cebadores y plantillas al preparar el sistema de reacción.
2.3.1 Compatible con instrumentos qPCR de todas las plataformas
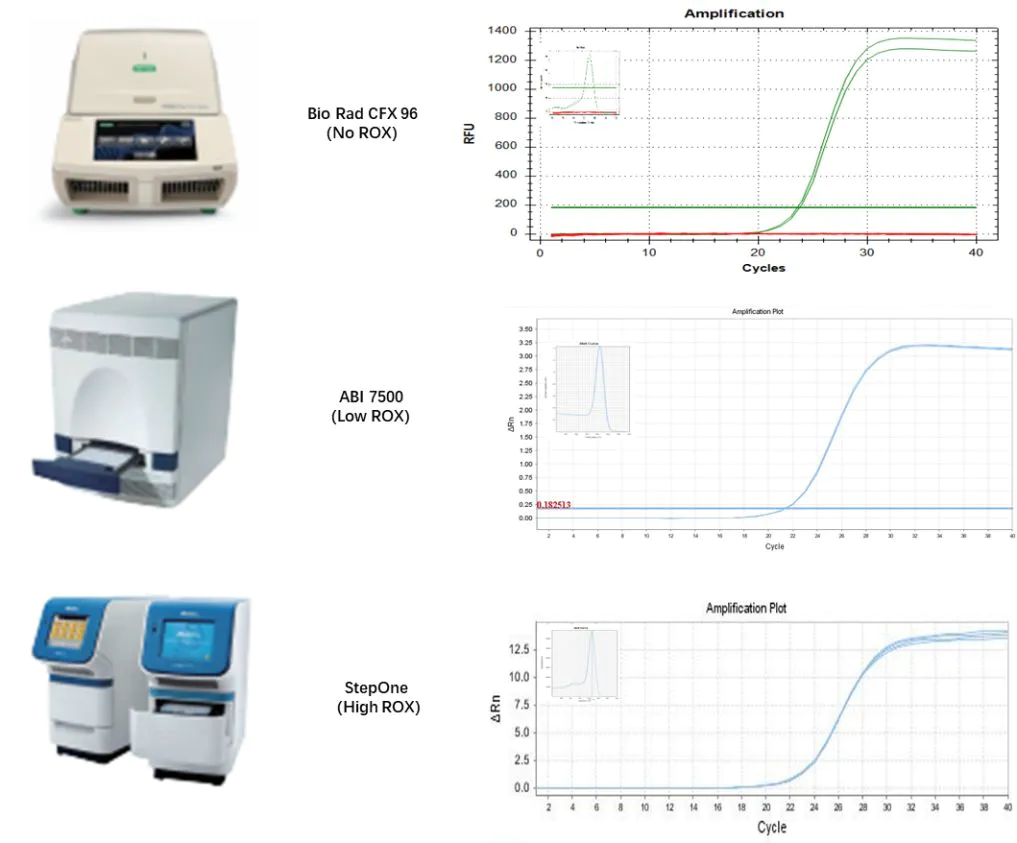
Figura 17. Resultados compatibles con diferentes instrumentos de qPCR
Utilizando ARN total de células de hígado de ratón como plantilla, se realizó la transcripción inversa con Hifair™ III 1st Strand
Se utilizó el kit de síntesis de ADNc (digestor de ADNg plus) (Cat# 11139ES), y el ADNc obtenido se diluyó 40 veces y se utilizó Hieff™ miRNA Universal qPCR SYBR Master Mix (Cat# 11170ES) para amplificar el gen humano mmu-miR-125a, respectivamente.
2.3.2 Alta sensibilidad, hasta 10 pg

Figura 18. Detección de sensibilidad
Utilizando ARN total de células de hígado de ratón como plantilla, se realizó una transcripción inversa con el kit de síntesis de ADNc de primera hebra Hifair™ III (gDNA digester plus) (Cat# 11139ES), y el ADNc obtenido se diluyó en serie 10 veces (rango 10 pg/μL). -0,1 μg/μL), para amplificar los genes mmu-miR-125a, mmu-miR-132 y mmu-miR-351 de origen murino.
2.3.3 Alta especificidad, capaz de distinguir diferencias de bases individuales entre miRNAs de la misma familia
Figura 19. Distinguir diferencias de bases individuales entre los miRNA
La familia hsa-let-7 humana tiene múltiples miRNA, con pocas bases que difieren entre sí, o incluso con una sola base de diferencia. Estos miRNA se amplificaron por separado con diferentes cebadores utilizando Hieff™ miRNA Universal qPCR SYBR Master Mix (Cat# 11170ES), y la tasa de coincidencia se calculó utilizando la fórmula 2^-Δct x 100%.
2.3.4 Buena repetibilidad

Figura 20. Detección de repetibilidad
Utilizando el ARN total de células humanas 293T como plantilla para la transcripción inversa, la transcripción inversa se realizó con el kit de síntesis de ADNc de primera hebra Hifair™ III (digestor de ADNc plus) (n.° de cat. 11139ES), y el ADNc obtenido se diluyó 50 veces y se utilizó Hieff™ miRNA Universal qPCR SYBR Master Mix (n.° de cat. 11170ES) para amplificar el gen hsa-let-7a humano en un experimento de réplica de 90 pocillos.
2.3.5 Buena estabilidad

Figura 21.Análisis de estabilidad de 11170ES a diferentes temperaturas durante 14 días
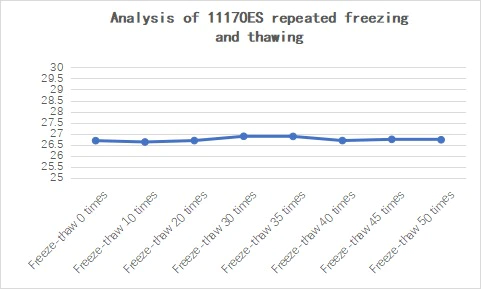
Figura 22. Análisis de la congelación y descongelación repetidas de 11170ES

Figura 23. Propiedades de análisis estables del sistema de reacción cuantitativa a diferentes temperaturas durante 24 h.
El ARN total de células hepáticas de ratón se utilizó como plantilla para la transcripción inversa con el kit de síntesis de ADNc de primera hebra Hifair™ III (digestor de ADNc plus) (n.° de cat. 11139ES). El ADNc obtenido se diluyó 100 veces para amplificar el gen murino mmu- miR-132.
3. Lista de productos relacionados
Yeasen Biotechnology (Shanghai) Co., Ltd., fundada en 2014, es una empresa de alta tecnología dedicada a la investigación y desarrollo y producción de materias primas enzimáticas y anticuerpos antigénicos. Sus productos incluyen enzimas, proteínas y anticuerpos de diagnóstico molecular utilizados en productos farmacéuticos, pruebas de seguridad alimentaria, crianza, justicia y otras industrias. Estamos comprometidos a proporcionar a los clientes en el campo de las ciencias biológicas productos y servicios de alta calidad. Los productos relacionados proporcionados por Yeasen son los siguientes:
Tabla 2. Lista de productos relacionados
| Tipo de método | Procedimiento experimental | Nombre del producto | Código del producto |
| Cola | Transcripción inversa | 11148ES | |
| PCR cuantitativa | 11171ES | ||
| Tallo-bucle | Transcripción inversa | Hifair™ Kit de síntesis de ADNc de primera cadena (digestor de ADNg plus) (Consulta) | 11139ES |
| PCR cuantitativa | Hieff™ Mezcla maestra universal qPCR SYBR de miRNA (Consulta) | 11170ES |