¿Cómo elegir tus adaptadores NGS?
El adaptador NGS, una parte esencial de la biblioteca de secuenciación de próxima generación, desempeña un papel en la conexión del fragmento de ADN analizado y la celda de flujo (chip de secuenciación). La eficiencia de la unión es un factor importante para determinar la calidad y el rendimiento de la biblioteca. Entonces, ¿qué es un adaptador NGS? ¿Cuáles son los tipos comunes de adaptadores NGS? ¿Y cómo elegir los adaptadores NGS adecuados para sus plataformas de secuenciación?
1. ¿Qué es el adaptador NGS?
2. ¿Qué factores debes tener en cuenta a la hora de elegir el índice NGS?
3. ¿Cuáles son los tipos comunes de índices?
4. ¿Cuáles son los tipos comunes de adaptadores NGS?
- Adaptador UMI
- Adaptadores completos
- Adaptadores incompletos
- Adaptadores Tn5
5. ¿Cómo elegir los adaptadores NGS adecuados para sus plataformas de secuenciación?
6. Respecto a la lectura
1. ¿Qué es el adaptador NGS?
El adaptador NGS, una serie de adaptadores en secuenciación, es una secuencia corta de nucleótidos con una secuencia conocida. Se liga a ambos extremos del fragmento de ácido nucleico objetivo. Durante la secuenciación, comienza a secuenciarse hibridándose con la secuencia conocida en la celda de flujo para combinar la biblioteca con el chip. Entonces, ¿cuál es la estructura del adaptador NGS?
Tomando la plataforma Illumina como ejemplo, un adaptador NGS se puede dividir en tres partes:
P5 y P7: Secuencia combinada con los extremos P5 y P7 en la celda de flujo para fijar la biblioteca en el chip de secuenciación, facilitando la reacción en grupo a través de Bridge-PCR.
Rd1 SP y Rd2 SP (cebador de secuenciación Read1/Read2): regiones de unión de los cebadores de secuenciación, que indican la posición donde comienza a leerse la secuencia.
Índice (también conocido como código de barras): secuencia sintética conocida que se utiliza para distinguir diferentes muestras en la secuenciación de la biblioteca mixta.
Higo. 1 biblioteca de índice único de la plataforma Illumina

Higo. 2 Biblioteca de índices de un solo extremo de la plataforma MGI
Con el aumento del rendimiento de la secuenciación, se pueden secuenciar varias muestras al mismo tiempo. Por lo tanto, es especialmente importante saber distinguir las distintas muestras. Como se mencionó anteriormente, las secuencias de índice del adaptador NGS se utilizan para diferenciar varias muestras en la secuenciación de próxima generación (NGS). ¿Qué factores debe tener en cuenta al elegir el índice? Continúe leyendo...
2. ¿Qué factores debes tener en cuenta al elegir un índice?
El índice tiene generalmente una longitud de 6 nt-18 nt y se divide en un índice simple y un índice doble según la cantidad de índices. El índice doble se ubica en ambos extremos del fragmento que se va a analizar. Se debe tener en cuenta el equilibrio de bases y el equilibrio de fluorescencia al seleccionar una combinación de índices.
El balance base se refiere al balance entre varios índices, en lugar del balance base en un solo índice. Debe considerarse tanto desde el punto de vista de los tipos de base como de la distribución de la base. El principio de combinación es que las cuatro bases A/T/C/G en el mismo grupo de índices deben incluirse, y la proporción de estas cuatro bases es cercana, representando aproximadamente el 25% respectivamente.
El equilibrio de la señal de fluorescencia se refiere a la elección para garantizar el equilibrio de las señales fluorescentes cuando no se puede garantizar el equilibrio de la base. En el secuenciador de 4 canales de la plataforma Illumina, dG/dT se etiqueta con fluorescencia verde y dC/dA se etiqueta con fluorescencia roja. Durante la secuenciación, deben existir señales de fluorescencia verde y roja en cada ciclo para garantizar una secuenciación exitosa. Por lo tanto, al seleccionar el índice se debe tener en cuenta el equilibrio entre la señal verde y la señal roja.
3. ¿Cuáles son los tipos comunes de índices?
Los índices duales comunes suelen incluir el índice dual único (UDI), el código de barras dual único (UDB) y el índice dual combinado (CDI), que reducen significativamente los saltos de índice y las asignaciones incorrectas.
UDI y UDB: los índices en ambos extremos corresponden uno a uno, están diseñados en grupos y se pueden verificar de forma cruzada en ambos extremos;
Kit de cebadores Stubby UDI para Illumina suministrado por Yeasen (Cat. n.° 12404ES/12405ES)>>
CDI: los índices en ambos extremos se pueden combinar según ciertos requisitos para formar una biblioteca de índices de doble extremo;
Un cebador CDI 384 para Illumina, Set1-Set2 suministrado por Yeasen (Cat. n.º12412ES/12413ES)>>
Con el fin de mejorar el rendimiento y la eficiencia de la amplificación y reducir el costo de secuenciación, Illumina introdujo la tecnología de agrupamiento de celdas de flujo de matriz (PFCT) y de amplificación exclusiva (ExAmp) para Novaseq y otros secuenciadores de alto rendimiento, pero inadvertidamente amplificó el fenómeno de desajuste de etiquetas de muestra y el salto de índice.

Fig. 3 Los diferentes modelos de instrumentos de Illumina adoptan una celda de flujo sin patrón o una celda de flujo con patrón
Para compensar el problema de salto de índices que se destaca en las plataformas de secuenciación como HiSeq3000/4000, HiSeq X Series y NovaSeq, Illumina propuso la estrategia de colocar el índice en ambos extremos de la biblioteca, lo que puede realizar una verificación bilateral y eliminar los adaptadores no coincidentes. Al utilizar los índices únicos en ambos extremos, la tasa de asignación de errores de índice se reducirá al 0,01 %. En comparación con el método de combinación de grupos de permutación de índices convencional anterior, el salto de índices se reducirá en dos órdenes de magnitud.
En la construcción de una biblioteca sin PCR, se dispone de un adaptador de índice de extremo único. La falta de coincidencia de etiquetas se debe principalmente a errores de secuenciación. En general, la tasa de falta de coincidencia de etiquetas es baja (promedio de 0,0004 %, hasta 0,001 %). Sin embargo, en la construcción de una biblioteca de captura dirigida, el problema de diafonía se amplifica porque varios pasos darán lugar a una falta de coincidencia de etiquetas y, por lo general, se utilizan los adaptadores UDI/UDB/CDI.
4.¿Cuáles son los tipos comunes de adaptadores NGS?
Con el desarrollo de la tecnología de secuenciación, existen cada vez más tipos de adaptadores, como adaptadores de índice único/doble índice (como se mencionó en la sección 3), adaptadores UMI, adaptadores de transposasa, adaptadores completos/incompletos, etc., que son adecuados para una variedad de escenarios de aplicación. Esta parte clasifica sistemáticamente estos adaptadores para brindarle las bases para la selección de adaptadores.
4.1 Adaptador UMI
El adaptador de identificador molecular único (UMI) es una herramienta de vanguardia para la detección de mutaciones de baja frecuencia y la cuantificación absoluta. El UMI es una secuencia sintética aleatoria con una secuencia conocida. Puede diseñarse como una cadena de nucleótidos completamente aleatoria, una cadena de nucleótidos parcialmente degenerada o una cadena de nucleótidos fija.La longitud suele ser de 10 nt (UMI de un solo extremo) o de 5-8 nt (UMI de doble extremo). Su función es congelar el estado de los fragmentos de ADN antes de la amplificación, y cada molécula de ADN corresponde a un UMI. Por lo tanto, durante el análisis de la bioinformática, puede distinguir las plantillas de ADN de diferentes fuentes, distinguir cuáles son mutaciones falsas positivas causadas por errores aleatorios en el proceso de amplificación y secuenciación por PCR, y cuáles son realmente portadoras de los pacientes, para así filtrar el ruido de fondo, realizar la detección precisa de mutaciones de baja frecuencia y de frecuencia extremadamente baja, y llevar a cabo la cuantificación absoluta de diferentes moléculas de ADN. Se utiliza ampliamente en la detección de mutaciones de baja frecuencia, especialmente en el campo de la investigación tumoral.

Fig. 4 Diagrama esquemático del adaptador UMI Estructura de la plataforma Illumina
4.2 Adaptadores completos
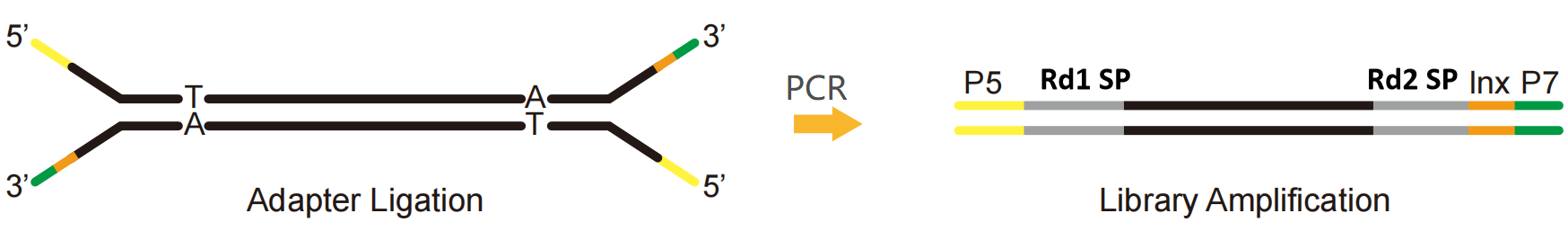
Los adaptadores completos, un producto necesario para una biblioteca sin PCR, contienen todas las secuencias requeridas para las secuenciaciones, como P5, P7, RdS1 y RdS2 en la plataforma Illumina, también secuencias de índice y secuencias UMI de acuerdo con los requisitos para la secuenciación. Con los adaptadores completos, se puede secuenciar directamente sin introducir otros adaptadores a través de PCR. Por lo tanto, los adaptadores completos se pueden utilizar para construir una biblioteca sin PCR. Las bibliotecas sin PCR pueden reducir el sesgo de amplificación de PCR, la tasa de error y la duplicación de secuencias, lo que aumenta la cobertura de algunas regiones de alto GC o alto AT que se utilizan ampliamente en la investigación del genoma de la población.
Un producto adaptador completo para la plataforma Illumina suministrado por Yeasen (Cat. n.° 13519ES/13520ES)>>
Un producto adaptador completo para la plataforma MGI suministrado por Yeasen (Cat. n.° 13360ES/13361ES)>>
Fig. 5 Diagrama completo del adaptador
4.3 Adaptadores incompletos
Los adaptadores incompletos necesitan introducir otras secuencias mediante PCR después de la ligadura del adaptador para formar un adaptador completo. Se caracterizan por una alta eficiencia de conexión y una alta tasa de biblioteca efectiva. El proceso de PCR es un efecto de enriquecimiento para la biblioteca completa para garantizar la concentración de la biblioteca efectiva, y también puede introducir índices de doble extremo y secuencias UMI.
4.4 Adaptadores Tn5
Los adaptadores Tn5 conectan parte de la secuencia adaptadora a ambos extremos de los fragmentos de ADN a través de la actividad de la endonucleasa de restricción de Tn5. Hacen que la fragmentación y la ligadura del adaptador se realicen simultáneamente para ahorrar tiempo y muestras. Finalmente, el resto de la secuencia de enlace, el índice, el UMI y otras secuencias se introducen mediante PCR para formar una biblioteca completa. Puede usarse para construir una biblioteca Cut&tag.
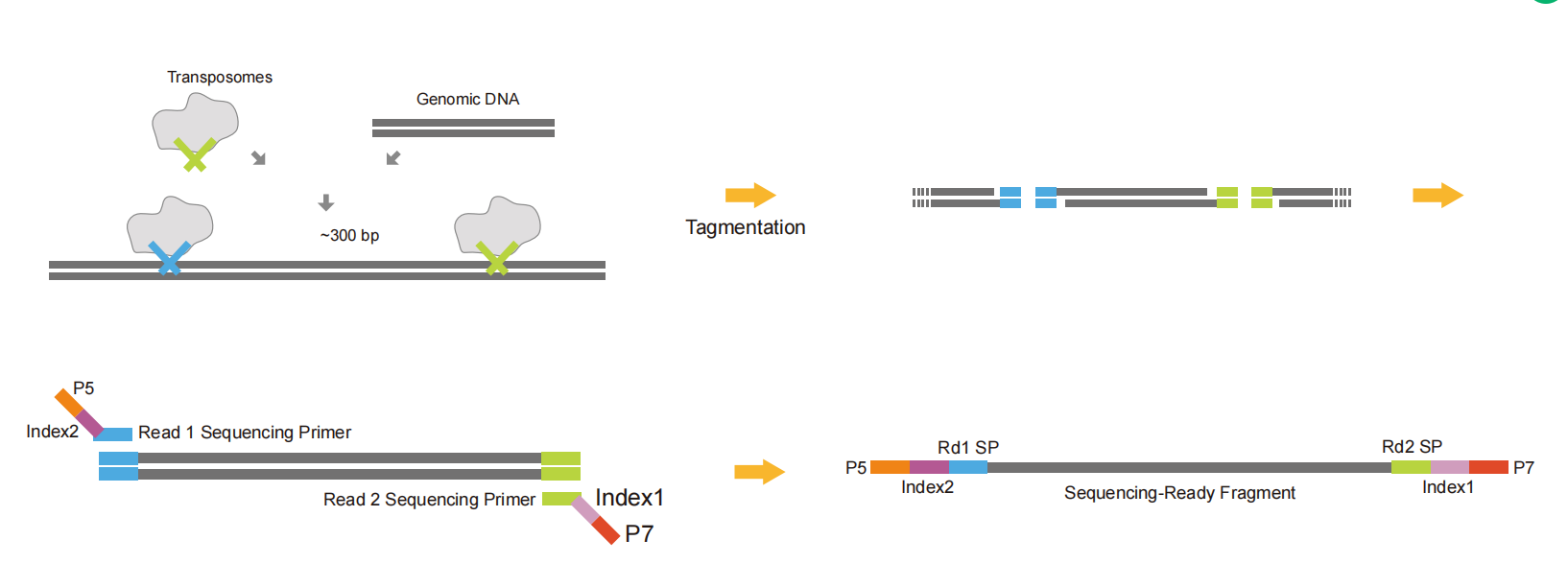
Fig.6 Diagrama esquemático de la construcción de la biblioteca del adaptador Tn5
5. ¿Cómo elegir los adaptadores NGS adecuados para sus plataformas de secuenciación?
Actualmente, existen dos plataformas de secuenciación convencionales, Illumina y MGI. Yeasen, un proveedor de soluciones completas para secuenciación de nueva generación, ha desarrollado varios adaptadores de secuenciación de nueva generación adecuados para las plataformas Illumina o MGI.
En términos de la plataforma Illumina, los adaptadores Illumina NGS suministrados por Yeasen contienen tres tipos, incluidos UDI, CDI y un índice único. En términos de la plataforma MGI, los adaptadores MGI NGS ofrecidos por Yeasen tienen dos tipos, que incluyen Dual UMI-UDB e Índice Único.Hemos enumerado la información del producto en la siguiente tabla, incluidos los tipos de adaptadores, los tamaños disponibles y la concentración de adaptador y cebador, respectivamente.
Los adaptadores NGS completos y UDI no necesitan preocuparse por problemas de acoplamiento, adecuados para clientes que desean un uso fácil; los adaptadores NGS CDI tienen menos tubos y un tamaño pequeño, lo que es adecuado para clientes que desean almacenar y transportar fácilmente. PCR-free requiere el uso de adaptadores NGS completos.
Para Illumina
| En tubo | Alto NGS® DPreparación para la biblioteca de NA 384 CDI Primer para Illumina, juego 1 (índice 8 x 12, 96) | 12412ES |
| Alto NGS® DPreparación para la biblioteca de NA 384 CDI Primer para Illumina, juego 2 (índice 8 x 12, 96) | 12413ES | |
| Cebador Hieff NGS® RNA Lib Prep 384 CDI para Illumina, juego 1 (índice 96) | 12414ES | |
| Cebador Hieff NGS® RNA Lib Prep 384 CDI para Illumina, juego 1 (índice 96) | 12415ES | |
| En placa | Kit de cebadores Hieff NGS® Stubby UDI para Illumina (índice 1-384), juego 1-4 | 12407ES |
| Kit de cebadores UDI Hieff NGS® Stubby para Illumina Conjunto 1(Placa de 96 pocillos, índice 1-96) Conjunto 1 | 12327ES | |
| Kit de cebadores UDI Hieff NGS® Stubby para Illumina Conjunto 2(Placa de 96 pocillos, índice 97-192) Conjunto 2 | 12328ES | |
| Kit de cebadores UDI Hieff NGS® Stubby para Illumina Conjunto 3(Placa de 96 pocillos, índice 193-288) Conjunto 3 | 12329ES | |
| Kit de cebadores UDI Hieff NGS® Stubby para Illumina Conjunto 4(Placa de 96 pocillos, índice 289-384) Conjunto 4 | 12330ES |
Para MGI
| Hieff NGS™ Kit adaptador dual UMI UDB para MGI, Set1/Set2 | 96 tipos de índice | |
| Alto NGS™ Kit adaptador completo para MGI, Conjunto 1/Conjunto 2/Conjunto 3 (Consultar) | 13360ES | 8 tipos de índice, 41-48 |
| 13361ES | 16 tipos de índice, 57-72 | |
| 13362ES | 96 tipos de índice, 1-96 |
Respecto a la lectura
Enzimas clave implicadas en la construcción de bibliotecas NGS
Sobre la tecnología relacionada con NGS, ¿cuánto sabes?
Diversos tipos de perlas magnéticas en NGS: perlas magnéticas de ADN\ARN\ARNm
Cuantificación de la biblioteca NGS: ¿Qubit rápido y preciso o qPCR preciso? ¡Todo necesario!
