Que faire lorsque les résultats du qPCR sont étranges ?
Les expériences de qPCR peuvent sembler faciles et parfois difficiles. Une fois la conception de l'amorce terminée, les échantillons de qPCR sont ajoutés étape par étape comme la PCR, et l'expérience peut être réalisée sur la machine. Il existe plusieurs problèmes courants dans les expériences de qPCR par la méthode du colorant et compte tenu des causes et des solutions possibles, nous espérons que vous pourrez les utiliser pour résoudre les problèmes des expériences de qPCR.
1. Qu'est-ce que la qPCR ?
2. Courbe d'amplification qPCR anormale
3. Courbe de fusion qPCR anormale
4. Informations de commande
5. Produits connexes
6. Concernant la lecture
1. Qu'est-ce que la qPCR ?
Depuis l'invention de la technique de réaction en chaîne par polymérase (PCR), la PCR est probablement la technique la plus largement utilisée en biologie moléculaire en raison de sa simplicité, de son faible coût, de sa fiabilité, de sa rapidité et de sa grande sensibilité. La qPCR est une technologie développée par la technologie PCR. Au cours du processus d'amplification de l'ADN, la méthode de détection de la quantité totale de produits après chaque cycle de PCR avec des colorants fluorescents a non seulement la rapidité et la sensibilité de la PCR, mais aussi une plus grande spécificité et une surveillance élevée en temps réel, une quantification répétable et précise, ainsi que d'autres avantages. La qPCR est une technologie qui utilise un dispositif d'amplification PCR quantitative en temps réel, à savoir un instrument qPCR, pour surveiller les produits d'amplification d'acide nucléique en temps réel pendant le processus de PCR. La qPCR réalise l'analyse quantitative du modèle de départ grâce à l'analyse de la valeur Ct et de la courbe standard. En 1992, le japonais Higuchi a proposé pour la première fois la « technologie PCR quantitative fluorescente en temps réel ». En 1996, la société American Biological Company a lancé le premier instrument de PCR quantitative fluorescent au monde, qui est composé d'un système de cycle thermique d'amplification PCR, d'un système optique de détection de fluorescence et d'un logiciel informatique et d'application. Il peut surveiller les produits d'amplification d'acide nucléique en temps réel grâce à des colorants fluorescents ou des sondes fluorescentes. , grâce à la relation de fonction mathématique, combinée au logiciel d'analyse des résultats, le calcul de la quantité initiale de modèle de l'échantillon à tester est réalisé. Par conséquent, la technologie de PCR quantitative fluorescente en temps réel a été largement utilisée. Les méthodes de marquage fluorescent pour la qPCR sont divisées en méthode de mosaïque de colorant fluorescent basée sur la méthode du colorant SYBR Green I, méthode de sonde fluorescente basée sur la méthode de sonde Taqman (Cycling Probe, Molecular Bracon, etc.), méthode d'amorce de colorant extincteur.
Les expériences de qPCR peuvent sembler faciles et parfois difficiles. Une fois la conception de l'amorce terminée, les échantillons de qPCR sont ajoutés étape par étape comme la PCR, et l'expérience peut être réalisée sur la machine. Cependant, de nombreux petits détails nécessitent une attention particulière, comme l'absence de liquide accroché à la paroi du tuyau, l'absence de bulles dans le tuyau et la nécessité de minimiser le contact direct entre la paroi du tuyau, le couvercle du tuyau, etc., afin de réduire les erreurs expérimentales ou les résultats expérimentaux étranges. Xiaoyi a compilé plusieurs problèmes courants dans les expériences de qPCR par la méthode du colorant et a donné les causes et solutions possibles, que nous espérons que vous pourrez utiliser pour résoudre les problèmes dans les expériences de qPCR.
2. Courbe d'amplification qPCR anormale
2.1 Anomalies dans la phase de base du tracé logarithmique de la courbe d'amplification
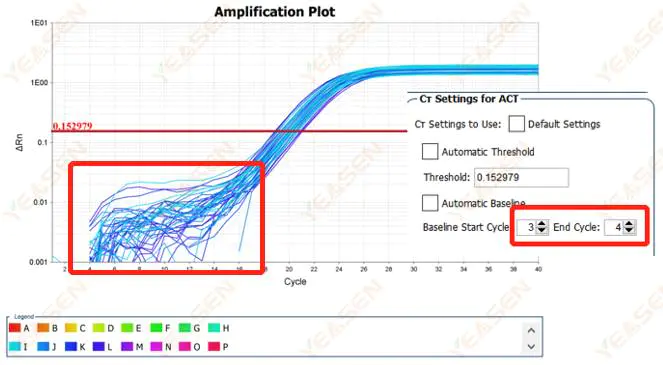
Figure 1. Diagramme d'amplification
Causes possibles :
Paramétrage de base incorrect.
Solutions:
Il est recommandé d'augmenter la valeur du point final de la ligne de base.L'exemple de diagramme de réglage est le suivant.

Figure 2. Diagramme d'amplification
2.2 Segmentation de la courbe d'amplification
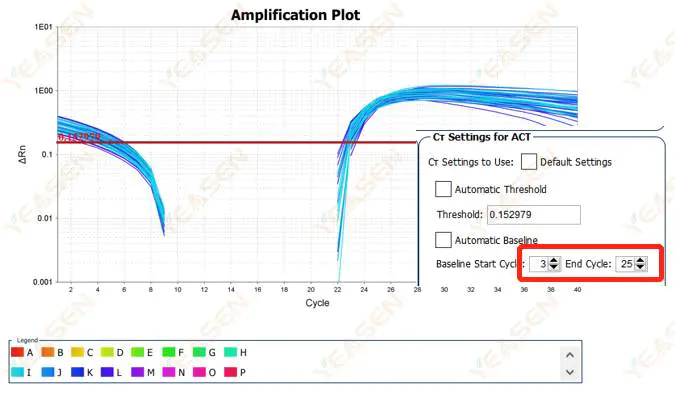
Figure 3. Diagramme d'amplification
Causes possibles :
La ligne de base est définie trop haut.
Solutions:
Il est recommandé de réduire la valeur du point final de la ligne de base. Le diagramme d'exemple d'ajustement est le suivant.
2.3 Graphique de linéarité de la courbe d'amplification non lisse
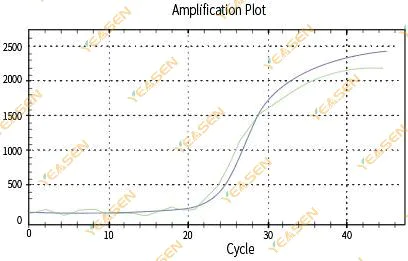
Figure 4. Diagramme d'amplification
Causes possibles :
1) Le tube de réaction PCR n’était pas hermétiquement fermé et la solution de réaction a fui.
2) Solution de réaction PCR avec paroi suspendue.
3) L'instrument n'est pas étalonné (y compris l'étalonnage automatique ou l'étalonnage ROX).
4) Le système comporte de nombreux inhibiteurs, ce qui entraîne une fluorescence instable.
5) Une utilisation excessive de l’instrument entraîne une collecte de fluorescence instable.
Solutions:
1) Appuyez fermement sur le couvercle du tuyau.
2) Mélangez soigneusement le réactif et placez-le soigneusement dans l'instrument quantitatif après centrifugation complète.
3) Calibrer l'instrument.
4) Améliorer la pureté de l’ARN et sélectionner les réactifs de transcription inverse appropriés.
2.4 Graphique linéaire à courbe amplifiée en zigzag sur un plateau
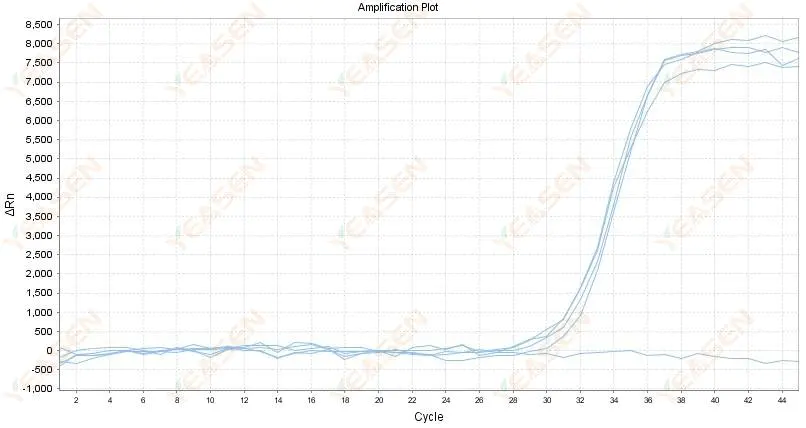
Figure 5. Diagramme d'amplification
Causes possibles :
1) La pureté de l’ARN est faible et il contient de nombreuses impuretés.
2) L'instrument est utilisé trop longtemps.
Solutions:
1) Réextraire l'ARN de haute qualité.
2) Diluez le modèle d’ARN pour réduire la concentration d’impuretés.
3) Calibrer l'instrument.
2.5 Le tracé linéaire de la courbe d'amplification ne parvient pas à atteindre un plateau
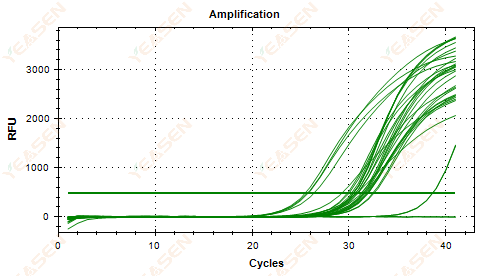
Figure 6. Diagramme d'amplification
Causes possibles :
1) Faible concentration en gabarit (valeur Ct autour de 35).
2) Trop peu de cycles d'amplification.
3) Faible efficacité de l'amplification du réactif (faible Ct, mais également incapacité à atteindre le plateau).
Solutions:
1) Augmenter la concentration du modèle.
2) Augmenter le nombre de cycles.
3) Augmentation du Mg2+ concentration.
2.6 Courbe d'amplification tracé linéaire plateau affaissement
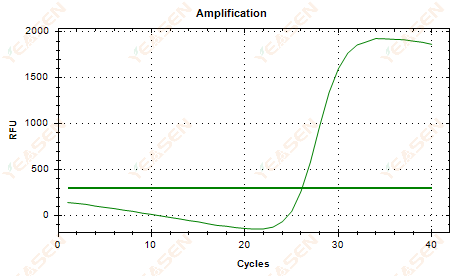
Figure 7. Diagramme d'amplification
Causes possibles :
1) Présence de dégradation (dégradation du produit d'amplification, dégradation SYBR).
2) Le bouchon du tube n’est pas correctement recouvert et le réactif est volatilisé.
3) La concentration d'ADNc est trop élevée (si la valeur Ct est trop petite, le seuil de fluorescence est élevé et l'affaissement est plus sévère).
4) Des bulles d'air dans le tube, puis elles disparaissent.
Solutions:
1) Améliorer la pureté du système.
2) Réduire la quantité d’ADNc (diluer le modèle).
3) Diminuer la valeur du point final de la ligne de base.
2.7 Grande valeur de Ct dans la courbe d'amplification
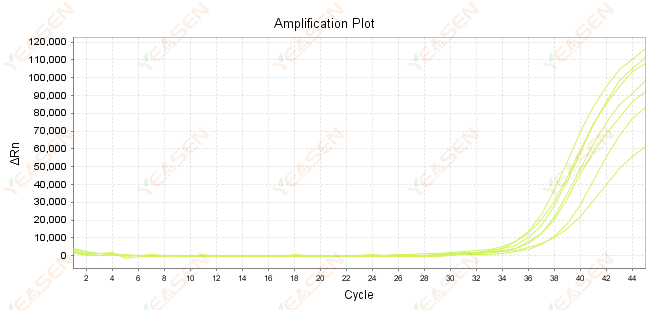
Figure 8. Diagramme d'amplification
Causes possibles :
1) Faible quantité de modèles.
2) Faible efficacité d'amplification.
3) Le fragment PCR est trop long.
4) Présence d'inhibiteurs dans le système réactionnel.
Solutions:
1) Réduisez les temps de dilution ou augmentez la quantité de modèle afin que la valeur Ct se situe autant que possible entre 15 et 30.
2) Optimisez les conditions de réaction, essayez une procédure d’amplification en trois étapes ou reconcevez les amorces.
3) La longueur du produit PCR est conçue pour être comprise entre 100 et 150 pb, il n'est pas recommandé de dépasser 300 pb.
4) Reproduction de modèles de pureté supérieure.
2.8 Faible répétabilité des courbes d'amplification
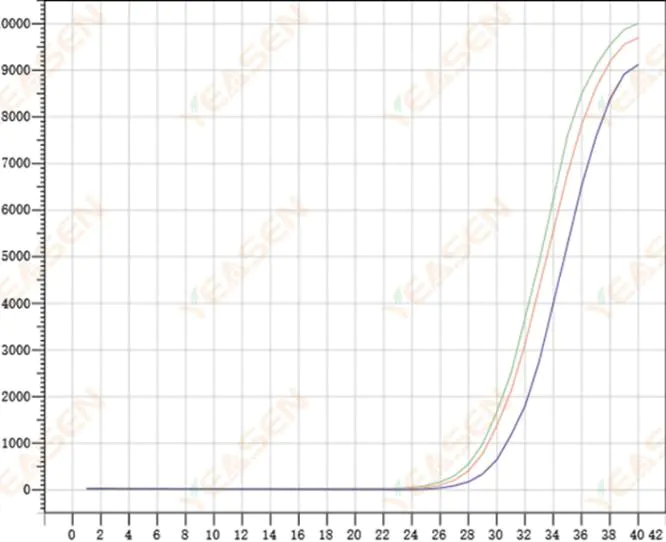
Figure 9. Diagramme d'amplification
Causes possibles :
1) Grosses erreurs de pics.
2) Les réactifs et les systèmes ne sont pas bien mélangés.
3) Faible nombre de copies d'ADNc.
4) Aucun étalonnage Rox n'a été utilisé.
Solutions:
1) Calibrage des pipettes.
2) Mélange minutieux du système réactionnel.
3) Une faible concentration de modèle, une faible répétabilité, 4 à 6 trous composés et 1 à 2 écarts de valeurs plus importantes peuvent être éliminés de manière appropriée.
4) Il est préférable d'utiliser l'étalonnage Rox. Si le réactif utilisé ne contient pas de Rox, le colorant de référence doit être sélectionné comme Aucun.
2.9 Courbes d'amplification aléatoires
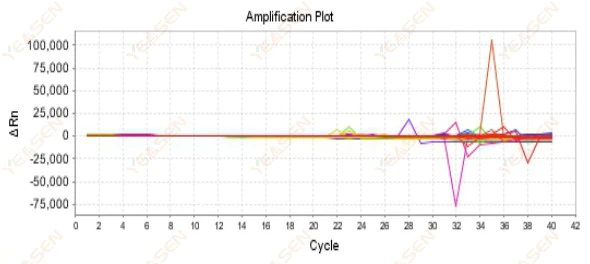
Figure 10. Diagramme d'amplification
Causes possibles :
L'inadéquation entre la concentration de Rox et le modèle.
Solutions:
Modifiez le paramètre de colorant de référence sur l'instrument de ROX à AUCUN et voyez si la courbe d'amplification revient à la normale.
2.10 Pic NTC
2.10.1 Ct>35, valeur Tm de la courbe de fusion <80℃
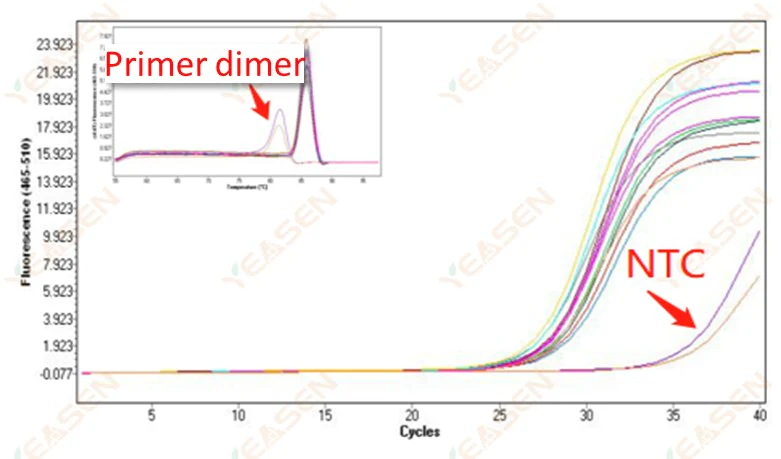
Figure 11. Diagramme d'amplification
Causes possibles :
Résultats de la dimérisation de l'amorce.
Solutions:
Amorces optimisées.
2.10.2 Valeur Ct < 35, la forme du pic de la courbe de fusion NTC et de la courbe de fusion des gènes se chevauchent
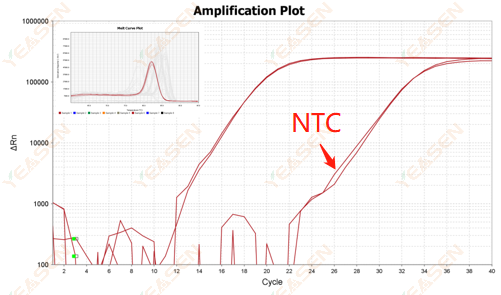
Figure 12. Diagramme d'amplification
Causes possibles :
Contamination du système réactionnel.
Solutions:
Vérifiez les sources de pollution une par une.
3. Courbe de fusion qPCR anormale
3.1 Courbe de fusion à pic unique mais pas nette
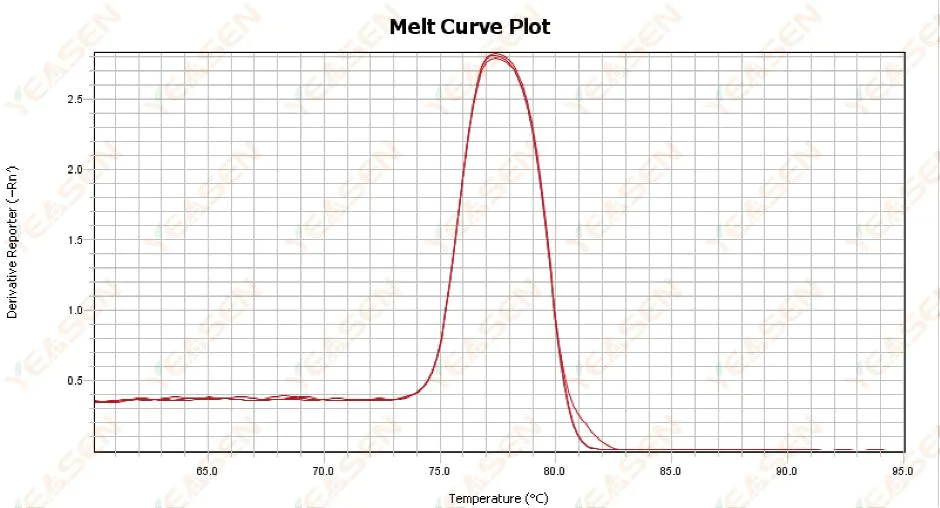
Figure 13. Courbe de fusion
Causes possibles :
1) Cela est lié à la composition du réactif et au modèle de l'instrument.
2) Présence d'amplification non spécifique de fragments de taille similaire.
Solutions:
1) La plage de température entre le début du pic et sa fin ne dépasse pas 7°C. Les résultats sont considérés comme utilisables, c'est-à-dire à pic unique.
2) Une électrophorèse sur agarose à haute concentration (par exemple 3 % d'agarose) a été réalisée pour faciliter la détermination.
3.2 La courbe de fusion est à double pic et le pic inférieur Tm est avant 80°C
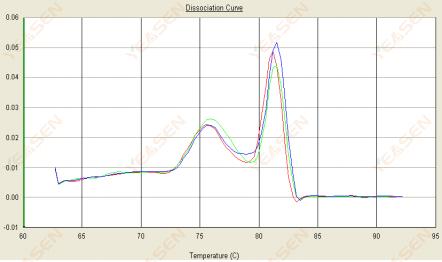
Figure 14. Courbe de dissociation
Causes possibles :
Présence de dimères d'amorces.
Solutions:
Augmentez la température de recuit, diminuez la concentration d’amorce ou reconcevez les amorces.
3.3 La courbe de fusion est à double pic et le pic Tm le plus bas se situe après 80°C
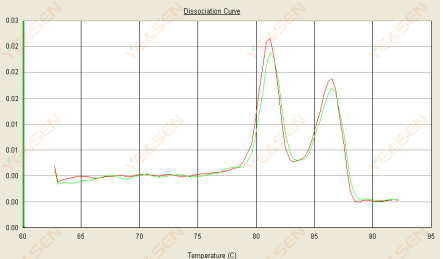
Figure 15. Courbe de dissociation
Causes possibles :
1) Une faible spécificité de l’amorce conduit à l’amplification de produits non spécifiques.
2) Contamination par l'ADNg.
Solutions:
1) Contrôle de la spécificité de l'amorce par explosion, reconception si mauvaise.
2) La confirmation par le contrôle négatif du NRC, le cas échéant, nécessite de préparer à nouveau le modèle.
3.4 Pics de courbe de fusion désordonnés
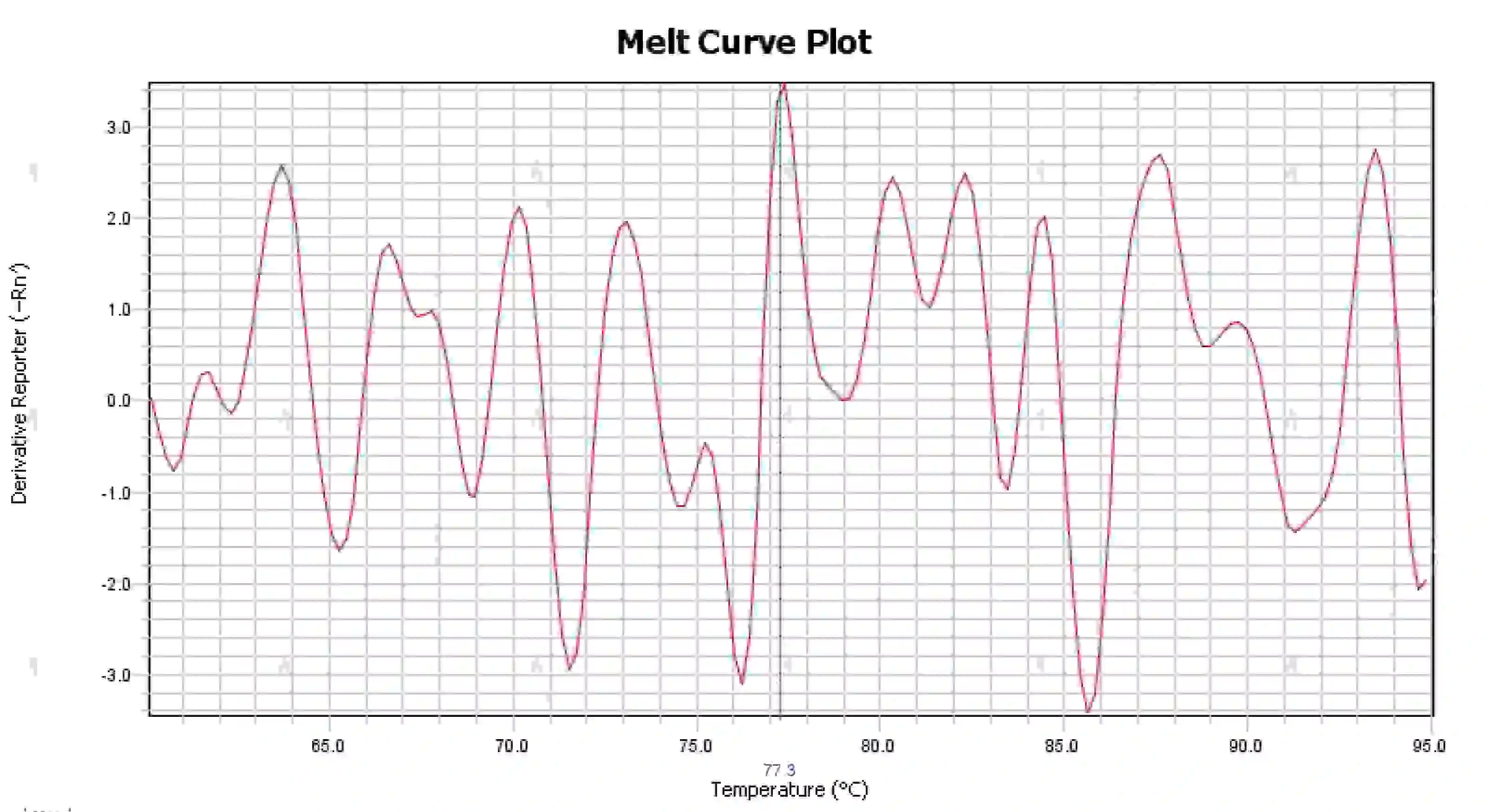
Figure 16. Courbe de fusion
Causes possibles :
1) Contamination du système réactionnel.
2) L’exposition des réactifs à une lumière vive ou à une température élevée entraîne une défaillance du réactif.
3) L'instrument n'a pas été étalonné depuis longtemps.
4) Inadéquation entre les consommables et les instruments.
Solutions:
1) En combinant les résultats NTC et NRC pour confirmer la contamination, il est recommandé d'exclure la contamination de l'eau, des amorces, des enzymes et de l'environnement un par un.
2) Il est recommandé de faire des expériences de comparaison avec de nouveaux réactifs.
3) Un entretien régulier de l'étalonnage des instruments est recommandé.
4) Confirmer les exigences de l’instrument correspondant pour les consommables.
3.5 Pics parasites à l'extrémité avant de la courbe de fusion
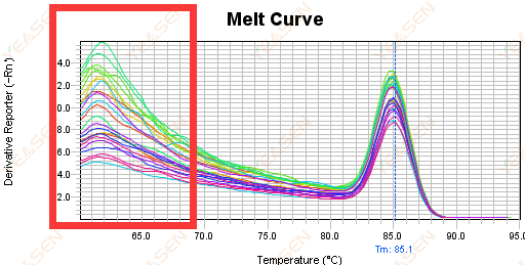
Figure 17. Courbe de fusion
Causes possibles :
La concentration de Rox ne correspond pas au modèle.
Solutions:
Il est recommandé d'annuler la correction Rox pour voir si la courbe de fusion est normale.
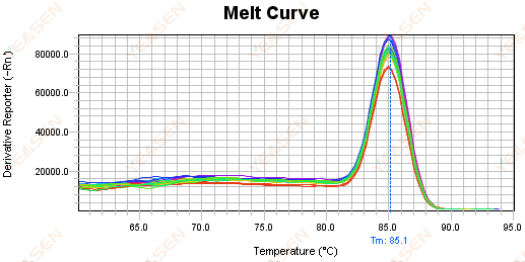
Figure 18. Courbe de fusion
4. Informations de commande
Tableau 1. Informations de commande
| Nom du produit | Code produit | Spécification |
| Mélange maître qPCR bleu universel Hieff Unicon™ (à base de colorant) | 11184ES03 | 1 mL |
| 11184ES08 | 5×1 mL | |
| 11184ES50 | 50×1 mL | |
| 11184ES60 | 100×1 mL |
5. Produits connexes
Les produits fournis par Yeasen sont les suivants.
Tableau 2. Produits connexes
| Positionnement du produit | Nom du produit | Code produit |
| Élimination de l'ADN génomique et transcription inverse en une seule étape | Digestion RT-gDNA en une étape Hifair™V SuperMix pour qPCR (Enquête) | 11142ES |
| Master Mix haute sensibilité (avec étape d'élimination de l'ADNg) | Hifair™ III SuperMix de synthèse d'ADNc 1er brin pour qPCR (digesteur d'ADNg plus) | 11141ES |
| Kit haute sensibilité (avec étape d'élimination de l'ADN génomique) | Kit de synthèse d'ADNc 1er brin Hifair™ III (digesteur d'ADNg plus) (Enquête) | 11139ES |
| Mélange maître quantitatif ordinaire (méthode de la sonde) | Mélange maître de sonde Hieff™ qPCR TaqMan (Enquête) | 11205ES |
6. Concernant la lecture
DNase I et leurs applications en biomédecine
Guide de sélection de la transcriptase inverse
