Comment choisir vos adaptateurs NGS ?
L'adaptateur NGS, un élément essentiel de la bibliothèque de séquençage de nouvelle génération, joue un rôle dans la connexion du fragment d'ADN testé et de la Flow cell (puce de séquençage). L'efficacité de la jonction est un facteur important pour déterminer la qualité et le rendement de la bibliothèque. Alors, qu'est-ce qu'un adaptateur NGS ? Quels sont les types courants d'adaptateurs NGS ? Et comment choisir les bons adaptateurs NGS pour vos plateformes de séquençage ?
1. Qu'est-ce que l'adaptateur NGS ?
2. Quels facteurs devez-vous prendre en compte lors du choix de l’indice NGS ?
3. Quels sont les types d’index courants ?
4. Quels sont les types courants d’adaptateurs NGS ?
- Adaptateur UMI
- Adaptateurs complets
- Adaptateurs incomplets
- Adaptateurs Tn5
5. Comment choisir les bons adaptateurs NGS pour vos plateformes de séquençage ?
6. Concernant la lecture
1. Qu'est-ce que l'adaptateur NGS ?
L'adaptateur NGS, une série d'adaptateurs dans le séquençage, est une courte séquence nucléotidique avec une séquence connue. Il est lié aux deux extrémités du fragment d'acide nucléique cible. Pendant le séquençage, il commence le séquençage en s'hybridant avec la séquence connue sur la Flow cell pour combiner la bibliothèque à la puce. Quelle est donc la structure de l'adaptateur NGS ?
En prenant la plateforme Illumina comme exemple, un adaptateur NGS peut être divisé en trois parties :
P5 et P7 : Séquence combinée avec les extrémités P5 et P7 sur la Flow cell pour fixer la bibliothèque sur la puce de séquençage, facilitant la réaction de cluster via Bridge-PCR.
Rd1 SP et Rd2 SP (amorce de séquençage Read1/Read2) : régions de liaison des amorces de séquençage, indiquant la position où la séquence commence à être lue.
Index (également appelé code-barres) : séquence synthétique connue utilisée pour distinguer différents échantillons dans le séquençage de la bibliothèque mixte.
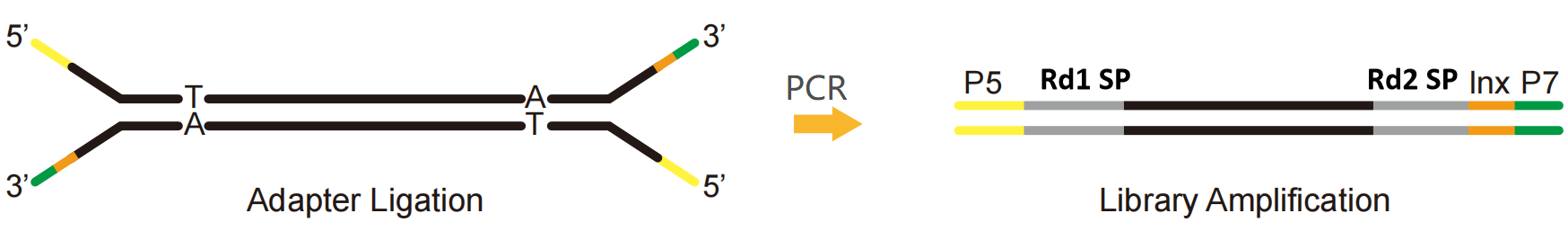
Figue. 1 bibliothèque d'index unique de la plateforme Illumina

Figue. 2 Bibliothèque d'index à extrémité unique de la plate-forme MGI
Avec l'augmentation du débit de séquençage, plusieurs échantillons peuvent être séquencés en même temps. Il est donc particulièrement important de savoir distinguer les différents échantillons. Comme mentionné précédemment, les séquences d'index de l'adaptateur NGS sont utilisées pour différencier les différents échantillons dans le séquençage de nouvelle génération (NGS). Quels facteurs devez-vous prendre en compte lors du choix de l'index ? Veuillez continuer à lire...
2. Quels facteurs devez-vous prendre en compte lors du choix d’un index ?
L'index est généralement d'une longueur de 6 à 18 nt et est divisé en un index simple et un index double en fonction du nombre d'index. L'index double est situé aux deux extrémités du fragment à tester. L'équilibre des bases et l'équilibre de fluorescence doivent être pris en compte lors de la sélection d'une combinaison d'indices.
Le solde de base fait référence au solde entre plusieurs indices, plutôt qu'au solde de base d'un seul indice. Il doit être pris en compte à la fois pour les types de base et la distribution de base. Le principe de combinaison est que les quatre bases A/T/C/G d'un même groupe d'indices doivent être incluses, et la proportion de ces quatre bases est proche, représentant respectivement environ 25 %.
L'équilibre du signal de fluorescence fait référence au choix de garantir l'équilibre des signaux fluorescents lorsque l'équilibre de base ne peut être garanti. Dans le séquenceur à 4 canaux de la plateforme Illumina, dG/dT est marqué par une fluorescence verte et dC/dA par une fluorescence rouge. Lors du séquençage, des signaux de fluorescence verts et rouges doivent exister dans chaque cycle pour garantir un séquençage réussi. Par conséquent, l’équilibre entre le signal vert et le signal rouge doit être pris en compte lors de la sélection de l’indice.
3. Quels sont les types d’index courants ?
Les index doubles courants incluent généralement l'index double unique (UDI), le code-barres double unique (UDB) et l'index double combiné (CDI), qui réduisent considérablement les sauts d'index et les erreurs d'affectation.
UDI&UDB : les index aux deux extrémités sont bijectifs, conçus en groupes et peuvent être vérifiés de manière croisée aux deux extrémités ;
Un kit d'amorces UDI Stubby pour Illumina fourni par Yeasen (Cat#12404ES/12405ES) >>
CDI : les index aux deux extrémités peuvent être combinés selon certaines exigences pour former une bibliothèque d'index à double extrémité ;
Une amorce 384 CDI pour Illumina, Set1-Set2 fournie par Yeasen (Cat#12412ES/12413ES)>>
Afin d'améliorer le débit et l'efficacité de l'amplification et de réduire le coût du séquençage, Illumina a introduit la cellule d'écoulement en réseau (PFCT) et la technologie de clustering d'amplification exclusive (ExAmp) pour Novaseq et d'autres séquenceurs à haut débit, mais a amplifié par inadvertance le phénomène de non-concordance des étiquettes d'échantillons et le saut d'index.

Fig. 3 Différents modèles d'instruments Illumina adoptent une cellule d'écoulement non structurée ou une cellule d'écoulement structurée
Afin de remédier au problème de cerclage d'index mis en évidence par les plates-formes de séquençage telles que HiSeq3000/4000, HiSeq X Series et NovaSeq, Illumina a proposé la stratégie consistant à placer l'index aux deux extrémités de la bibliothèque, ce qui permet d'effectuer une vérification bilatérale et d'éliminer les adaptateurs incompatibles. En utilisant les index uniques aux deux extrémités, le taux d'allocation d'erreur d'index sera réduit à 0,01 %. Par rapport à la méthode de combinaison de groupes de permutation d'index conventionnelle précédente, le saut d'index sera réduit de deux ordres de grandeur.
Lors de la construction d'une bibliothèque sans PCR, un adaptateur d'index à extrémité unique est disponible. La non-concordance des étiquettes est principalement due à des erreurs de séquençage. Dans l'ensemble, le taux de non-concordance des étiquettes est faible (en moyenne 0,0004 %, jusqu'à 0,001 %). Cependant, lors de la construction d'une bibliothèque de capture ciblée, le problème de diaphonie est amplifié car plusieurs étapes conduiront à une non-concordance des étiquettes et les adaptateurs UDI/UDB/CDI sont généralement utilisés.
4. Quels sont les types courants d’adaptateurs NGS ?
Avec le développement de la technologie de séquençage, il existe de plus en plus de types d'adaptateurs, tels que les adaptateurs à index simple/double index (comme mentionné dans la section 3), les adaptateurs UMI, les adaptateurs de transposase, les adaptateurs complets/incomplets, etc., qui conviennent à une variété de scénarios d'application. Cette partie trie systématiquement ces adaptateurs pour vous donner les bases de la sélection des adaptateurs.
4.1 Adaptateur UMI
L'adaptateur d'identifiant moléculaire unique (UMI) est un outil de pointe pour la détection de mutations à basse fréquence et la quantification absolue. L'UMI est une séquence synthétique aléatoire avec une séquence connue. Elle peut être conçue comme une chaîne nucléotidique complètement aléatoire, une chaîne nucléotidique partiellement dégénérée ou une chaîne nucléotidique fixe.La longueur est généralement de 10 nt (UMI à extrémité unique) ou de 5 à 8 nt (UMI à double extrémité). Sa fonction est de geler l'état des fragments d'ADN avant l'amplification, et chaque molécule d'ADN correspond à une UMI. Par conséquent, lors de l'analyse bioinformatique, il peut distinguer les modèles d'ADN de différentes sources, distinguer les mutations faussement positives causées par des erreurs aléatoires dans le processus d'amplification et de séquençage par PCR, et celles qui sont réellement portées par les patients, afin de filtrer le bruit de fond, réaliser la détection précise des mutations à basse et à très basse fréquence, et effectuer une quantification absolue de différentes molécules d'ADN. Il est largement utilisé dans la détection des mutations à basse fréquence, en particulier dans le domaine de la recherche sur les tumeurs.

Fig 4 Schéma de principe de l'adaptateur UMI structure de la plateforme Illumina
4.2 Adaptateurs complets
Les adaptateurs complets, un produit nécessaire pour une bibliothèque sans PCR, contiennent toutes les séquences requises pour les séquençages, telles que P5, P7, RdS1 et RdS2 dans la plateforme Illumina, ainsi que les séquences d'index et les séquences UMI en fonction des exigences de séquençage. Avec les adaptateurs complets, il peut être directement séquencé sans introduire d'autres adaptateurs par PCR. Ainsi, les adaptateurs complets peuvent être utilisés pour créer une bibliothèque sans PCR. Les bibliothèques sans PCR peuvent réduire le biais d'amplification PCR, le taux d'erreur et la duplication de séquence, augmentant la couverture de certaines régions à GC élevé ou à AT élevé qui sont largement utilisées dans la recherche sur le génome de la population.
Un produit adaptateur complet pour la plate-forme Illumina fourni par Yeasen (Cat#13519ES/13520ES)>>
Un produit adaptateur complet pour la plate-forme MGI fourni par Yeasen (Cat#13360ES/13361ES) >>
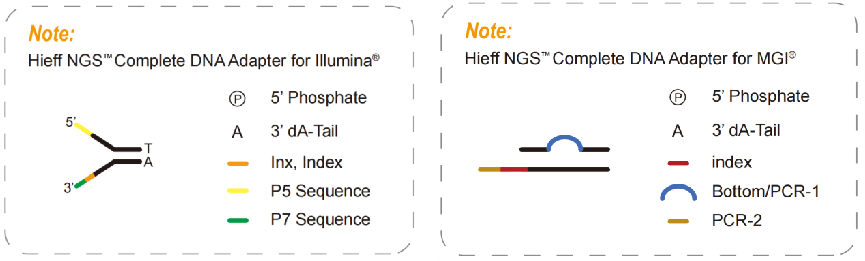
Fig. 5 Schéma complet de l'adaptateur
4.3 Adaptateurs incomplets
Les adaptateurs incomplets doivent introduire d'autres séquences par PCR après la ligature de l'adaptateur pour former un adaptateur complet. Ils se caractérisent par une efficacité de connexion élevée et un taux de bibliothèque efficace élevé. Le processus PCR est un effet d'enrichissement de la bibliothèque complète pour assurer la concentration de la bibliothèque efficace, et peut également introduire des index à double extrémité et des séquences UMI.
4.4 Adaptateurs Tn5
Les adaptateurs Tn5 connectent une partie de la séquence de l'adaptateur aux deux extrémités des fragments d'ADN grâce à l'activité d'endonucléase de restriction de Tn5. Ils permettent de réaliser simultanément la fragmentation et la ligature de l'adaptateur pour économiser du temps et des échantillons. Enfin, le reste de la séquence de liaison, l'index, l'UMI et d'autres séquences sont introduits par PCR pour former une bibliothèque complète. Elle peut être utilisée pour créer une bibliothèque Cut&tag.
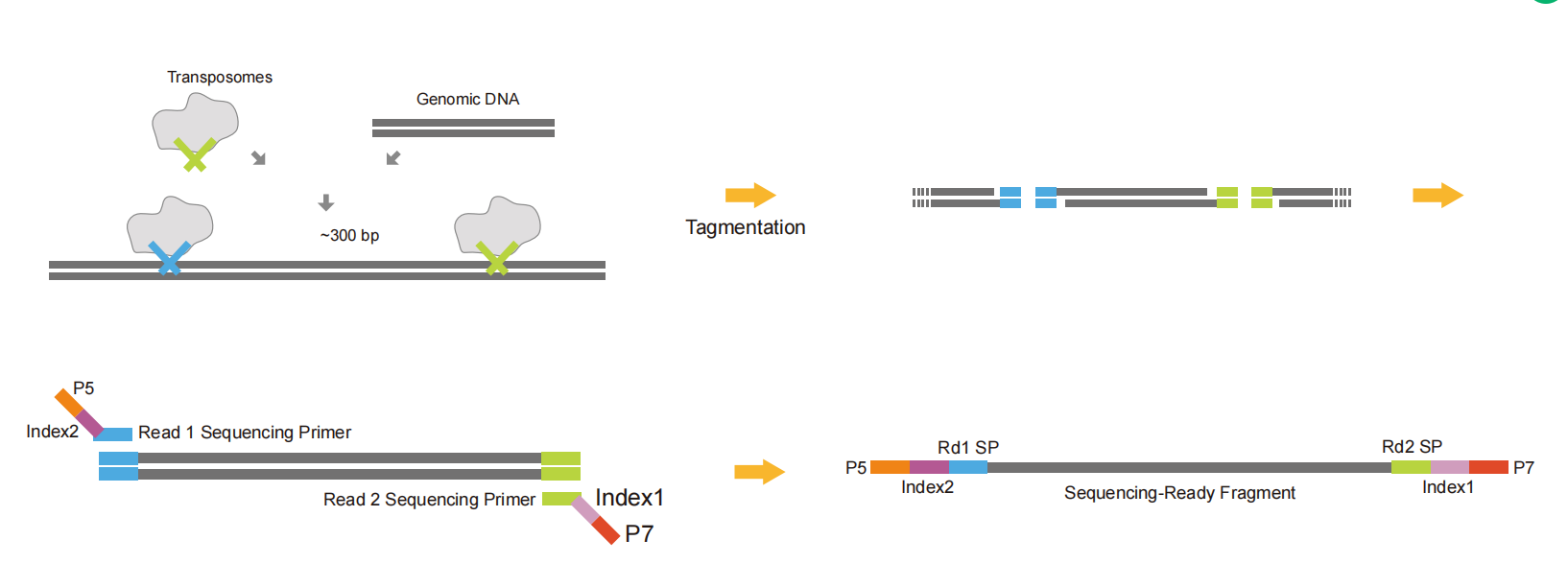
Fig.6 Schéma de la construction de la bibliothèque d'adaptateurs Tn5
5. Comment choisir les bons adaptateurs NGS pour vos plateformes de séquençage ?
Il existe actuellement deux principales plateformes de séquençage, à savoir Illumina et MGI. Yeasen, fournisseur de solutions NGS complètes, a développé plusieurs adaptateurs NGS adaptés aux plateformes Illumina ou MGI.
En ce qui concerne la plateforme Illumina, les adaptateurs Illumina NGS fournis par Yeasen contiennent trois types, notamment UDI, CDI et un seul index. En ce qui concerne la plateforme MGI, les adaptateurs MGI NGS proposés par Yeasen ont deux types, impliquant Dual UMI-UDB et index unique.Nous avons répertorié les informations sur le produit dans le tableau suivant, y compris les types d'adaptateurs, les tailles disponibles et la concentration de l'adaptateur et de l'apprêt, respectivement.
Les adaptateurs NGS complets et UDI n'ont pas à se soucier des problèmes de couplage, adaptés aux clients qui souhaitent une utilisation facile ; les adaptateurs NGS CDI ont moins de tubes et une petite taille, ce qui convient aux clients qui souhaitent stocker et transporter facilement. L'absence de PCR nécessite l'utilisation d'adaptateurs NGS complets.
Pour Illumina
| DansTube | Hieff NGS® DPréparation à la Libération NA Amorce 384 CDI pour Illumina, jeu 1 (8*12, index 96) | 12412ES |
| Hieff NGS® DPréparation à la Libération NA Amorce 384 CDI pour Illumina, lot 2 (8*12, index 96) | 12413ES | |
| Amorce Hieff NGS® RNA Lib Prep 384 CDI pour Illumina, ensemble 1 (index 96) | 12414ES | |
| Amorce Hieff NGS® RNA Lib Prep 384 CDI pour Illumina, ensemble 1 (index 96) | 12415ES | |
| Dans la plaque | Kit d'amorces UDI Stubby Hieff NGS® pour Illumina (index 1-384) Ensemble 1-4 | 12407ES |
| Kit d'amorces UDI Hieff NGS® Stubby pour Illumina Ensemble 1(Plaque à 96 puits, index 1-96) Ensemble 1 | 12327ES | |
| Kit d'amorces UDI Hieff NGS® Stubby pour Illumina Ensemble 2(Plaque à 96 puits, 97-192 Index) Ensemble 2 | 12328ES | |
| Kit d'amorces UDI Hieff NGS® Stubby pour Illumina Ensemble 3(Plaque à 96 puits, 193-288 Index) Ensemble 3 | 12329ES | |
| Kit d'amorces UDI Hieff NGS® Stubby pour Illumina Ensemble 4(Plaque à 96 puits, 289-384 Index) Ensemble 4 | 12330ES |
Pour MGI
| Chef Système de gestion de la sécurité nationale (NGS™) Kit adaptateur UDB double UMI pour MGI, Set1/Set2 | 96 types d'index | |
| Système de surveillance de la glycémie haute performance (NGS) de Hieff Kit adaptateur complet pour MGI, Set1/Set2/Set3 (Demande) | 13360ES | 8 types d'index, 41-48 |
| 13361ES | 16 types d'index, 57-72 | |
| 13362ES | 96 types d'index, 1-96 |
Concernant la lecture
Enzymes clés impliquées dans la construction de la bibliothèque NGS
Que savez-vous de la technologie liée au NGS ?
Différents types de billes magnétiques en NGS : billes magnétiques ADN\ARN\ARNm
Quantification de la bibliothèque NGS : Qubit rapide et précis ou qPCR précis ? Tout est nécessaire !
