DNasi I e le loro applicazioni in biomedicina
La Deossiribonucleasi I (DNasi I) è un'endonucleasi, la sua applicazione non è solo per mantenere l'integrità dell'RNA ma anche per l'analisi dell'impronta del DNA, la generazione di librerie di DNA casuali, la riduzione dell'appiccicosità nei lisati cellulari o negli estratti proteici, ecc. In una parola, la DNasi I può essere utilizzata in quasi tutte le applicazioni che richiedono scissioni enzimatiche del DNA. Quella che segue è un'introduzione dettagliata alla DNasi I e alla sua applicazione specifica.
1. Che cos'è la DNasi I?
2. DNasi I per la preparazione dell'estrazione di RNA senza DNA
3. DNasi I per la trascrizione in vitro per rimuovere il DNA stampo
4. DNasi I per la rimozione dell'rRNA
5. DNasi I per l'etichettatura del DNA
6. Altre applicazioni
7. Guida alla selezione del prodotto DNasi I
1. Che cos'è la DNasi I?
Desossiribonucleasi I (DNasi I) è un'endonucleasi non specifica che può digerire DNA a singolo o doppio filamento, presente in diversi tessuti e fluidi corporei. Può idrolizzare legami fosfodiesterici per produrre mono- e oligodesossinucleotidi contenenti un gruppo 5'-fosfato e un gruppo 3'-OH. L'intervallo di pH di lavoro ottimale della DNasi I è 7-8, la sua attività dipende da Ca2+ e può essere attivata da ioni metallici bivalenti come Mn2+, Mg2+, Zn2+, ecc. In presenza di Mg2+, la DNasi I taglia casualmente qualsiasi sito di DNA a doppio filamento; in presenza di Mn2+, la DNasi I può tagliare il DNA a doppio filamento nello stesso sito per formare un'estremità smussata o una sporgenza di estremità appiccicose di 1-2 nucleotidi.
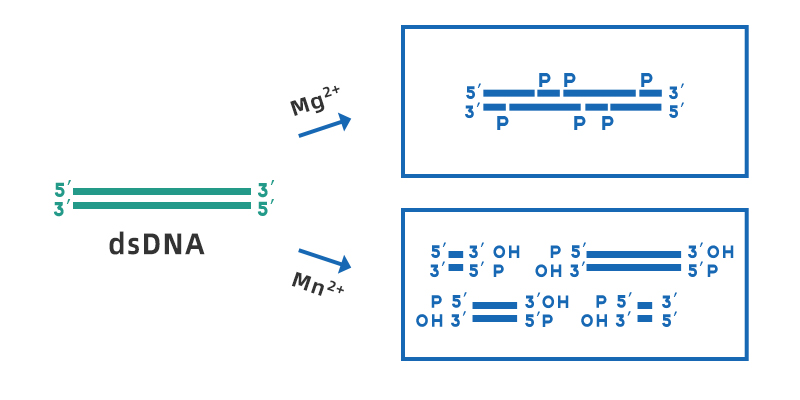
Figura 1. Diagramma schematico della scissione del dsDNA da parte della DNasi I in presenza di Mg2+ e Mn2+.
Sebbene le scissioni della DNasi I siano generalmente considerate scissioni non specifiche, è più probabile che la DNasi I agisca su determinati frammenti di sequenza, come la regione del solco minore, ed è più incline alle scissioni di sequenze purina-pirimidina. Tuttavia, quando la DNasi I agisce su dsDNA eterogeneo, tutte e quattro le basi saranno scisse e l'effetto su una base specifica non sarà più di 3 volte maggiore di quello di altre basi.
2. DNasi I per la preparazione dell'estrazione di RNA senza DNA
Negli esperimenti biologici, il primo passo è preparare l'acido nucleico per studiare le varie funzioni dell'RNA. Tuttavia, poiché DNA e RNA vengono spesso rilasciati insieme durante il processo di lisi cellulare, l'interferenza tra i due non può essere evitata indipendentemente dalla soluzione di estrazione utilizzata, quindi è necessario utilizzare enzimi specifici per rimuovere l'interferenza. Per un'estrazione di RNA di alta qualità, la DNasi I viene utilizzata per rimuovere il DNA residuo dal campione.
La DNasi I può degradare il DNA a doppio e singolo filamento in oligonucleotidi e singoli nucleotidi, e il DNA nel prodotto di preparazione dell'RNA può essere efficacemente degradato. La DNasi I viene quindi inattivata riscaldando con un tampone di arresto. Durante il processo di riscaldamento, la struttura a forcina della molecola di RNA può essere aperta, il che facilita l'ingresso diretto dell'RNA nel processo di trascrizione inversa.
La qualità dell'RNA influenzerà direttamente i dati sperimentali in larga misura. In generale, i residui di gDNA non possono essere completamente evitati durante l'estrazione dell'RNA, pertanto, si raccomanda generalmente di trattare i campioni di RNA con DNasi I per digerire il gDNA residuo prima di affrontare applicazioni downstream impegnative (ad esempio analisi dell'espressione di mRNA, analisi del trascrittoma, ecc.). La fase di digestione del gDNA può essere eseguita durante l'estrazione dell'RNA, dopo l'estrazione dell'RNA o prima della trascrizione inversa dell'RNA.In base al posizionamento del prodotto, i prodotti forniti da Yeasen sono i seguenti:
Tabella 1: Elenco dei prodotti correlati alla rimozione del DNA dall'estrazione dell'RNA o prima della trascrizione inversa
| Posizionamento del prodotto | Nome del prodotto | Gatto # |
| Estrazione dell'RNA | Reagente di estrazione dell'RNA totale TRIeasy™ [informarsi] | 10606ES |
| 19221ES | ||
| Kit RNA MolPure™ Plant Plus [informarsi] | 19292ES | |
| Kit DNA/RNA virale MolPure™ [informarsi] | 19321ES | |
| rimozione del gDNA | 10325ES | |
| Trascrizione inversa | Hifair™Ⅲ1st Strand cDNA Synthesis SuperMix per qPCR (gDNA digester plus) | 11141ES |
| PCR-q | 11184ES |
3. DNasi I per la trascrizione in vitro per rimuovere il DNA stampo
La trascrizione in vitro (IVT) utilizza principalmente il DNA come stampo, più substrati e tamponi corrispondenti per ottenere RNA tramite trascrizione in vitro. Negli esperimenti di trascrizione in vitro, le RNA polimerasi come T7, T3 e SP6 sono comunemente utilizzate per la sintesi di RNA. L'RNA sintetizzato può avere residui di DNA. L'eliminazione dei residui di DNA è vantaggiosa per lo sviluppo di esperimenti a valle. Ad esempio, nella fase di sviluppo del vaccino mRNA, la rimozione dei residui è un passaggio fondamentale, che può ridurre la difficoltà della purificazione a valle e aumentare la purezza del prodotto. Il modello di DNA viene in genere rimosso utilizzando DNasi I ricombinante (RNasi-free).Secondo il processo di sintesi dell'mRNA, i prodotti forniti da Yeasen sono i seguenti:
| Processo di sintesi dell'mRNA | Nome del prodotto | Gatto# |
| Preparazione del modello | Hieff Canace™ Plus DNA polimerasi ad alta fedeltà [informarsi] | 10148ES |
| Hieff Clone™Kit di clonazione universale in un unico passaggio | 10922ES | |
| 10125ES | ||
| FuniCut™BsaI [informarsi] | 15005ES | |
| Dispositivo FuniCut™ XbaI [informarsi] | 15033ES | |
| BspQI[richiedi informazioni] [informarsi] | 16215ES | |
| Trascrizione in vitro | 10623ES | |
| 10624ES | ||
| T7 RNA polimerasi (50 U/μL)[informarsi] | 10618ES | |
| 10133ES | ||
| 10620ES | ||
| 10621ES | ||
| Rimuovere il modello di DNA | 10611ES | |
| Modifica dell'mRNA | 10614ES | |
| 10612ES | ||
| 10132ES | ||
| 10619ES | ||
| Purificazione dell'mRNA | 12602ES |
4. DNasi I per la rimozione dell'rRNA
In vivo, l'rRNA è molto abbondante e molto conservativo, il che ha poca importanza nell'ottenimento di informazioni biologiche; per questo motivo, spesso l'rRNA viene rimosso per primo durante la costruzione e il sequenziamento delle librerie di RNA.Attualmente, il metodo di rimozione dell'rRNA è principalmente la digestione con RNasi H. I passaggi principali della deplezione enzimatica dell'rRNA sono mostrati nella figura 2: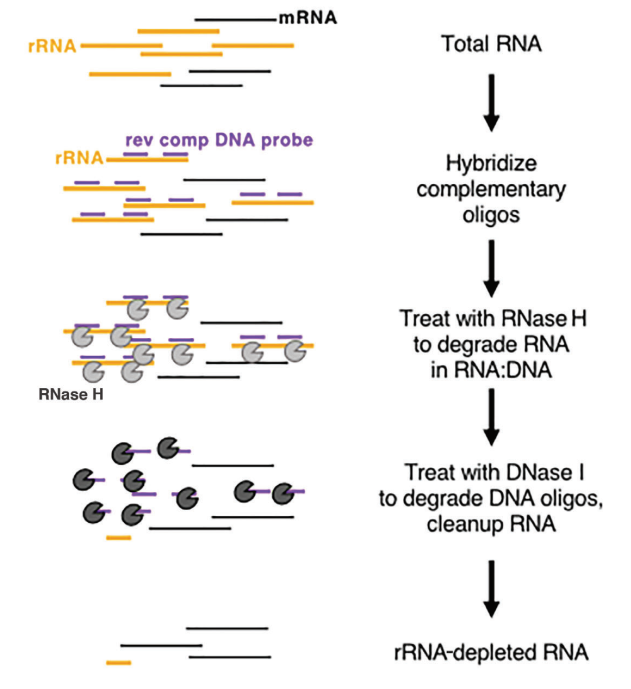
Figura 2: Diagramma schematico del principio di deplezione dell'rRNA basata sugli enzimi (Baldwin, A. et al. 2021, Current Protocols)
Per prima cosa, estrai l'RNA totale, quindi ibridizza la sonda di DNA a singolo filamento con rRNA, progetta e sintetizza la sonda di DNA a singolo filamento specifica per rRNA, quindi usa RNasi H per degradare l'rRNA ibridato e usa DNasi I per degradare la sonda di DNA. Infine, lascia il modello di RNA non rRNA. I prodotti correlati alla rimozione di rRNA forniti da Yeasen sono i seguenti:
| Processo di sintesi dell'mRNA | Nome del prodotto | Gatto# |
| Umano/Topo/Ratto Deplezione dell'rRNA | Sistema NGS™ Hieff Kit di deplezione dell'rRNA MaxUp (umano/topo/ratto) MaxUp [informarsi] | 12253ES |
| Deplezione dell'rRNA nelle piante | Sistema NGS™ Hieff Kit di deplezione dell'rRNA MaxUp (pianta) | 12254ES |
| Rimozione dell'RNA ribosomiale e delle regioni 45S ITS/ETS dall'RNA totale umano | Sistema NGS™ Hieff Kit di deplezione dell'rRNA umano MaxUp (rRNA e ITS/ETS) | 12257ES |
| Degradazione dell'rRNA | 12906ES | |
| Degradazione della sonda del DNA | 10325ES |
5.DNasi I per l'etichettatura del DNA
La traduzione nick è uno dei metodi di etichettatura delle sonde di acido desossiribonucleico più comunemente utilizzati in laboratorio. Questo metodo utilizza varie attività enzimatiche della DNA polimerasi I per incorporare trifosfati di desossiribonucleosidi marcati in catene di DNA appena sintetizzate. In questo modo, vengono sintetizzate sonde di DNA marcate in modo uniforme per un'elevata attività specifica. Le caratteristiche della traduzione nick sono sonde veloci, semplici, deliberate, ad alta specificità e marcate in modo uniforme, adatte per DNA a doppio filamento più lungo.Il metodo è realizzato dall'azione combinata di DNasi I e DNA Polimerasi I di E. coli. I passaggi principali della marcatura del DNA mediante traduzione nick sono mostrati nella figura 3:
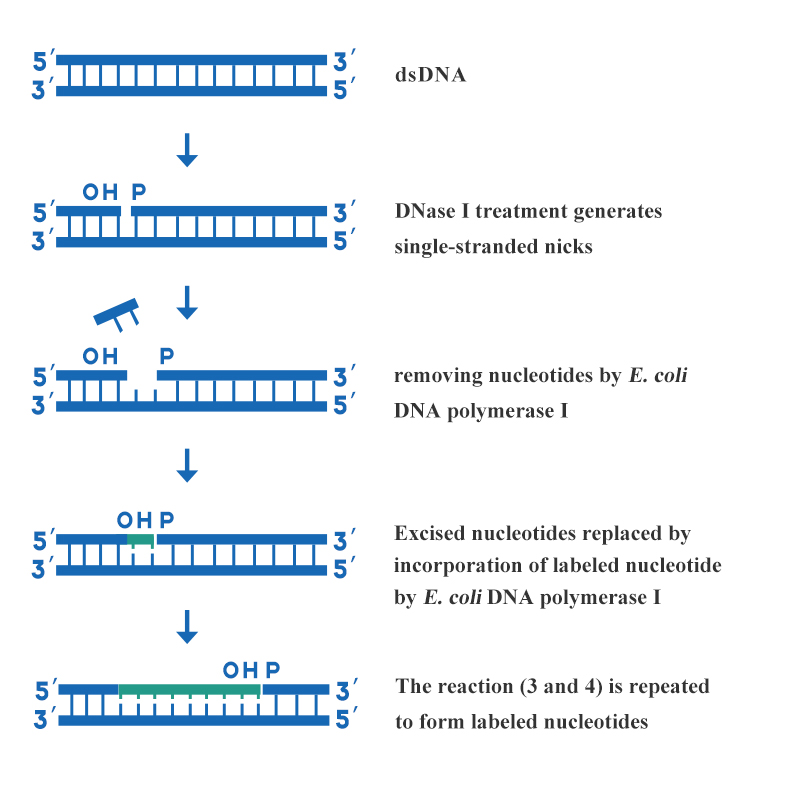
Figura 3: Diagramma schematico dell'etichettatura del DNA mediante traduzione nick
Una concentrazione adatta di DNasi I viene utilizzata per creare diversi gap a singolo filamento su ogni filamento del DNA a doppio filamento da etichettare, e il terminale idrossilico 3' viene formato nel gap. Utilizzare l'attività esonucleasica 5'→3' della DNA Polimerasi I di E. coli per tagliare un nucleotide dall'estremità 5' del taglio, e allo stesso tempo l'attività polimerasica 5'→3' della DNA Polimerasi I di E. coli introduce un nucleotide etichettato con l'estremità 3' del gap per riparare il gap. Mentre il gap si sposta lungo il filamento di DNA, i nucleotidi etichettati vengono incorporati nel filamento appena sintetizzato.I prodotti correlati all'etichettatura del DNA forniti da Yeasen sono i seguenti:
| Posizionamento del prodotto | Nome del prodotto | Gatto# |
| Ordinario | Desossiribonucleasi I (DNasi I) dal pancreas bovino [informarsi] | 10607ES/10608ES |
| Senza RNasi | 10325ES | |
| Escherichia coli fonte | 12903ES |
6. Altre applicazioni
Le applicazioni sopra menzionate sono diverse e di uso comune. Altre applicazioni della DNasi I includono le seguenti, come il test di footprinting della DNasi I e i siti ipersensibili alla DNasi I. Il test di footprinting della DNasi I è un metodo di rilevamento che può identificare con precisione i siti di legame delle proteine che legano il DNA sul DNA. Quando una proteina si lega a un frammento di DNA, può proteggere il sito di legame dal danneggiamento da parte della DNasi I e i frammenti di DNA saranno lasciati indietro dopo la digestione enzimatica ("impronta") e la sua sequenza può essere determinata. Nell'immagine del gel, non ci sono bande in cui il DNA si lega alla proteina. Per saperne di più clicca sul link.I siti ipersensibili alla DNasi I si riferiscono a scissioni in un piccolo numero di siti specifici quando la cromatina viene trattata con bassi livelli di DNasi I e questi siti specifici sono chiamati siti ipersensibili alla DNasi I. Il principio è che quando un gene è in uno stato trascrizionalmente attivo, la cromatina contenente il gene è significativamente più sensibile alla degradazione della DNasi rispetto alla regione inattiva. Per saperne di più clicca sul link.
7. Guida alla selezione del prodotto DNasi I
Yeasen Biotechnology (Shanghai) Co., Ltd., fondata nel 2014, è un'impresa high-tech impegnata nella R&S e nella produzione di materie prime enzimatiche per utensili e anticorpi antigeni. I suoi prodotti includono enzimi diagnostici molecolari, proteine e anticorpi utilizzati in prodotti farmaceutici, test di sicurezza alimentare, allevamento, giustizia e altri settori. Ci impegniamo a fornire ai clienti nel campo delle scienze della vita prodotti e servizi di alta qualità. Le linee guida per l'acquisto del prodotto per DNase I sono le seguenti:| Nome del prodotto (Cat#) | Posizionamento del prodotto | Applicazioni consigliate |
| DNasi I dal pancreas bovino (CAT#10607,10608)[informarsi] | RNasi rimossa, non rilevata | Utilizzato principalmente nella ricerca sulle proteine: rimozione del DNA da preparati proteici. |
| DNasi I ricombinante (RNasi-free)(CAT#10325) | RNasi-Free, per la ricerca | Ideale per una vasta gamma di applicazioni: rimozione del DNA da preparazioni di RNA e proteine come librerie di cDNA sensibili alla RNasi o preparazione di campioni per esperimenti di RT-PCR. |
| UCF.ME™Desossiribonucleasi I (DNasi I) di grado GMP(CAT#10611) | Senza RNasi, di qualità farmaceutica GMP. | Ideale per una vasta gamma di applicazioni: rimozione del DNA da preparazioni di RNA e proteine come librerie di cDNA sensibili alla RNasi o preparazione di campioni per esperimenti di RT-PCR. |
Per quanto riguarda la lettura:
Reagenti di grado GMP per la sintesi in vitro di mRNA
I principi del footprinting della DNasi I e le sue applicazioni biomediche
Riferimenti
1. Baldwin A, Morris AR, Mukherjee N. Un metodo semplice, conveniente e scalabile per esaurire l'RNA ribosomiale umano per RNA-seq[J]. Current Protocols, 2021.
2. Song C, Zhang S, Huang H. Scelta di un metodo adatto per l'identificazione delle origini di replicazione nei genomi microbici[J]. Frontiers in Microbiology, 2015, 6:1049.
