Apprendimento approfondito delle materie prime enzimatiche principali utilizzate nel Rilevamento PCR di SARS-CoV-2
Da marzo 2022, l'astuto ceppo mutante Omicron ha nuovamente spezzato la vita pacifica delle persone, le nuove epidemie di coronavirus sono scoppiate in tutto il paese e hanno colpito 30 province (regioni autonome e municipalità). Come mezzo efficace di prevenzione e controllo precisi dell'epidemia di SARS-CoV-2, il rilevamento dell'acido nucleico è diventato un normale modo di vivere. "Hai fatto il test dell'acido nucleico oggi?" È diventato anche un saluto quotidiano delle persone. A proposito, sai quali materie prime fondamentali sono necessarie nel rilevamento dell'acido nucleico? Questo articolo presenterà la materia prima fondamentale fondamentale nell'enzima di rilevamento dell'acido nucleico.
1. Processo di rilevamento dell'acido nucleico per SARS-CoV-2
2. Enzimi principali nell'estrazione degli acidi nucleici
3. Enzimi principali durante RT-qPCR
4. Enzimi principali del rilevamento dell'acido nucleico SARS-CoV-2 da Yeasen
1. Processo di rilevamento dell'acido nucleico per SARS-CoV-2
Un enzima è una classe estremamente importante di biocatalizzatori con elevata efficienza catalitica e specificità di reazione. La maggior parte delle reazioni biochimiche richiede la partecipazione di enzimi. Nel processo di rilevamento dell'acido nucleico del 2019-nCoV (mostrato nella Figura 1), diversi tipi di enzimi molecolari svolgono un ruolo importante in diverse fasi sperimentali come l'estrazione dell'acido nucleico e la RT-qPCR. Successivamente, in base ai diversi collegamenti sperimentali nel rilevamento dell'acido nucleico, selezioneremo le materie prime enzimatiche principali utilizzate nel processo di rilevamento dell'acido nucleico.
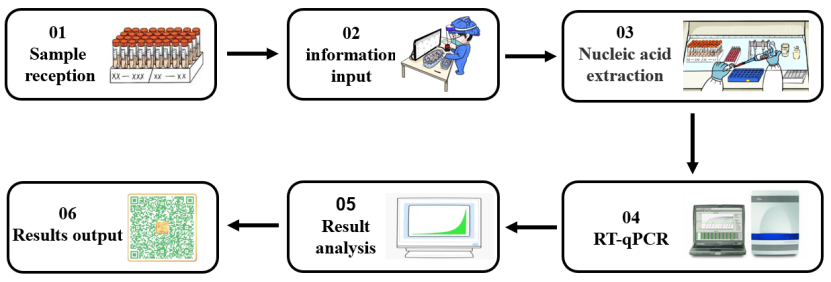
Figura 1. Processo di rilevamento dell'acido nucleico per SARS-CoV-2
2. Enzimi principali nell'estrazione degli acidi nucleici
Il nuovo processo di estrazione dell'acido nucleico del coronavirus comprende principalmente due fasi: lisi e purificazione. La lisi è il processo di distruzione della struttura cellulare del campione in modo che l'acido nucleico nel campione sia libero nel sistema di lisi; la purificazione è la separazione completa dell'acido nucleico dagli altri componenti nel sistema di lisi, come proteine, sale e altre impurità, e il processo di reazione richiede la partecipazione di proteinasi K, deossiribonucleasi I e inibitori della RNasi.
2.1 Proteasi K
La proteinasi K è una serina proteasi con ampia attività di scissione, i siti di scissione sono i legami peptidici carbossi-terminali degli amminoacidi alifatici e aromatici (Figura 2). Nel processo di estrazione dell'acido nucleico, la proteinasi K può degradare gli istoni che si legano strettamente agli acidi nucleici, promuovere la separazione degli acidi nucleici e rendere più facile l'estrazione dell'acido nucleico campione. Inoltre, la proteinasi K può degradare l'attività dell'RNA idrolasi (RNasi) e inibire l'idrolisi dell'RNA stampo da parte dell'RNasi.
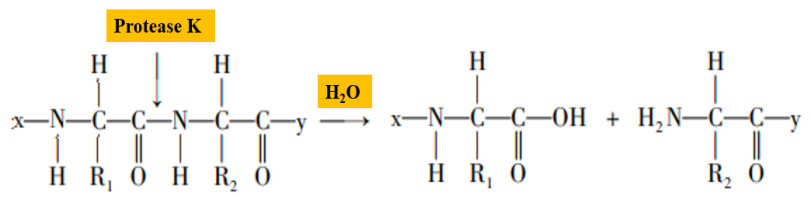
Figura 2. Diagramma schematico della proteinasi K che idrolizza i legami peptidici
Biotecnologia Yeasen Proteinasi K (Cat#10401ES) è derivato da un ceppo di lievito ricombinante, attività specifica ≥30 U/mg, privo di RNasi e DNasi e attività enzimatica stabile in soluzione di urea e SDS. È attivo in un ampio intervallo di pH (pH 4,0~12,0), adatto per la scissione e la rimozione di proteine come la preparazione di campioni di ibridazione in situ e la purificazione di acidi nucleici.
2.2 Desossiribonucleasi I
La desossiribonucleasi I (DNasi I) può catalizzare varie forme di DNA, agire sulla scissione dei legami fosfodiesterici adiacenti alle pirimidine e generare polinucleotidi con un gruppo fosfato all'estremità 5' e un gruppo idrossilico all'estremità 3'; il prodotto medio della digestione è il più piccolo politetranucleotide.Nel processo di estrazione dell'acido nucleico SARS-CoV-2, la DNasi I viene utilizzata principalmente per rimuovere la contaminazione genomica nei campioni di RNA, evitare residui di DNA nei modelli di RNA e migliorare la purezza dei modelli.
Biotecnologia Yeasen DNasi I (Cat#10325ES) è derivato da ceppi di E. coli ricombinanti, privo di RNasi, e può essere utilizzato per il trattamento di vari campioni di RNA. L'intervallo di pH di lavoro ottimale è 7,0-8,0. In presenza di Mg2+, la DNasi I può scindere casualmente qualsiasi sito di DNA a doppio filamento; mentre in presenza di Mn2+, La DNasi I può scindere il DNA a doppio filamento nello stesso sito, formando estremità smussate o estremità appiccicose sporgenti di 1-2 nucleotidi (mostrate nella Figura 3).
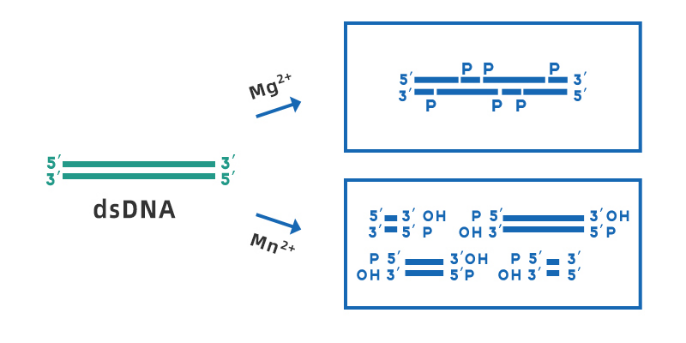
Fig 3. Diagramma schematico della scissione del dsDNA da parte della DNasi I in presenza di Mg2+ e Mn2+
2.3 Inibitore della RNasi
Nel processo di rilevamento dell'acido nucleico SARS-CoV-2, l'estrazione e la purificazione dell'acido nucleico campione o la preparazione del sistema di reazione sperimentale possono introdurre contaminazione da ribonucleasi (RNasi), con conseguente degradazione del modello di RNA. Per evitare la contaminazione da RNasi, è richiesto l'inibitore di RNasi.
RNase Inhibitor è uno specifico inibitore della RNasi nella placenta umana, che può legare specificamente la RNasi per formare un complesso con un legame non covalente e inattivare la RNasi. Yeasen Biotech Inibitore RNasi murino (Cat#10603ES) non contiene due cisteine che sono molto sensibili all'ossidazione nelle proteine umane. Ha una maggiore attività antiossidante e può inibire ampiamente vari tipi di RNasi (RNasi A, B, C), più adatto per esperimenti sensibili a ditiotreitolo elevato (DTT) come qPCR.
3. Enzimi principali durante RT-qPCR
Dopo aver completato l'estrazione dell'acido nucleico del campione SARS-CoV-2, il rilevamento dell'acido nucleico può essere completato tramite RT-qPCR. Nel corso di questi esperimenti, la DNA polimerasi, la trascrittasi inversa e l'uracil DNA glicosilasi sono tutte materie prime essenziali per l'enzima principale.
3.1 Trascrittasi inversa
Dopo l'estrazione e la purificazione, l'RNA SARS-CoV-2 necessita della trascrittasi inversa per catalizzare la polimerizzazione dNTP per generare una sequenza di cDNA complementare all'RNA stampo (Figura 4). Per la reazione RT-qPCR, dovrebbe essere selezionata una trascrittasi inversa resistente alle alte temperature. Attualmente, la trascrittasi inversa MMLV è la più ampiamente utilizzata, a causa della sua mancanza di attività endonucleasica del DNA e della bassa attività RNasi H, presenta maggiori vantaggi nell'applicazione della clonazione del cDNA.
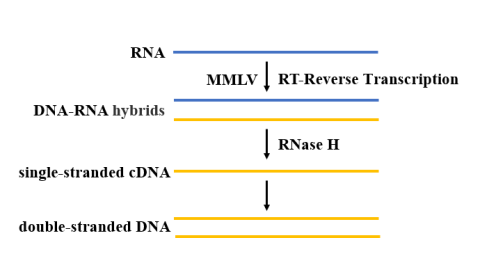
Figura 4. Diagramma schematico del processo di trascrizione inversa
Biotecnologia Yeasen Alta qualità™ V Trascrittasi inversa (Cat#11300ES) è una nuova trascrittasi inversa ottenuta tramite tecnologia di ingegneria genetica. Ha una buona stabilità termica e può sopportare temperature di reazione fino a 60°C. Ed è anche adatto per la trascrizione inversa di modelli di RNA con strutture secondarie complesse. Allo stesso tempo, l'enzima aumenta l'affinità con il modello, è adatto per la trascrizione inversa di un piccolo numero di modelli e geni a bassa copia e può amplificare il cDNA fino a 10 kb.
3.2 DNA polimerasi
Una volta completato il processo di trascrizione inversa del modello per generare cDNA a doppio filamento, è necessario che la DNA polimerasi "soul player" nella reazione PCR faccia la sua comparsa, polimerizzando i deossiribonucleotidi liberi per estendere la catena del DNA, e una grande quantità di DNA modello viene amplificata in vitro per raggiungere lo scopo di rilevamento dell'acido nucleico virale.
La DNA polimerasi comunemente usata nelle reazioni RT-qPCR è la Taq DNA polimerasi hot-start. Questo tipo di enzima è inattivo a temperatura ambiente.Ha attività di polimerizzazione solo dopo l'avvio a caldo, il che può ridurre al minimo la generazione di segnali di fondo. Risolve i problemi di amplificazione non specifica causati dalla generazione di primer-dimeri o dalla mancata corrispondenza nelle reazioni PCR convenzionali. Attualmente, i metodi di modifica hot-start della DNA polimerasi comunemente utilizzati includono principalmente la modifica chimica, la modifica del ligando e la modifica dell'anticorpo. I principi dei diversi metodi di modifica hot-start sono mostrati nella Figura 5.
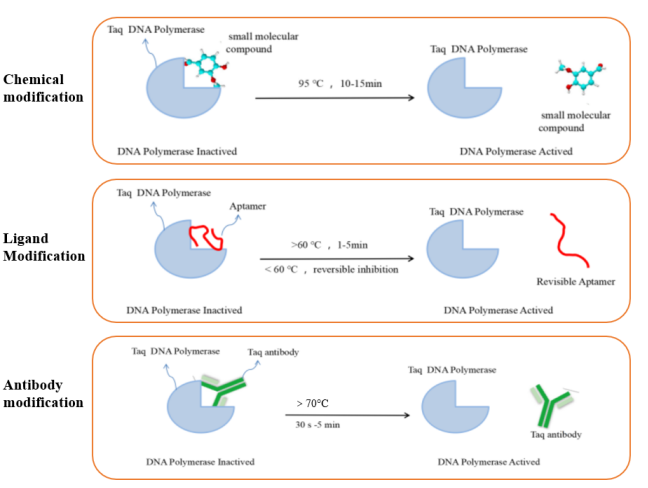
Figura 5. Diagramma schematico di diversi tipi di enzimi hot-start modificati
Biotecnologia Yeasen UNICONTM Hotstart Taq DNA polimerasi altamente specifica, 5 U/μL (Cat#10726ES) è una DNA polimerasi hot-start a doppio blocco con elevata affinità per il template. A temperatura ambiente, non solo il 5'→3' l'attività della polimerasi è bloccata, ma anche la 5'→3' l'attività esonucleasica è bloccata. La denaturazione a 95°C per 30 sec può inattivare completamente l'anticorpo bloccante, rilasciando l'attività della DNA polimerasi e l'attività esonucleasica. La caratteristica del doppio blocco può non solo prevenire efficacemente l'amplificazione non specifica causata da mismatch o primer-dimeri, ma anche inibire efficacemente la diminuzione del segnale di fluorescenza causata dalla degradazione della sonda. La doppia garanzia rende i reagenti di rilevamento in vitro più stabili durante il trasporto o gli utilizzi a temperatura ambiente.
3.3 Uracile DNA glicosilasi
Nel processo di rilevamento dell'acido nucleico del nuovo coronavirus, l'inquinamento da aerosol nell'ambiente operativo è il fattore più comune che causa risultati PCR falsi positivi. L'aggiunta dell'enzima UDG (Uracil DNA Glycosylase, uracil DNA glycosylase) al sistema di amplificazione può eliminare efficacemente gli inquinanti residui di amplificazione (principalmente sotto forma di aerosol) miscelati nel sistema PCR per garantire l'accuratezza dei risultati di amplificazione. Il principio anti-inquinamento dell'enzima UDG è mostrato nella Figura 6.
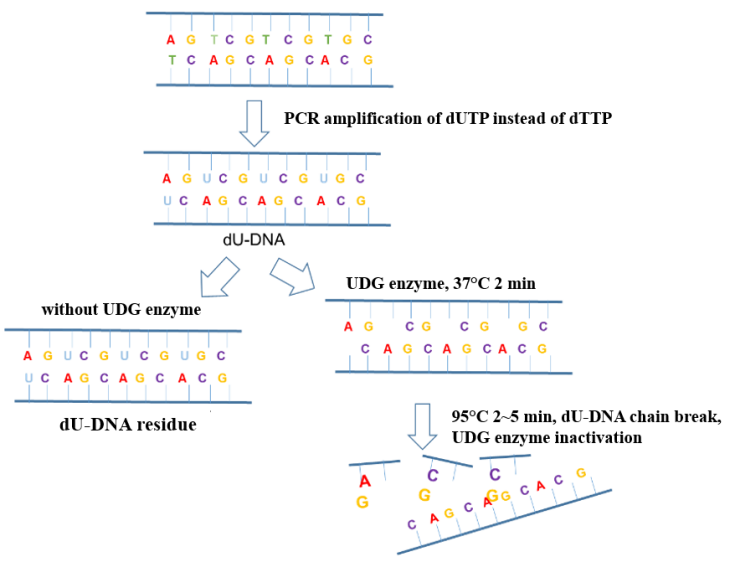
Figura 6. Diagramma schematico del principio anti-inquinamento dell'enzima UDG
Biotecnologia Yeasen L'Uracil DNA Glycosylase (UDG/UNG), labile al calore, 1 U/μL (Cat#10303ES) è attiva a 25-37°C, sensibile al calore e irreversibilmente inattivata a 50°C per 10 min o 95°C per 2 min. Non vi è alcun residuo di endonucleasi e RNasi e il residuo del genoma dei batteri ospiti è inferiore a 10 copie. Può essere utilizzata insieme alla Taq DNA polimerasi hot-start per garantire la specificità della reazione di amplificazione.
4.Enzimi principali del rilevamento dell'acido nucleico SARS-CoV-2 da Yeasen
| Processo | Descrizione | Nome del prodotto | Codice Prodotto |
| Elaborazione del campione | Digestione delle proteine | 10401ES | |
| Estrazione dell'RNA | Ricombinante DNasi I (senza RNasi) (Richiedi informazioni) | 10325ES | |
| Inibizione della RNasi | Inibitore RNasi murino (40U/μL) | 10603ES | |
| Trascrizione inversa | Adatto per RT-qPCR | Alta qualità™ V Trascrittasi inversa (200U/μL) | 11300ES |
| Trascrittasi inversa HifairTM V (600U/μL) senza GLyceroL (Richiedi informazioni) | 11301ES | ||
| Amplificazione PCR | DNA polimerasi a caldo | 10726ES | |
| UDG termico | Uracile DNA glicosilasi (UDG/UNG), termolabile, 1 U/μL | 10303ES |
Per quanto riguarda la lettura:
Selezione della trascrittasi inversa
YEASEN UDG termolabile: controlla facilmente l'inquinamento da aerosol
Inibitori della RNasi murina: eliminano con successo la contaminazione da RNasi e preservano l'RNA
